导读
本研究描述了一个连接分析工具箱(CATO),用于基于扩散加权成像(DWI)和静息态功能磁共振成像(rs-fMRI)数据来重建大脑结构和功能连接。CATO是一个多模态软件包,使研究人员能够运行从MRI数据到结构和功能连接组图的端到端重建,定制其分析并利用各种软件包对数据进行预处理。结构和功能连接组图可以根据用户定义的(亚)皮层图谱进行重建,为集成多模态分析提供对齐的连接矩阵。CATO是在麻省理工学院(MIT)许可下发布的开源软件,可作为MATLAB工具箱和独立应用程序使用,该软件可在网站www.dutchconnectomelab.nl/CATO上获取。
前言
对宏观大脑结构和功能连接数据的网络分析已成为神经科学中广泛使用的方法。包括磁共振成像(MRI)在内的神经成像数据可用于重建大脑的解剖连接和功能同步模式,并研究衍生网络的拓扑结构。通过对人类和动物大脑的网络研究发现了健康大脑结构和功能的一般组织原则,以及与精神和神经疾病相关的异常。
MRI连接组研究旨在通过扩散加权成像(DWI)和静息态功能MRI(rs-fMRI)数据重建和研究结构和功能连接图,该领域正在迅速开发用于重建大脑连接的工具。这些重建工具和管道在目标(例如,成像模式和处理步骤)、编程语言(例如,C、MATLAB和python)和方法(例如,概率性与确定性纤维追踪、数据驱动与预定义的结构划分)方面有所不同。多种软件包的可用性有利于研究人员选择适合其研究、专业知识水平和首选编程框架的软件包。
本文描述了CATO——连接分析工具箱,一个从DWI和rs-fMRI数据来重建结构和功能连接组的灵活工具箱。CATO提供了一个简单的软件包,可使用常用和已发布的算法将原始DWI/rs-fMRI数据映射到连接体,并具有可配置的选项,用户可以根据所需的细节水平进行调整。对于刚开始研究连接组学的研究人员,本研究在(www.dutchconnectomelab.nl/CATO)上提供了文档,以及指导用户为其特定数据集创建配置文件的在线配置助手。该工具箱提供了可配置的模块化结构,具有可定制的预处理和执行特定结构和功能处理步骤的选项。这种灵活性允许用户利用CATO从FreeSurfer、FSL或其他数据预处理工具进行预处理的成像数据中映射连接体。此外,用户还可以选择将自己的处理步骤作为附加组件添加到管道中,以执行特定的后处理和其他任务。
CATO允许在相同的图谱模板集上重建结构和功能连接。匹配连接重建使研究人员能够研究两种模式之间的相互作用,这可以为功能活动的动力学或脑疾病的生物机制提供新的见解。CATO中的结构管道使用扩散张量成像(DTI)、广义q采样成像(GQI)、约束球面反卷积(CSD)和确定性纤维追踪算法来实现扩散重建。CATO还具有将高级扩散重建方法(GQI,CSD)与简单且稳健的扩散重建方法(DTI)相结合的功能。功能连接可以使用Pearson相关系数或用户定义的测量(如偏相关系数或协方差测量)来估计。
CATO是在MATLAB中开发的,可以作为一个基于MATLAB的工具箱和独立的应用程序使用,也可以在Docker容器中使用。该工具箱是在MIT许可下发布的开源软件,可在GitHub存储库www.GitHub.com/dutchconnectomelab/CATO中获取。本文描述了结构和功能管道中的步骤,然后对配置助手(Configuration Assistant)进行了说明,该助手提供了一个图形用户界面,用于创建和修改不同管道的配置文件。
方法
CATO可作为MATLAB的软件包,也可作为编译的独立可执行文件在高性能计算机集群上使用。它有两个独立的管道用于重建结构和功能连接,每个管道包含多个处理步骤,这些步骤使用MATLAB函数来实现(见图1)。structural_pipeline和functional_pipeline主函数验证用户提供的输入参数和文件,并执行所需的管道步骤。预处理和分割函数通过执行用户指定的bash shell脚本来利用外部软件(见图2)。CATO提供的示例预处理脚本和图谱分割脚本是使用FreeSurfer和FSL来执行预处理和分割的。
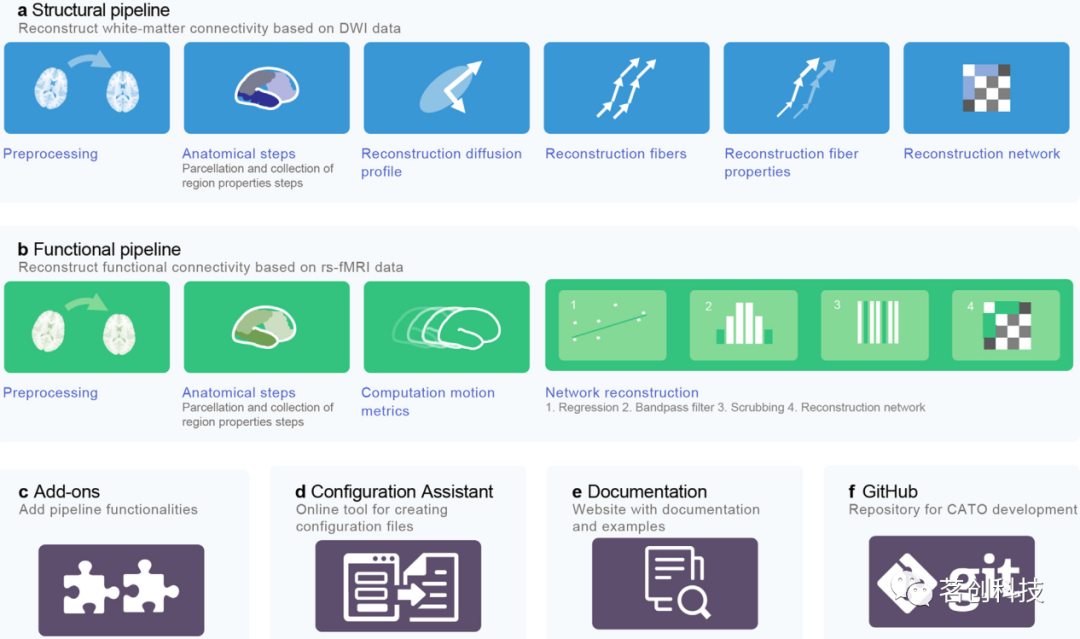
图1.CATO工具箱和管道概述。
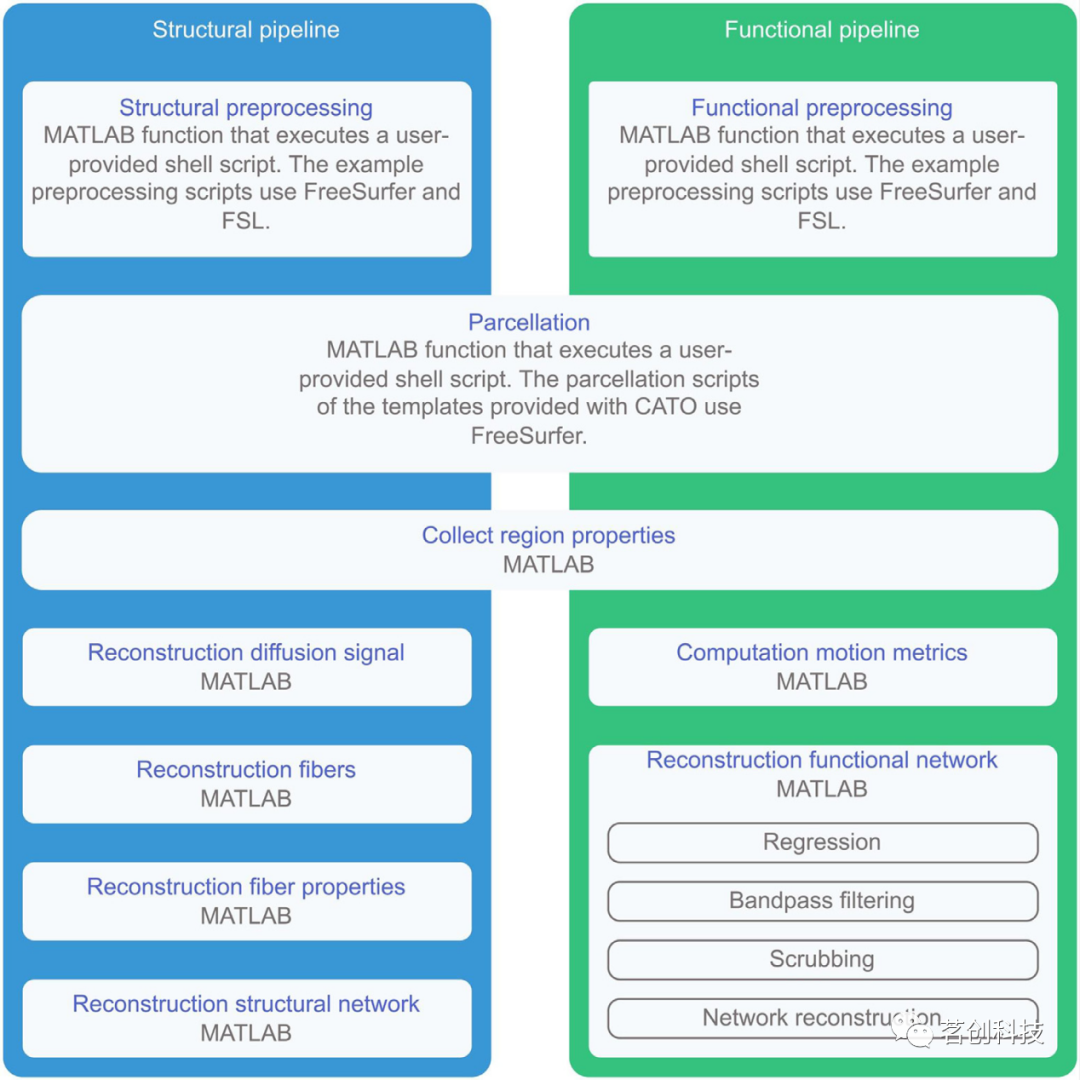
图2.结构和功能管道及其依赖项的概述。
Part I:结构管道
结构管道的重建参数可以在命令行或配置文件中以JSON格式指定。重建参数描述了所需的处理步骤(详细内容如下)、FreeSurfer解剖目录的位置、DWI文件的位置、梯度信息文件的位置、皮层图谱类型,以及预处理、扩散重建、纤维追踪和网络重建的设置。
①结构预处理
结构管道需要预处理的T1数据(用于组织分类和皮层分割)和DWI集(建议30个或更多的扩散方向)。本研究推荐使用FreeSurfer皮层自动重建过程来预处理结构T1数据。在运行CATO之前进行FreeSurfer皮层重建,可以提供下游网络重建所需的文件,并提高了并行处理被试时的计算效率。对于DWI数据,本研究建议使用FSL软件包对DWI数据进行预处理。所需的结构预处理步骤可以由参数preprocessingScript指定的bash-shell脚本执行,该脚本允许用户自定义最适合其数据的预处理脚本,或者直接从已经预处理过的数据开始。结构管道提供了三个示例预处理脚本(FSL topup-eddy-preprocessing、FSL eddy-preprocessing和minimal-preprocessing),用于执行后续纤维追踪和网络重建所需的所有步骤和数据输出文件。这些示例预处理脚本调用文档良好且经过全面评估的FSL topup和FSL eddy工具来校正DWI数据中磁化率引起的失真、涡流失真和运动伪影,更新b向量以调整DWI校正,根据校正后的扩散未加权(b0)体积计算DWI参考图像,使用FSL的bbregister计算DWI参考图像和解剖T1图像之间的配准矩阵,并将FreeSurfer分割配准到DWI参考图像上。
②解剖分割
相对于参考图谱,分割产生了表面的皮层分区。CATO包含的图谱有FreeSurfer中的Desikan-Killiany图谱,以及Desikan-Killiany图谱的120、250和500区域Cammoun亚分区和Von Economo-Koskinas皮质区和皮质型图谱。每个参考图谱在TOOLBOXDIR/templates中都有一个目录,其中包含一个分割脚本(bash script)、一个ROI文件和特定于模板的文件。文件名在配置文件中定义为变量(templatesDir、templateScript、ROIsFile),用户可以对其进行修改。
③解剖分割-汇总区域属性。
collect_region_properties步骤收集大脑区域的体积和表面数据,并将这些数据汇总在regionPropertiesFile(MATLAB-file)中。每个脑区都包含以下统计信息:i)每个区域的质心(根据分割文件计算),ii)顶点数,iii)表面积(mm2),iv)灰质体积(mm3),以及v)平均厚度(mm)(均来自FreeSurfer的统计文件)。这些度量将在管道的后续步骤中使用。
④重建扩散信号
reconstruction_diffusion步骤根据测量的DWI数据估计每个体素中的白质纤维组织。结构连接建模是基于白质纤维限制水分子运动的原理,从而导致体素中扩散信号的峰值(优选扩散方向)。reconstruction_diffusion步骤提供了三种从DWI数据推断扩散峰值的方法,包括扩散张量成像(DTI)、约束球面反卷积(CSD)和广义Q采样成像(GQI),用户也可以根据需要执行其他方法。对于每种方法,在配置文件中都提供了重建参数,所有体素的重建峰值都保存在diffusionPeaksFile (MATLAB文件)中,相关的扩散测量值保存在differsionMeasuresFile (MATLAB文件)中。
⑤扩散张量成像(DTI)
DTI重建通过单个张量对体素中测量的信号进行建模,使用RESTORE算法执行张量估计,该算法在拟合过程中执行张量估计,同时识别和去除异常值,减少了生理噪声伪影对扩散张量建模的影响。基于Gavin(2019)执行的Levenberg-Marquardt方法用于解决非线性最小二乘问题。根据估计张量计算出四种扩散系数,包括分数各向异性、轴向扩散率、径向扩散率和平均扩散率。
⑥约束球面反卷积(CSD)
CSD通过对测量信号与纤维相关的扩散分布进行去卷积来重建体素中的纤维取向分布函数(fODF)。在超分辨率球面谐波框架中执行信号去卷积,以允许对扩散过程的表面进行自然描述(球面谐波的阶数由参数shOrder设置)。球面反卷积约束于非负球面谐波上,从而减少了高频噪声,并能重建出一些明确的峰值。参数lambda指定正则化参数λ,该参数控制重建fODF的粗度。参数tau指定振幅阈值τ,低于该阈值对应的fODF被假定为零。从重建的fODF的局部最大值中选择扩散峰值。如果局部最大值的fODF值(通过fODF的最大值归一化)大于或等于minPeakRatio,则将其视为扩散峰。扩散峰值的最大局部极值的数量由outputPeaks参数设置。大于maxPeaks最大局部极值的体素被认为具有各向同性扩散分布,不选择扩散峰值。
⑦广义Q采样成像(GQI)
GQI利用扩散信号与潜在扩散位移之间的傅里叶变换关系来重建体素中的fODF。该实现遵循Yeh等人(2010)中描述的方法,以及http://dsi-studio.labsolver.org上提供的示例。参数meanDiffusionDistanceRatio用于调节fODF粗度的平均扩散距离比。从fODF重建扩散峰的方法与上述CSD重建方法相同。计算各峰的广义分数各向异性和定量各向异性,并将其添加到diffusionMeasuresFile中。
⑧联合实现
比较这三种重建方法,DTI是一种稳健且相对简单的方法,CSD和GQI是更先进的扩散模型,可以更好地描述更复杂白质体素中的扩散信号。为了结合简单和先进重建方法的优势,CATO为用户提供了将DTI与CSD或GQI相结合的选项。该选项在CSD或GQI仅估计一个峰值的体素中执行DTI建模(更稳健),并在检测到多个峰值的体素中使用CSD或GQI(在具有复杂纤维结构的体素上更灵活)。在配置文件中设置了每种重建方法的具体参数,并在在线文档中进行了更详细的描述。
⑨中间输出文件
reconstruction_diffusion步骤为用户提供了将扩散测量值导出到NIFTI文件diffusionMeasuresFileNifti的附加选项。导出的标准扩散测量值包括分数各向异性、轴向扩散率、径向扩散率和平均扩散率。
⑩重建纤维
reconstruction_fibers步骤基于每个体素的扩散峰值执行纤维追踪。纤维追踪步骤采用了标准的“连续追踪纤维分配”(FACT)算法。FACT描述了一种确定性追踪算法,该算法从白质中的种子开始纤维重建,并在体素的主扩散轴上传播流线,同时每当流线的尖端进入新的体素时更新传播方向。当使用CSD、GQI或二者结合的方法时,通过遵循最接近追踪方向的峰值方向,调整算法以兼容多个扩散峰,与其他FACT算法实现一致。FACT的CATO实现从所有体素中的一个或多个种子(由NumberOfSeedsPerVoxel设置的种子数量)开始,并具有与startRegions提供的列表匹配的分割标签。
⑪重建纤维特性
reconstruction_fiber_properties步骤识别连接大脑区域的纤维段,并计算纤维测量值,为网络重建步骤做准备。对于每个图谱和重建方法,reconstruction_fiber_properties步骤迭代所有纤维,如果纤维跨越两个或多个感兴趣的脑区(由感兴趣区域文件定义(ROIsFile,text文件)),则每个区域对之间最短的纤维段包含在纤维特性文件(fiberPropertiesFile,MATLAB文件)中。除了每个纤维段的起点和终点以及相关区域对之外,还存储了纤维段的其他测量指标,包括:最大转角、最小分数各向异性、纤维长度(物理长度mm)和平均分数各向异性,轴向扩散率、径向扩散率、平均扩散率和广义分数各向异性。扩散测量在体素上按通过每个体素的路径长度加权平均。
⑫结构连接矩阵
reconstruction_network步骤为每个选定的(亚)皮层图谱和重建方法建立连接矩阵。连接矩阵中包含的脑区及其顺序由感兴趣区域文件(ROIsFile)定义。通过迭代fiberPropertiesFile中的所有纤维段来构建网络矩阵,并将连接感兴趣区域的纤维段添加到连接矩阵中。
除了在reconstruction_fibers步骤中使用的纤维重建标准之外,还可以在reconstruction_network步骤中通过纤维的投影长度、最小分数各向异性(仅包括接触体素的分数各向异性高于minFA的纤维)和最大角度(仅包括轨迹转弯小于maxAngleDeg的纤维)来过滤纤维(只有长于minLengthMM的纤维被包含在网络重建中)。连接矩阵保存在文件connectivityMatrixFile(MATLAB文件)中。连接矩阵包括连接两个区域的流线数量(即纤维数量)、纤维长度(物理长度,单位为mm)、平均分数各向异性、轴向扩散率、径向扩散率、平均扩散率、广义分数各向异性、重建流线段的流线体积密度和流线表面密度等权重。
Part II:功能连接
功能连接管道建立在与结构连接管道相同的模块化组织上,包括一个预处理步骤、两个解剖步骤和一个连接重建步骤(见图1)。静息态fMRI处理从主函数functional_pipeline开始,该函数根据命令行或rs-fMRI管道配置文件(JSON文件)中提供的参数运行处理步骤。重建参数描述了所需的处理步骤(详见下文)、解剖FreeSurfer目录的位置、rs-fMRI文件的位置、皮层图谱类型以及预处理、回归、带通滤波、清洗和网络重建的设置。功能管道包括以下步骤:
①预处理
默认的预处理脚本执行以下步骤:使用FSL工具slicetimer校正时间层,使用FSL工具MCFLIRT校正运动伪影,通过对所有(运动校正后的)rs-fMRI帧进行平均来计算rs-fMRI参考图像(使用FSL工具),计算rs-fMRI参考图像和T1图像之间的配准矩阵(使用FreeSurfer),以及将T1分割配准到rs-fMRI图像上(使用FreeSurfer)。模块化管道允许使用其他软件包进行预处理,并在需要时完全自定义预处理。文档网站上提供了其他预处理示例脚本,例如BIDS组织的数据和使用ICA+FIX对rs-fMRI数据进行预处理。
②解剖分割
功能管道的第一步包括与结构管道相同的分割步骤parcellation和collect_region_properties,从而在与结构管道相同的图谱上分割皮层表面,这样就可以在后处理整合多模态分析中探索结构-功能关系。解剖步骤进一步计算这些区域的解剖统计信息,并将区域特性收集到regionPropertiesFile(MATLAB文件)中。
③计算运动指标
Compute_motion_metrics为每一帧计算运动度量逐帧位移(FD)和帧间信号强度的变化(称为“DVARS”)。度量源自motionParametersFile文件,该文件可以在预处理步骤中使用MCFLIRT创建,并保存在motionMetricsFile(MATLAB文件)中。逐帧位移(FD)计算为帧内估计的平移和旋转位移之和,旋转位移在motionParametersFile文件(MATLAB文件)中以度为单位定义。通过计算半径为50mm的球体表面的预期位移,将FD转换为毫米,作为大脑皮层的模型。
④功能连接矩阵
reconstruction_functional_network步骤先计算功能连接矩阵,然后对时间序列进行(可选)协变量回归、带通滤波和清洗。回归:对于每个体素,通过计算信号强度线性模型的残差,从rs-fMRI时间序列中去除协变量(称为回归变量),其中回归变量作为预测因子。带通滤波:对rs-fMRI数据进行(可选,但推荐)带通滤波,采用零相位巴特沃斯带通滤波器,其低通和高通截止频率由配置参数频率设定。清洗:从rs-fMRI时间序列中删除出现明显运动伪影的帧。可基于两个指标来识别具有运动伪影的帧:i)逐帧位移FD大于maxFD;ii)DVARS大于q3+maxDVARS×IQR,其中IQR是指四分位距IQR=q3-q1,q1和q3是指所有帧的DVARS的第一个和第三个四分位距。大于或等于minViolations的帧被标记为具有潜在运动伪影的帧,并且排除在进一步的分析之外。网络重建:reconstruction_functional_network步骤计算区域之间的功能连接。通过所选帧中这些区域的平均信号强度的相关系数来估计脑区之间的功能连接(由ROIsFile指定)。用于功能连接估计的相关性测量可以由用户指定(在reconstructionMethod参数中),选项包括Pearson相关系数、偏相关系数或协方差。虽然默认的相关性测量是Pearson相关系数,但考虑到各种测量都有其优缺点,用户可以选择自己喜欢的测量方法。连接矩阵保存在文件connectivityMatrixFile (MATLAB文件)中,每个区域的时间序列保存到文件timeSeriesFile (MATLAB文件)中。可以执行多个reconstruction_functional_network步骤来探索不同的重建设置,并使用参数methodDescription进行区分。
配置助手
配置助手是为结构和功能管道(http://www.dutchconnectomelab.nl/CATO/configuration-assistant)创建和修改配置文件的在线工具。配置助手检查所有配置参数的有效性。
自定义
除了CATO提供的标准功能外,用户还可以根据自己的特定研究需求灵活地定制工具箱。首先,用户可以通过提供自己的预处理脚本或分割脚本来定制工具箱,并可以选择调用其他软件工具。此外,用户可以通过向管道添加附加组件来扩展CATO的功能,该附加组件是一个MATLAB函数,可以将外部软件作为管道步骤执行。有了这些定制选项,研究人员可以根据自己的独特需求定制CATO。
结果
结构管道基准测试
空间重建。与ITC2015挑战赛20个不同站点提供的(共)96份基于纤维束成像的纤维云进行对比,验证了其性能。ITC2015提交的材料显示了空间重建质量测量的广泛得分:在25个基准真值束中,有效重建束的数量从5到24个束不等,无效束的数量从27到386个束不等(图3a)。在用CATO获得的重建中,25个基准真值束中有23个(92%)被重建,这与ITC2015提交的23(中位数)个束相似(图3a)。丢失的白质束包括前连合和后连合。在所有重建方法中,追踪到62至73个无效束,这些束不包括在真值白质骨架中(CSD:73、DTI:69、GQI:62、GQI-DTI:69和CSD-DTI:81),这些数据与ITC2015提交的数据相当,ITC2015提交数据的中值为77个无效束(图3a)。在ITC2015提交的数据中,与真值白质骨架在空间上重叠的纤维束的百分比(称为“有效纤维”)在3.75%至92.5%之间(中位数:62.6%),CATO值在43%至65%之间(CSD:43%,DTI:65%,GQI:53%,GQI-DTI:65%和CSD-DTI:48%,图3b)。
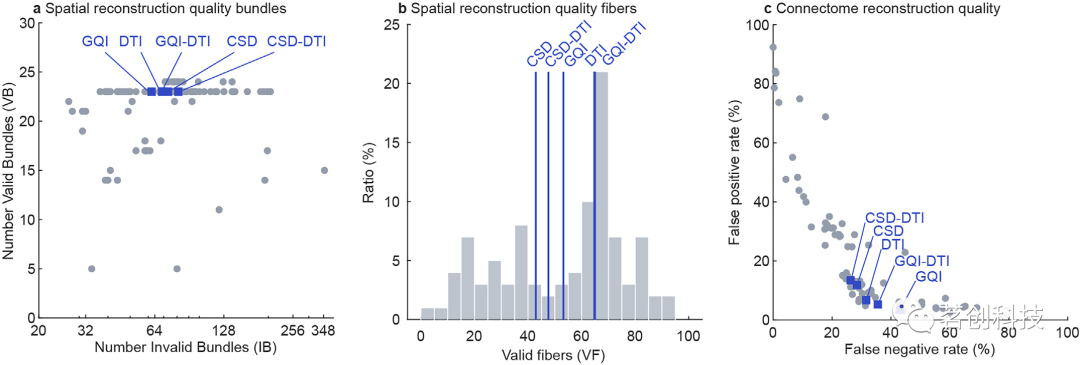
图3.基准测试重建。
连接体映射。ITC2015提交的结果(共67份)显示,假阳性率在3%-92%之间(中位数=114.8%),假阴性率在0%-69%之间(中位数=26.8%)。这强调了参加ITC2015的小组使用的不同策略之间的广泛特异性-敏感性权衡。CATO连接体重建的假阳性率在4%-12%之间(CSD:12%,DTI:7%,GQI:4%,GQI-DTI:5%和CSD-DTI:13%),假阴性率在29%-43%之间(CSD:29%,DTI:32%,GQI:43%,GQI-DTI:36%和CSD-DTI:26%,见图3c)。这种相对较低的假阳性率使CATO重建成为一种具有相对保守的重建敏感性和相对较高的连接重建特异性的连接体重建工具。连接体重建的敏感性和特异性水平可以通过改变所使用的重建参数和应用额外的后处理来进一步调整。在附加CATO通道步骤中使用COMMIT2进行额外的流线过滤,增加了连接体重建的严格性,假阳性率在1%-6%之间(CSD:5%,DTI:3%,GQI:1%,GQI-DTI:3%,CSD-DTI:6%),假阴性率在48%-62%之间(CSD:49%,DTI:54%,GQI:62%,GQI-DTI:54%,CSD-DTI:48%)。改变扩散重建和后处理参数对连接体重建的敏感性和特异性影响很小(<5%)。改变纤维重建参数对敏感性-特异性权衡有很大影响。
结构重测基准测试
在HCP重测数据集上对重建后的结构连接进行重测信度估计。通过ICC评估不同皮层分割、重建方法和连接强度的可靠性。流线数量(NOS)连接强度测量显示,在组掩蔽的连接体中,19%的连接信度“较差”,24%的连接“一般”,57%的连接“良好”至“极好”(中位数ICC=0.66,IQR=0.47-0.79,Desikan-Killiany图谱,GQI-DTI方法)。不同图谱的ICC中位数在0.59至0.70之间变化,信度“较差”的连接比例在13-22%之间,“一般”信度在21-29%之间,“良好”至“极好”信度在49-66%之间(图4a)。对于Desikan-Killiany皮层分割,使用DTI、CSD、GQI、GQI-DTI和CSD-DTI方法重建的连接体显示,ICC重测信度的中位数在0.52-0.66之间,“较差”信度的百分比在15-32%之间,“一般”信度的百分比在24-32%之间,以及“良好”至“极好”信度的百分比在37-61%之间(图4b)。分数各向异性加权连接体的重测信度也显示出类似的结果。使用GQI-DTI和Desikan-Killiany皮层分割重建的连接体显示,中位数ICC=0.44,42%的连接信度“较差”,37%的连接信度为“一般”,21%的连接信度为“良好”或“极好”(图4)。
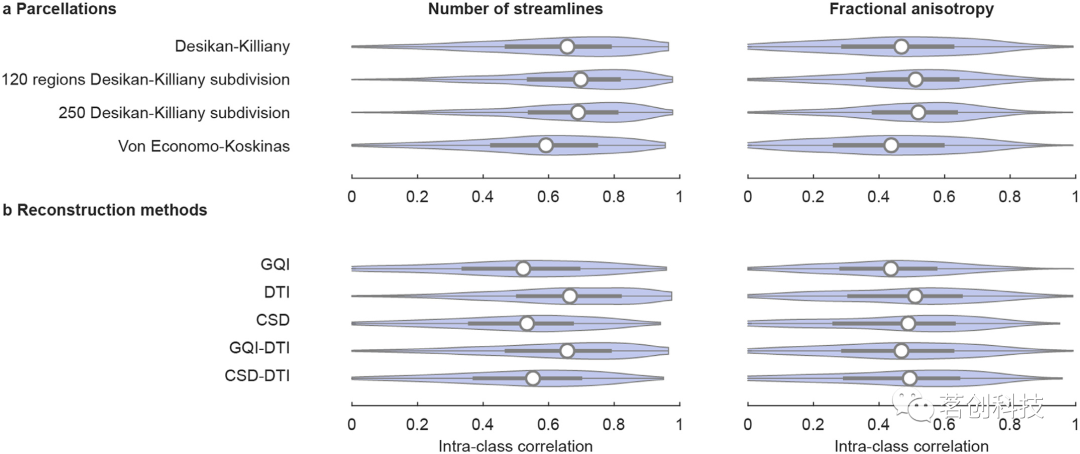
图4.结构连接组重测信度。
功能重测基准测试
功能连接体的重测信度显示,在各图谱中,有13-23%的连接信度“较差”,51-58%的信度“一般”和20-36%的信度“良好”至“极好”(图5a)。针对Desikan-Killiany分割的68个皮层区域,可以观察到包括全局平均回归的网络重建导致23%的连接信度“较差”、49%的信度“一般”和27%的信度“良好”至“极好”(中位数ICC=0.52,IQR=0.41-0.61)。省略清洗预处理步骤或带通滤波对重建可靠性没有太大影响(图5b)。

图5.功能管道重测信度。
结论
本文讨论了CATO,一个用于重建结构和功能连接组图的软件工具箱;描述了结构和功能管道中的模块化处理步骤,并详细说明了重建方法以及基准值,以帮助研究人员深入了解连接组重建的敏感性-特异性权衡和可靠性。多种连接重建工具包的可用性使神经科学社区能够使用多种分析管道来复制报告的结果,以证明研究结果对应用处理软件变化的稳健性。CATO的早期版本已用于研究健康大脑中的连接组组织、各种疾病、新生儿脑组织,以及进行连接组学的比较。CATO是一个开源的连接重建工具箱,在MIT许可证下共享。
原文:Siemon C. de Lange , Koen Helwegen , Martijn P. van den Heuvel , Structural and functional connectivity reconstruction with CATO - A Connectivity Analysis TOolbox, NeuroImage (2023), doi: https://doi.org/10.1016/j.neuroimage.2023.120108