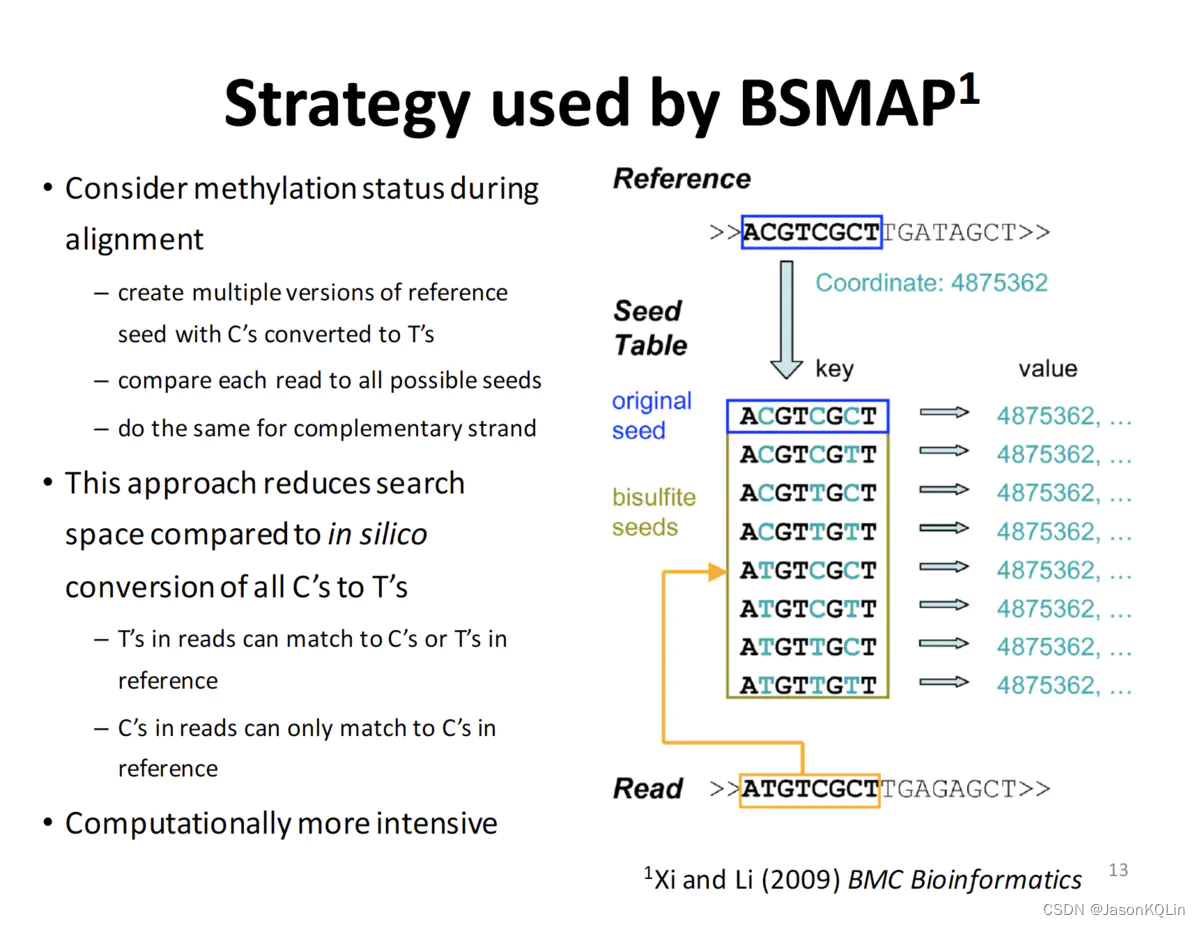
1,bismark比对方法比较简单粗暴,它制作两种类型的基因组:1),将所有的C转化为T的基因组;2),将所有的G转化为A的基因组。它将bisulfate(重chong硫酸盐)处理后的测序reads也进行上述转化,这样也得到两种类型的reads。将两种类型的reads分别比对到两种基因组上,就得到4中比对结果,选择得分最高的比对结果作为最终结果。
示意图如下:
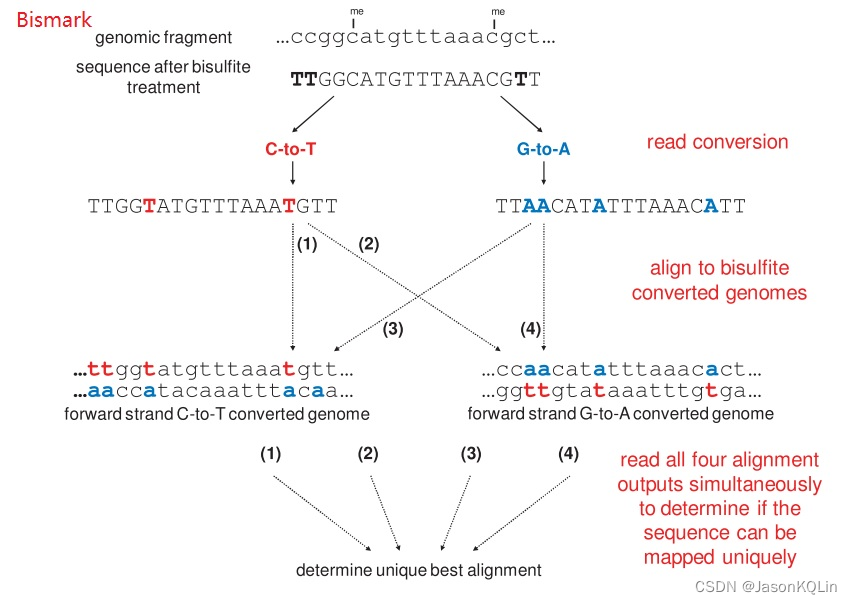
为什么这样做可以?
先看下下面的图:
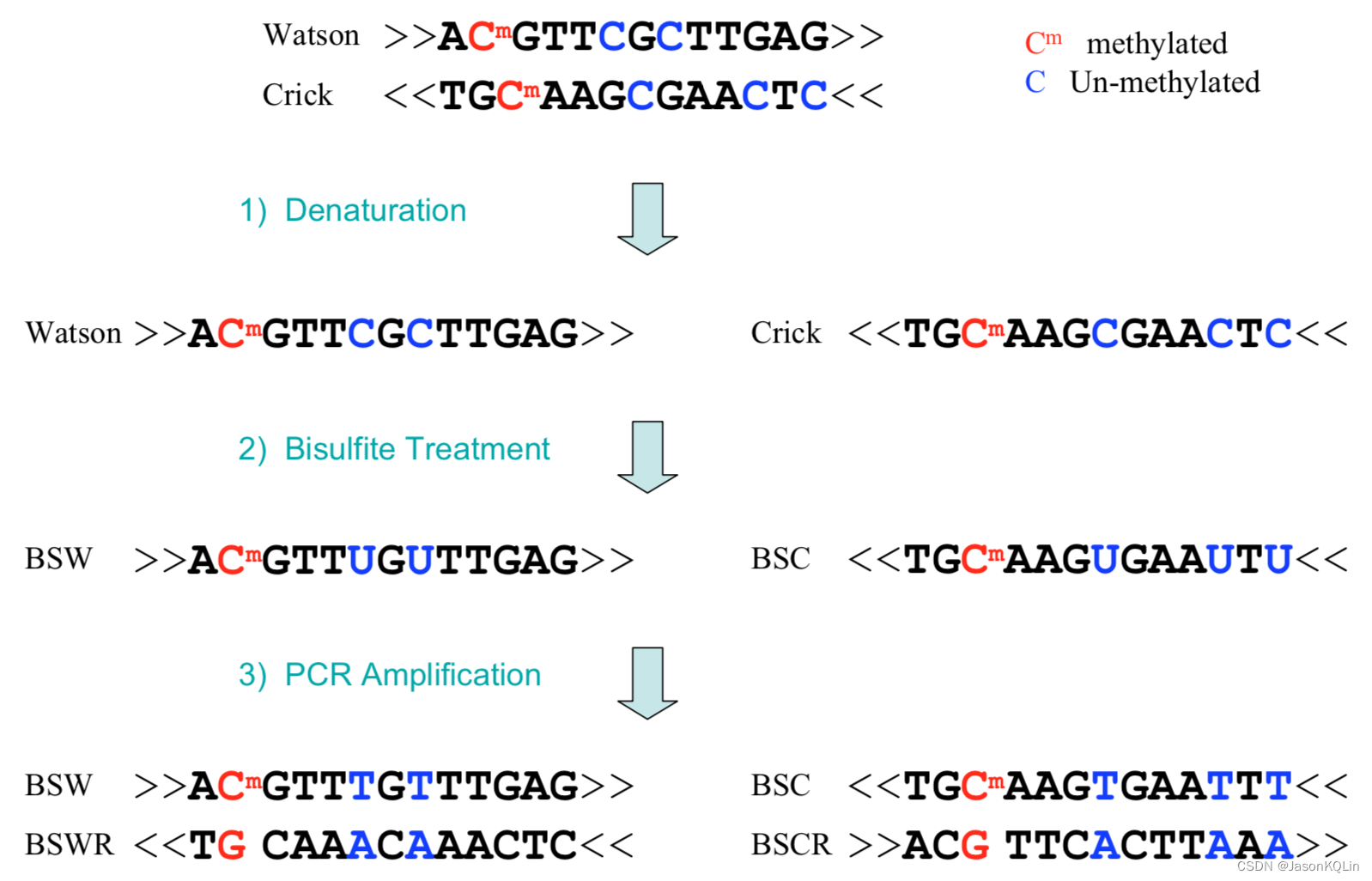
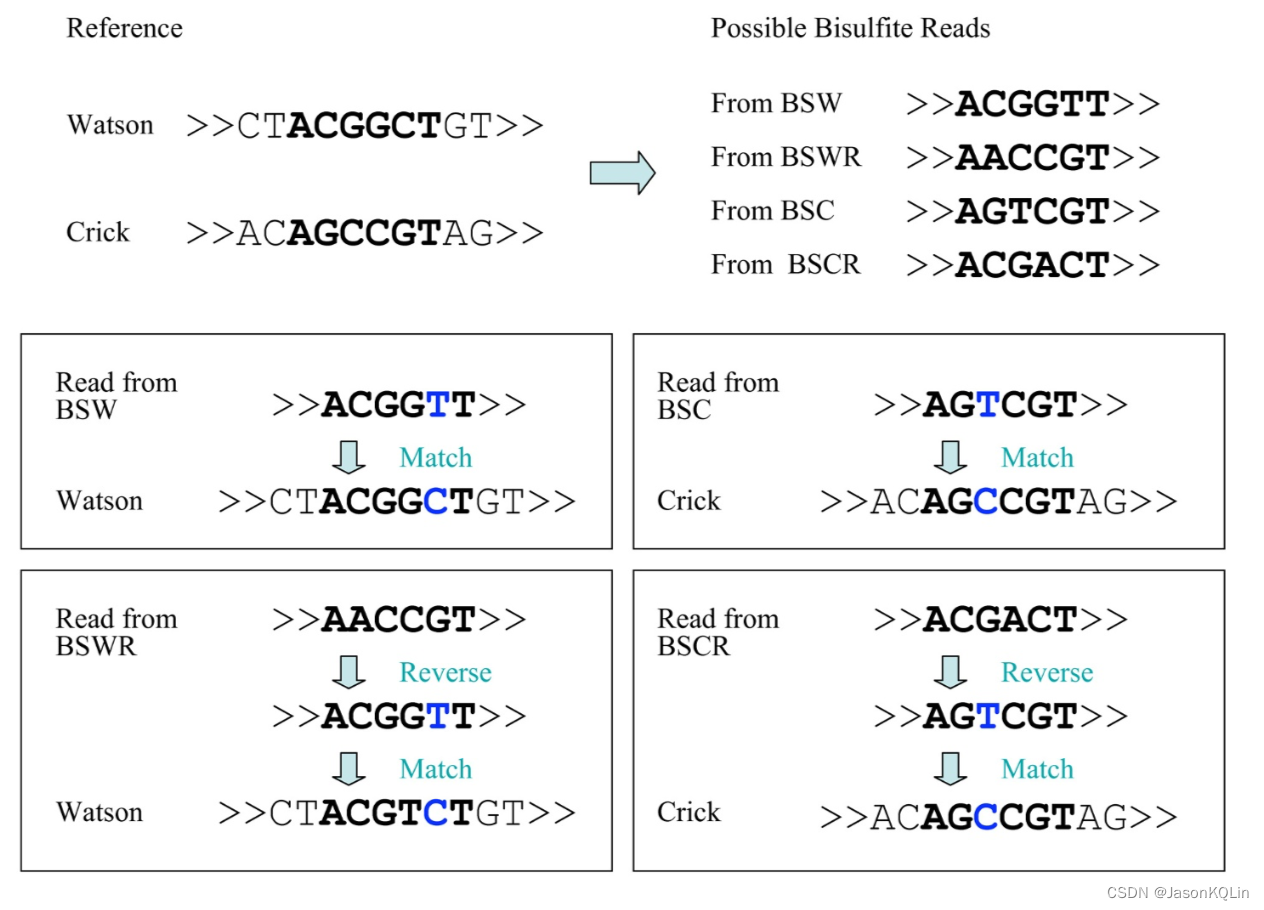
重硫酸盐处理将未甲基化的C转化为U,PCR后U变成A,进而变成T。任意取一条比对后的reads,经过PCR后得到BSW,BSWR,BSC,BSCR四种reads,因为BSW和BSWR完全互补,BSC和BSCR完全互补,我们只需要取BSW和BSCR去查看就好了(如果BSW能比对上,那么BSWR一定也可以比对上)。
参考基因组往往是单链,我们选择Watson链作为参考基因组,它可以制作两种类型的参考基因组:
1,ATGTTTGTTTGAG
2,ACATTCACTTAAA
BSW也可以制作两种:
1,ATGTTTGTTTGAG
2,ACATTTATTTAAA
BSCR也可以制作两种:
1,ATGTTTATTTAAA
2,ACATTCACTTAAA
可以看到BSW的1和BSCR的2分别与参考基因组的1和2完美匹配。
可能导致错误的地方:1,SNP位点;2,PCR引入的错误;3,重硫酸盐导致的突变;4,测序错误。
2,bsmap容许有T比对到C或者T上,而C只能比对到C上,用容许错配的方法代替了上述制作多个基因组的问题。
具体比对过程可以通过下面两种方式实现(bsmap文章中未写明)
1,watson链和crick链都用来做参考基因组,这样只要容许有T比对到C或者T上,就可以实现所有的比对;
2,只用watson链做参考基因组,既需要容许有T比对到C或者T上,又需要容许A比对到A或G上。