input

codes
load(file ="G:/r/duqiang_IPF/GSE70866—true—_BAL_IPF_donors_RNA-seq/expr17077.RData")
head(expr.17077clean)
head(meta.17077)[,1:4]
dim(expr.17077clean)
dim(meta.17077)colnames(meta.17077)=c('time','event','sex','diagnosis')phe=transform(meta.17077,time=as.numeric(time)) %>% transform(event=as.numeric(event))
head(phe)exprSet=expr.17077clean %>% transform(as.numeric()) %>% as.matrix()
head(exprSet)[,1:5]#生存分析
library(survival)
library(survminer)
# 利用ggsurvplot快速绘制漂亮的生存曲线图 根据性别
sfit <- survfit(data=phe,Surv(time, event)~sex)
sfit
head(summary(sfit))
ggsurvplot(sfit, conf.int=F, pval=TRUE)
ggsurvplot(sfit,palette = c("#E7B800", "#2E9FDF"),risk.table =TRUE,pval =TRUE,conf.int =TRUE,xlab ="Time in months", ggtheme =theme_light(), ncensor.plot = TRUE)#挑选感兴趣的基因做差异分析
phe$CBX4=ifelse(exprSet['CBX4',]>median(exprSet['CBX4',]),'high','low')
head(phe)
table(phe$CBX4)
ggsurvplot(survfit(Surv(time, event)~CBX4, data=phe), conf.int=F, pval=TRUE)
ggsurvplot(survfit(Surv(time, event)~phe[,'CBX4'], data=phe), conf.int=F, pval=TRUE)#挑选感兴趣的基因批量做差异分析
gene_interested=readClipboard()
head(gene_interested)
library(stringr)
gene_interested=str_split(pattern = ",",gene_interested)[[1]]
gene_interested=gene_interested[-which(gene_interested=="RAB40A")]
gene_interestedgetwd()
dir.create("G:/r/duqiang_IPF/GSE70866—true—_BAL_IPF_donors_RNA-seq/survival_for_genes-siena")
setwd("G:/r/duqiang_IPF/GSE70866—true—_BAL_IPF_donors_RNA-seq/survival_for_genes-siena")
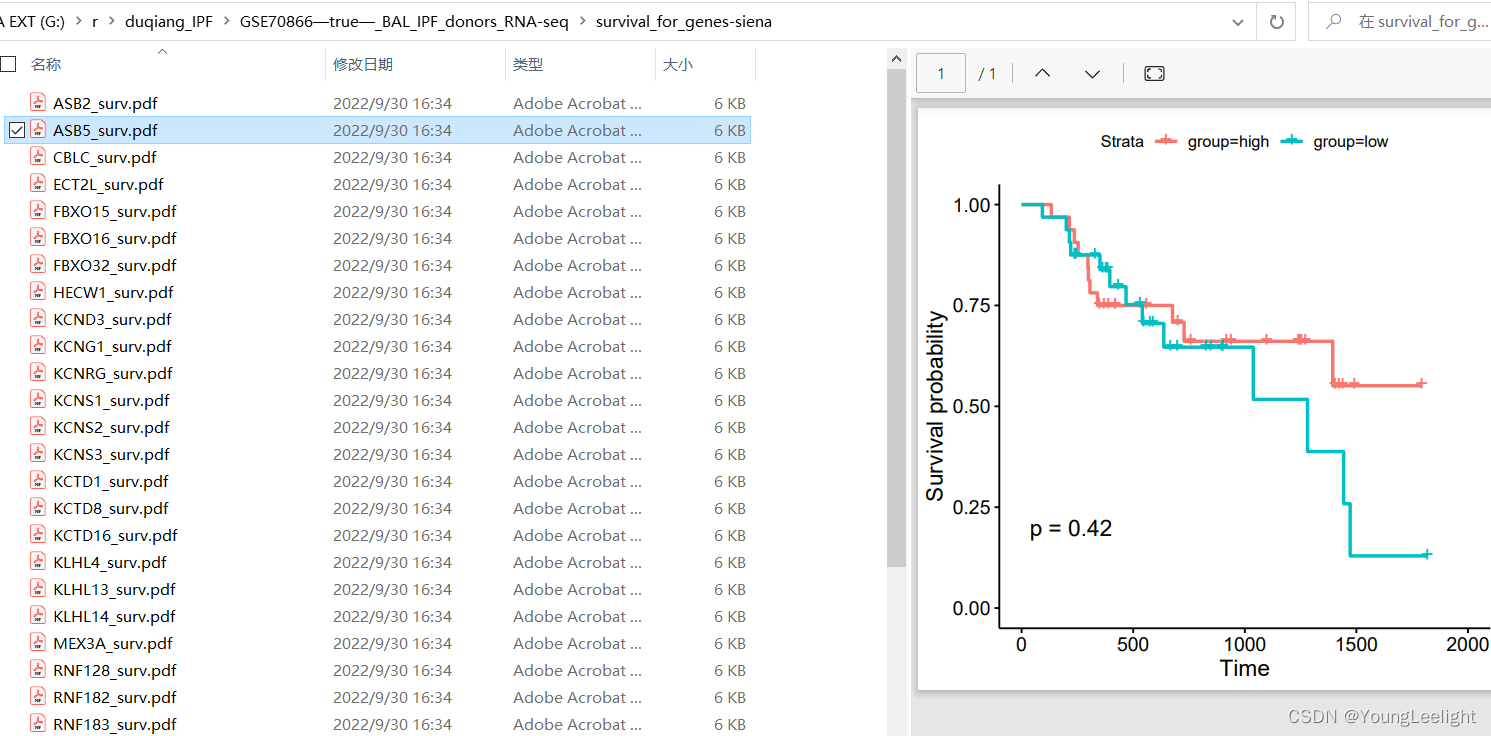
#mySurv=with(phe,Surv(time, event))for (eachgene in gene_interested) {phe$group=ifelse(exprSet[eachgene,]>median(exprSet[eachgene,]),'high','low')p=ggsurvplot(survfit(Surv(time, event)~group, data=phe), conf.int=F, pval=TRUE)pdf(paste0(eachgene, "_surv.pdf"),width = 5, height = 5)print(p, newpage = FALSE)dev.off()
}results结果展示