GWAS工具GAPIT最新版
本篇笔记主要内容是GWAS分析软件GAPIT最新版的安装和使用教程,包括常见的报错以及解决方案,主要出错位置在LDheatmap、stringi、nloptr、lme4等,测试安装的环境是东方天意的ECS云服务器(Linux centos7),R版本为4.2.2,虚拟环境使用conda。
什么是GWAS分析?
GWAS,即基因组关联分析(Genome-wide Association Study),是一种广泛应用于生物医学研究中的遗传学方法。其主要目的是在全基因组水平上,寻找基因与特定性状或疾病之间的关联性,从而揭示其遗传基础。
什么是GAPIT软件?
GAPIT(Genome Association and Prediction Integrated Tool)是一个R语言中的工具包,封装了多种GWAS分析方法,包括基于混合线性模型(MLM)的GWAS、基于广义线性模型(GLM)的GWAS等。GAPIT可以自动处理GWAS数据的预处理、基因型数据的质量控制、单倍型的估计、基因型-表型的联合分析等步骤。
GAPIT具体功能
-
数据预处理:通过对原始数据进行过滤、清洗、转换等操作,构建分析所需的数据集。
-
质量控制:对基因型数据进行清洗,剔除掉质量不好的标记点和个体。
-
单倍型估计:估计个体的单倍型,以便进行基因型-表型关联分析。
-
GWAS分析:使用MLM和GLM模型进行GWAS分析,找出和表型相关的标记点和基因。
-
群体结构控制:通过控制群体结构,减少假阳性结果。
-
基因型-表型关联分析:对经过控制群体结构后的基因型数据进行表型相关性分析,找出和表型显著相关的标记点和基因。
-
数据可视化:通过绘制散点图、直方图、QQ图等图表,直观展示统计结果。
综上所述,GAPIT包是一个功能较为完整的GWAS分析工具,具有数据处理、质量控制、GWAS分析、群体结构控制、基因型-表型关联分析和数据可视化等多种功能,能够方便快捷地进行基因组关联分析。
如何安装GAPIT?
方法一:Github
R> install.packages("devtools")
R> devtools::install_github("jiabowang/GAPIT",force=TRUE)
R> library(GAPIT)
该方法的前提是有网络,而且能访问Github,安装的过程比较友好。
方法二:ZZlab
R> source("http://zzlab.net/GAPIT/gapit_functions.txt")
该方法是直接运行安装函数,也可以访问网站下载后在本地安装。
方法三:本地安装
bash$ git clone git@github.com:jiabowang/GAPIT.git
bash$ R CMD build GAPIT
bash$ R CMD INSTALL GAPIT_3.2.0.9000.tar.gzR> install.packages("GAPIT_3.2.0.9000.tar.gz", repos = NULL, type="source")
该方法是先从Github下载最新的relase文件,然后本地进行安装,适合没有联网的服务器。
- 提示:设置清华大学的CRAN镜像,比官方提供的速度更快
options(repos=structure(c(CRAN="https://mirrors.tuna.tsinghua.edu.cn/CRAN/")))
报错与解决方案
直接运行安装命令后,最终可能会出现如下报错,主要是某些依赖包没有安装,因此需要逐个解决。
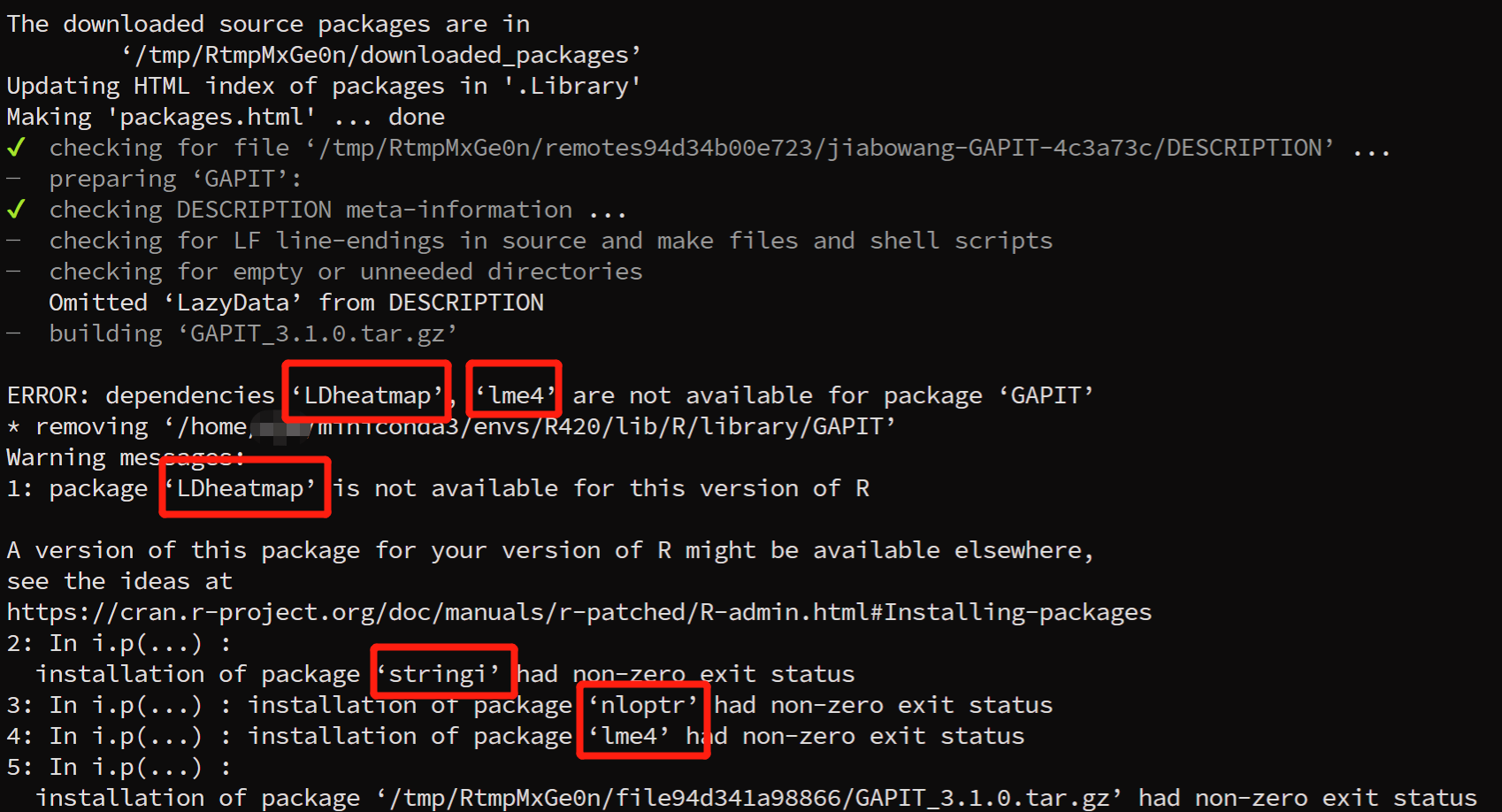
- nloptr
$ conda install r-nloptr
Collecting package metadata (current_repodata.json): done
Solving environment: doneadded / updated specs:- r-nloptrThe following packages will be downloaded:package | build---------------------------|-----------------r-nloptr-2.0.3 | r42hb5eb8f6_0 658 KB defaults------------------------------------------------------------Total: 658 KBThe following NEW packages will be INSTALLED:r-nloptr anaconda/pkgs/r/linux-64::r-nloptr-2.0.3-r42hb5eb8f6_0 Proceed ([y]/n)? Downloading and Extracting PackagesPreparing transaction: done
Verifying transaction: done
Executing transaction: done
- stringi
install.packages("stringi") # 先试试普通安装
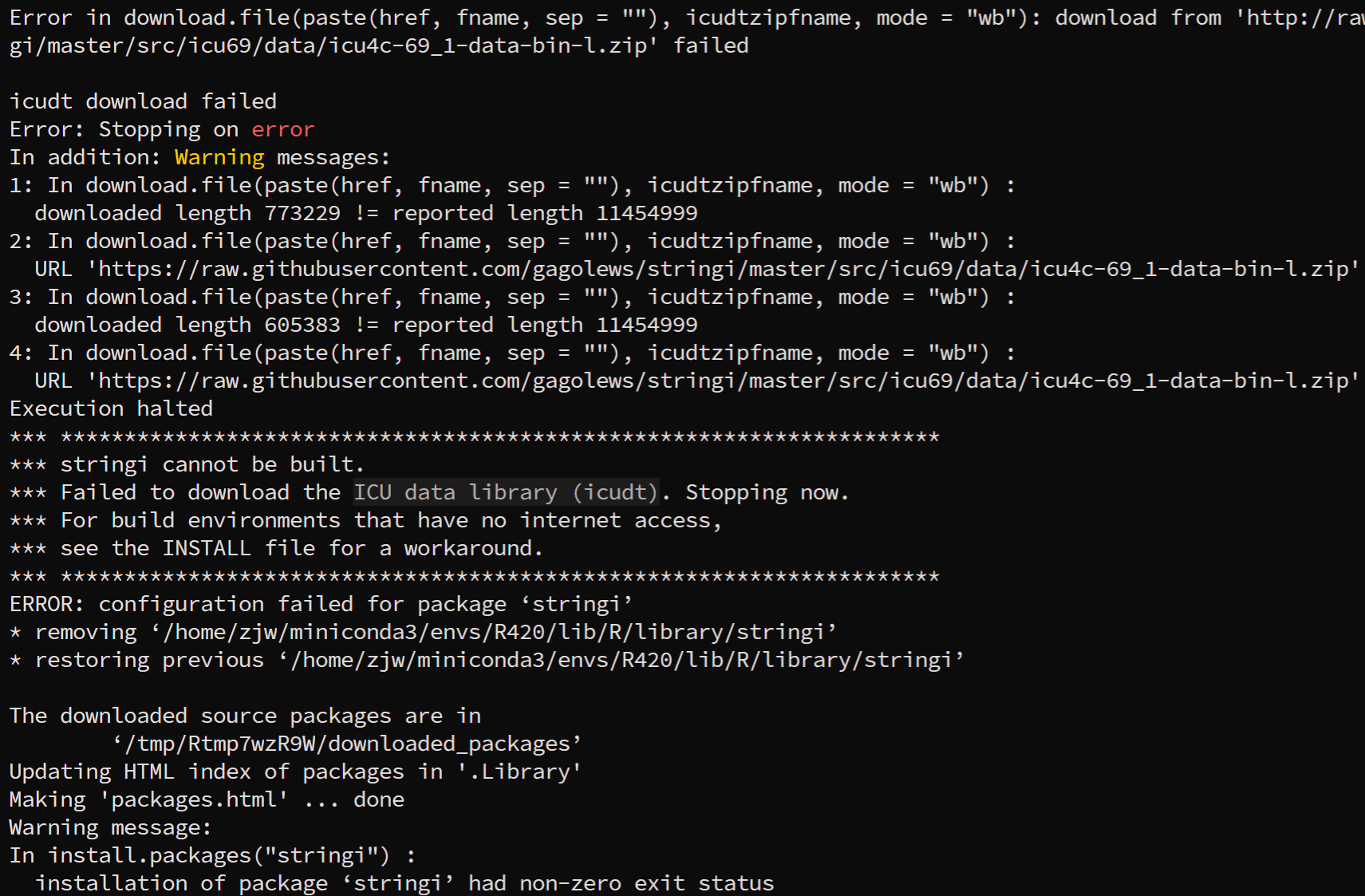
首先,从下面这个网站下载本地安装包,然后在手动安装。补充一点,这个包出错的主要原因是ICU data library (icudt)数据集下载不了,可能是网站自身有关,因此建议本地安装。
https://cran.r-project.org/src/contrib/stringi_1.7.12.tar.gz
下载后进行本地安装,出现下面的报错信息,推测可能是icu数据的链接问题,由于文件较大,网速不行导致断开。
Error: Stopping on error
In addition: Warning messages:
1: In download.file(paste(href, fname, sep = ""), icudtzipfname, mode = "wb") :downloaded length 1413993 != reported length 11454999
2: In download.file(paste(href, fname, sep = ""), icudtzipfname, mode = "wb") :URL 'https://raw.githubusercontent.com/gagolews/stringi/master/src/icu69/data/icu4c-69_1-data-bin-l.zip': Timeout of 60 seconds was reached
3: In download.file(paste(href, fname, sep = ""), icudtzipfname, mode = "wb") :downloaded length 1423942 != reported length 11454999
4: In download.file(paste(href, fname, sep = ""), icudtzipfname, mode = "wb") :URL 'https://raw.githubusercontent.com/gagolews/stringi/master/src/icu69/data/icu4c-69_1-data-bin-l.zip': Timeout of 60 seconds was reached
Execution halted
*** *********************************************************************
*** stringi cannot be built.
*** Failed to download the ICU data library (icudt). Stopping now.
*** For build environments that have no internet access,
*** see the INSTALL file for a workaround.
*** *********************************************************************
ERROR: configuration failed for package ‘stringi’
通过下面的网址可以直接下载该数据集,然后上传到服务器,进行安装
http://raw.githubusercontent.com/gagolews/stringi/master/src/icu69/data/icu4c-69_1-data-bin-l.zip
# icu数据的路径下运行下面一行代码
R CMD INSTALL --configure-vars='ICUDT_DIR=/home/zjw/download/' stringi_1.7.12.tar.gz
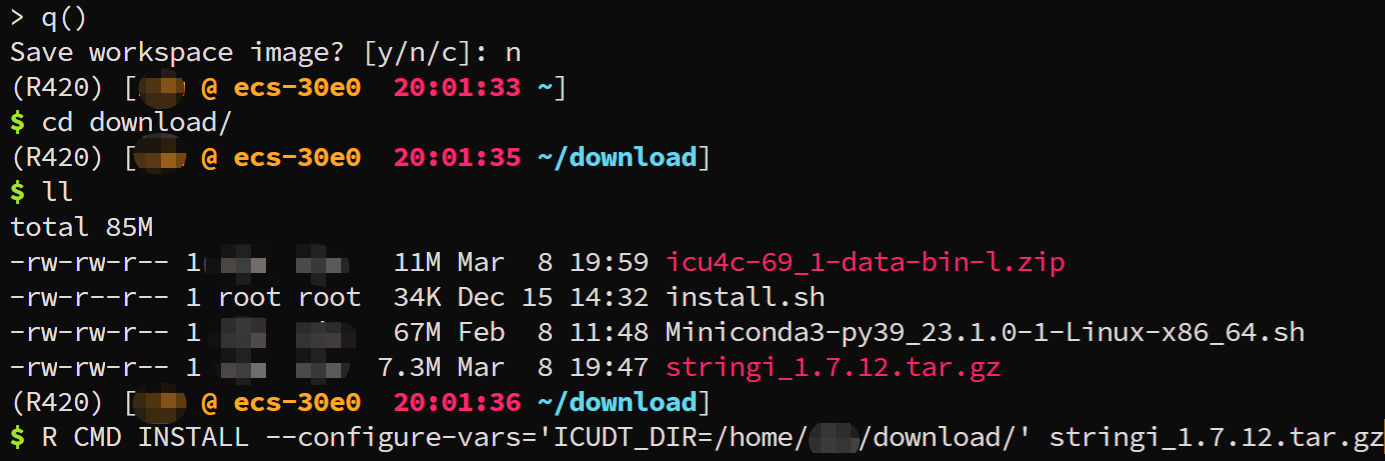
注意:该步骤需要退出R,回到bash进行执行,最终如果出现下面这样的界面说明安装成功
** R
** inst
** byte-compile and prepare package for lazy loading
** help
*** installing help indices** building package indices
** testing if installed package can be loaded from temporary location
** checking absolute paths in shared objects and dynamic libraries
** testing if installed package can be loaded from final location
** testing if installed package keeps a record of temporary installation pathDONE (stringi)
- lme4
$ conda install r-lme4
Collecting package metadata (current_repodata.json): done
Solving environment: doneadded / updated specs:- r-lme4
The following packages will be downloaded:package | build---------------------------|-----------------r-boot-1.3_28 | r42h6115d3f_0 612 KB defaultsr-lattice-0.20_45 | r42h76d94ec_0 1.1 MB defaultsr-lme4-1.1_29 | r42h884c59f_0 4.3 MB defaultsr-matrix-1.4_1 | r42h76d94ec_0 4.2 MB defaultsr-minqa-1.2.4 | r42hb5eb8f6_4 137 KB defaultsr-nlme-3.1_157 | r42h640688f_0 2.2 MB defaultsr-rcppeigen-0.3.3.9.2 | r42h884c59f_0 1.2 MB defaults------------------------------------------------------------Total: 13.7 MBThe following NEW packages will be INSTALLED:r-boot anaconda/pkgs/r/noarch::r-boot-1.3_28-r42h6115d3f_0 r-lattice anaconda/pkgs/r/linux-64::r-lattice-0.20_45-r42h76d94ec_0 r-lme4 anaconda/pkgs/r/linux-64::r-lme4-1.1_29-r42h884c59f_0 r-matrix anaconda/pkgs/r/linux-64::r-matrix-1.4_1-r42h76d94ec_0 r-minqa anaconda/pkgs/r/linux-64::r-minqa-1.2.4-r42hb5eb8f6_4 r-nlme anaconda/pkgs/r/linux-64::r-nlme-3.1_157-r42h640688f_0 r-rcppeigen anaconda/pkgs/r/linux-64::r-rcppeigen-0.3.3.9.2-r42h884c59f_0 Proceed ([y]/n)? Downloading and Extracting PackagesPreparing transaction: done
Verifying transaction: done
Executing transaction: done
- LDheatmap
# Install the latest development version from GitHub with
devtools::install_github("SFUStatgen/LDheatmap")
该方法需要首先安装devtools,但是linux下安装devtools很麻烦,容易出现以下报错信息:
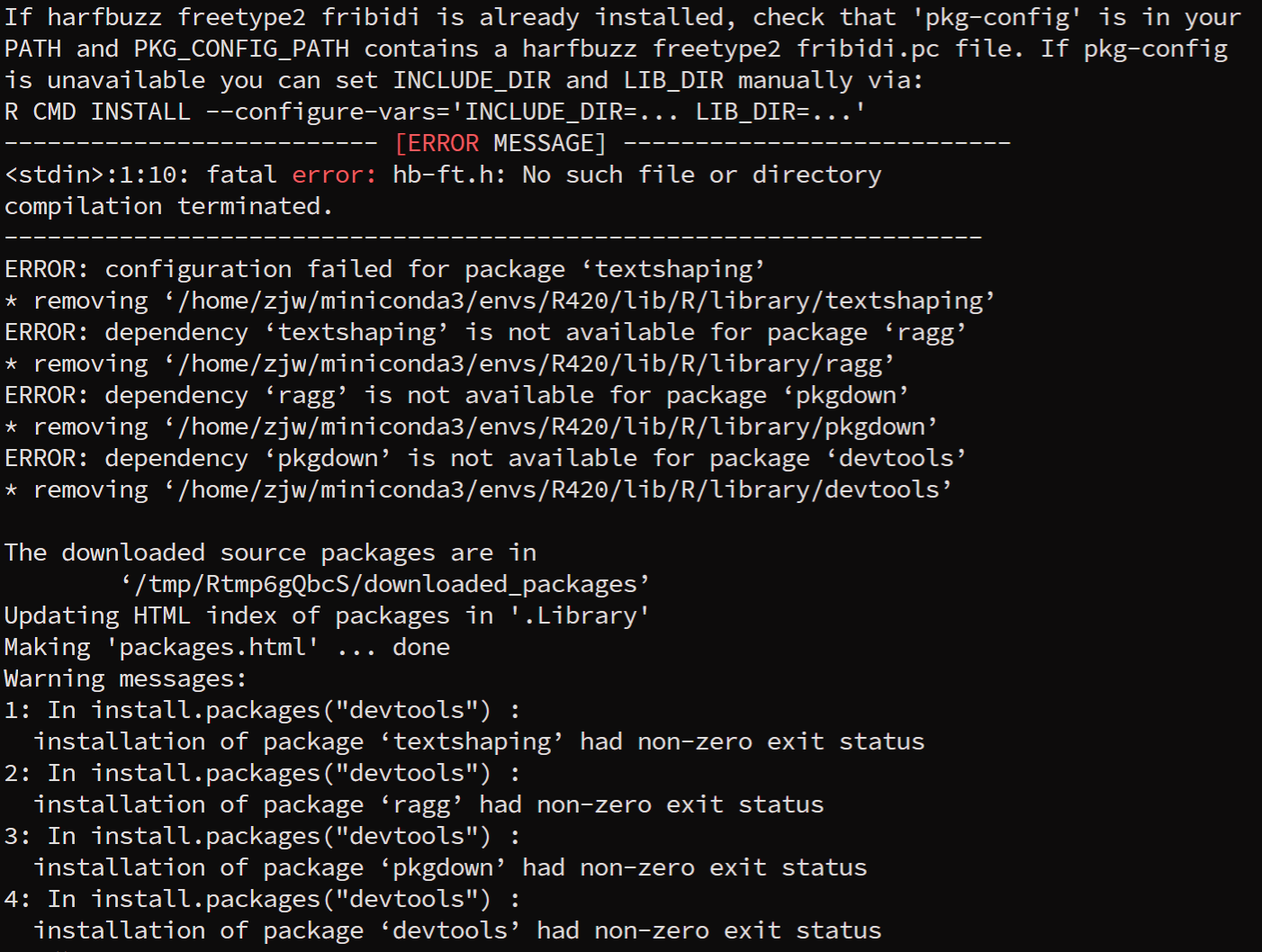
对于centos系统来说,使用sudo yum install harfbuzz-devel fribidi-devel安装依赖库文件,然后依次安装下列几个依赖包
Warning messages:
1: In install.packages("devtools") :installation of package ‘textshaping’ had non-zero exit status
2: In install.packages("devtools") :installation of package ‘ragg’ had non-zero exit status
3: In install.packages("devtools") :installation of package ‘pkgdown’ had non-zero exit status
4: In install.packages("devtools") :installation of package ‘devtools’ had non-zero exit status
安装方法:
conda install r-textshaping
conda install r-ragg
conda install r-pkgdown
conda install r-devtools
R #进入R操作界面
install.packages("devtools")
然后,又出现了新的报错,html相关的版本太低,需要更新,报错信息如下:
Error in loadNamespace(i, c(lib.loc, .libPaths()), versionCheck = vI[[i]]) : namespace ‘htmltools’ 0.5.2 is being loaded, but >= 0.5.4 is required
Calls: <Anonymous> ... namespaceImport -> loadNamespace -> namespaceImport -> loadNamespace
Execution halted
ERROR: lazy loading failed for package ‘devtools’* removing ‘/home/zjw/miniconda3/envs/R420/lib/R/library/devtools’The downloaded source packages are in‘/tmp/RtmpAvUGkq/downloaded_packages’
Updating HTML index of packages in '.Library'
Making 'packages.html' ... done
Warning message:
In install.packages("devtools") :installation of package ‘devtools’ had non-zero exit status
使用下面这行代码进行解决,即可安装成功!
install.packages("htmltools")
install.packages("devtools") #然后再次安装
最后,运行安装LDheatmap的命令,完成安装。
devtools::install_github("SFUStatgen/LDheatmap")
解决了所有的依赖包安装问题,可以再次运行GAPIT安装程序了,我这里使用的是Github方法。
> devtools::install_github("jiabowang/GAPIT",force=TRUE)
Downloading GitHub repo jiabowang/GAPIT@HEAD
编译安装中。。。。。。
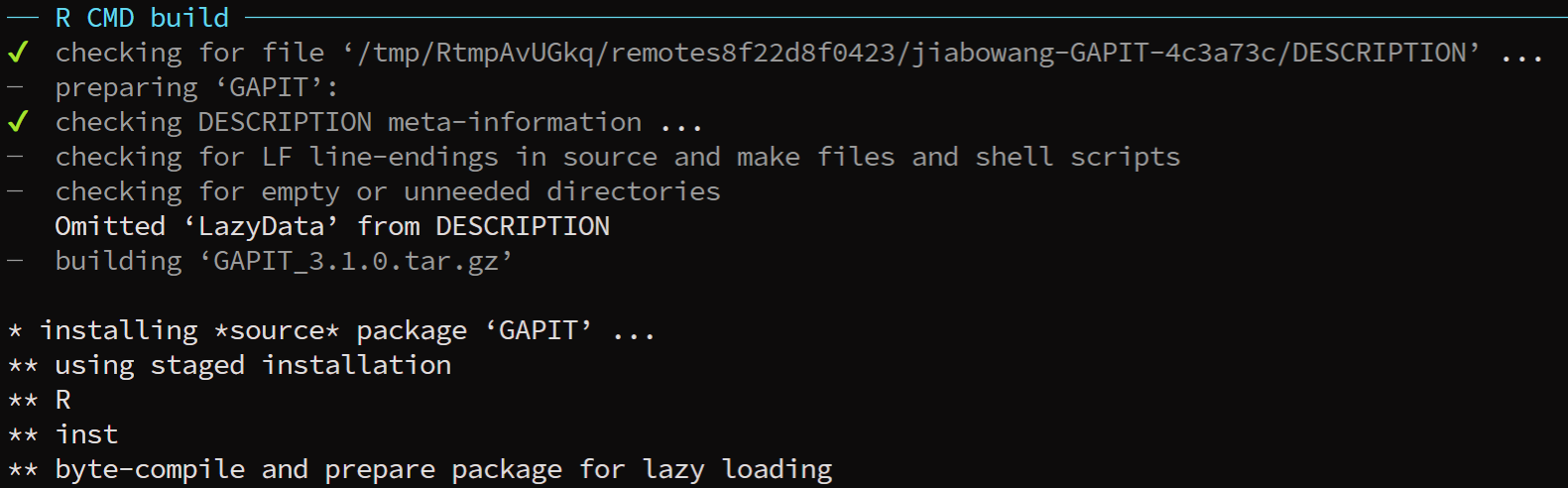
* DONE (GAPIT)> library(GAPIT)> # 没有报错,说明安装成功!yes
建议与提示
- 使用conda先创建一个虚拟环境,再利用conda直接安装r-base
- 安装R包通常有3种方法,官方的install.packages、Github上的包用devtools、下载文件到本地安装
- 如果上面的方法还装不上,试试退出R后用conda安装,说不定会有惊喜
- 依赖包的安装如果碰到系统依赖库文件缺失,需要管理员安装对应的库文件(一般是libxxx.so)
- 编译安装需要tar.gz格式的source资源包,成功率最高
本文由mdnice多平台发布