一、主成分分析
主成分分析是多元统计分析的一种常用的降维方法,它以尽量少的信息损失,最大程度将变量个数减少,且彼此间互不相关。提取出来的新变量成为主成分,主成分是原始变量的线性组合。
1.1 KMO检验和Bartlett球形检验
在进行主成分分析和因子分析之前,需要进行KMO和Bartlett球形检验。当KMO检验系数>0.5,Bartlett球形检验的P值<0.05时,数据才比较适合进行主成分分析或因子分析。这两个检验是用于检查变量的信息重叠度,当检验通过时说明多变量相关性较大,有信息重叠,才会适合做主成分分析降低维度。
KMO检验的实现来自[R] KMO sampling adequacy and SPSS -- partial solution
Bartlett球形检验使用psych包内的cortest.bartlett()函数
- kmo()用法:kmo(data)
kmo()函数只需要输入标准化数据即可,返回的overall为检验系数
- cortest.bartlett()用法:cortest.bartlett(R,n=NULL,diag=TRUE)
其中参数R为相关阵
n为样本量,即观测数量
diag=T时将对角线矩阵换成1,使其成为相关阵
例1:
试对下列数据进行主成分分析
| 序号 | 省份 | 工资性收入 | 家庭性收入 | 财产性收入 | 转移性收入 |
| 1 | 北京 | 4524.25 | 1778.33 | 588.04 | 455.64 |
| 2 | 天津 | 2720.85 | 2626.46 | 152.88 | 79.64 |
| 3 | 河北 | 1293.50 | 1988.58 | 93.74 | 105.81 |
| 4 | 山西 | 1177.94 | 1563.52 | 62.70 | 86.49 |
| 5 | 内蒙古 | 504.46 | 2223.26 | 73.05 | 188.10 |
| 6 | 辽宁 | 1212.20 | 2163.49 | 113.24 | 201.28 |
注:以上仅为部分表格
> data2=read.csv('Table_0.csv',encoding='UTF-8')
> rownames(data2)=data2[,1]
> data2=data2[,-1]
> st.data2=scale(data2) #标准化数据
> library(psych)
> source("kmo.R")
> kmo(st.data2)$overall #kmo检验
[1] 0.7404854
> cortest.bartlett(cor(st.data2),nrow(st.data2)) #巴特利特球形检验
$chisq
[1] 68.86313$p.value
[1] 6.992923e-13$df
[1] 6
由检验结果可见,kmo检验系数大于0.5,Bartlett球形检验的P值极小
说明这个数据的变量信息重叠较多,适合进行主成分分析和因子分析
1.2 主成分分析函数介绍
R语言中进行主成分分析的函数有自带的princomp()函数,也有psych包内的principal()函数。两个函数虽然都是用于主成分分析,但是两个函数会有所区别。本文会同时介绍两个函数
- princomp()用法:princomp(x,cor=FALSE,scores=TRUE,…)
princomp()也有formula参数用法,但是我们较少使用,所以只介绍默认用法。
x是数据矩阵或数据框,通常要先进行标准化
cor参数是一个逻辑值,当cor=T时使用相关阵进行主成分分析,默认cor=F,此时用协差阵进行主成分分析
scores参数也是一个逻辑值,表示是否计算主成分得分
- principal()用法:principal(r,nfactors=1,rotate=”varimax”,n.obs=NA,scores=TRUE,…)
r可以是数据矩阵或数据,也可以是相关阵
rotate参数指定主成分旋转方法,默认为最大方差法,其他的方法还有
”none”不进行旋转
“quartimax”、”promax”、”oblimin”、”simplimax”、”cluster”
简单的主成分分析的旋转方法除了”none”和”varimax”使用较多外,其他都较少使用
scores参数的用法和princomp()函数里的scores参数相同,都是表示是否计算主成分得分
n.obs是原始数据的样本量,也就是观测的个数。当r是相关阵时需要指定n.obs,但如果r是原始数据则不用指定
principal()与 princomp() 不同,它只返回最佳主成分个数的子集。特征向量按特征值的开方重新缩放,以产生在因子分析中更典型的分量载荷。principal()需要提前确定最佳主成分个数,而princomp()是直接把所有主成分提取出来,再通过方差累计贡献率确定主成分个数。所以在使用principal()进行主成分分析之前,我们需要通过一些方法确定主成分的个数
1.3 确定主成分个数(principal)
提前确定主成分个数的方法,无外乎画碎石图,我们可以用同样来自于psych包内的fa.parallel()函数来确定。fa.parallel()不仅可以用于确定主成分个数,也可以用于确定因子分析时因子的个数,这个函数在下文的因子分析也有用到
- fa.parallel()用:fa.parallel(x,n.obs=NULL,fm=”minres”,fa=”both”,…)
x可以是数据矩阵或数据框,也可以是相关阵
n.obs的用法和principal()内的n.obs用法相同,也就是当x取相关阵时需要指定的观测个数
fm指定提取因子的方法,默认为”minres”极小残差法。此外还可以选择
“ml”——极大似然法;“pa”——主轴迭代法
“wls”——加权最小二乘法;”gls”——广义最小二乘法
提取因子的方法用极大似然法计算会比较快,但是在某些情况可能不收敛,选用主轴迭代法会比较稳妥。这个主要在因子分析时会用到。
fa指定提取主成分还是因子,fa=”pc”时只提取主成分,fa=”fa”时只提取因子,fa=”both”时主成分和因子都提取
碎石图评估主成分个数的具体方法时查看高度为1的横线或两条红色虚线上方的散点个数。横线是特征值为1的高度,红色虚线是随机数据矩阵的平均特征值。在主成分或因子个数增加的时候,如果真实数据特征值低于随机数据的平均特征值,这时候说明之后的因子或主成分没有保留的价值。
续上例:
> fa.parallel(st.data2,fm='pa',fa='pc') #只确定主成分个数
Parallel analysis suggests that the number of factors = NA and the number of components = 1
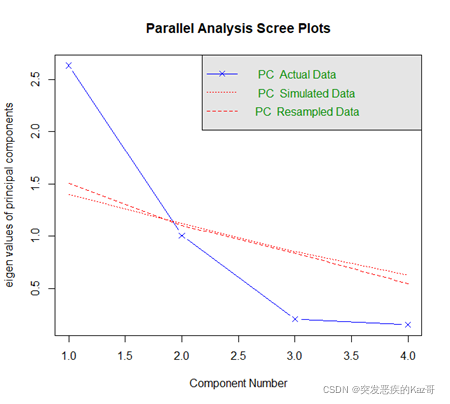
在随机数据平均特征值以上的只有第一特征值,函数推荐保留一个主成分。但是第二个特征值离随机数据的平均特征值也不远,而且到第三特征值的下降程度还比较大,只保留一个主成分的建议还比较存疑。到底需不需要只保留一个主成分可以在主成分分析完后查看方差累计贡献率确定。
1.4 进行主成分分析
如果用princomp()进行主成分分析可以跳过前一步,这个函数的主成分个数确定在主成分分析之后。而用principal()进行主成分分析则需要提前确定主成分个数。
对于princomp()的主成分分析结果,需要用summary()函数获取各个主成分的方差、方差累计贡献率和载荷阵,查看可以通过princomp对象的loadings组件获取,也可以在summary()函数内加入loadings参数获取。
而principal()的主成分分析结果,查看储存结果的变量可以获取大部分信息,只查看载荷阵和方差累计贡献率也可以通过principal对象的loadings组件获取。
续上例:
> prc1=princomp(st.data2,cor=T) #用princomp进行的主成分分析
> prc2=principal(st.data2,nfactor=1) #用principal进行的主成分分析
> summary(prc1,loadings=T) #查看princomp的主成分分析结果
Importance of components:Comp.1 Comp.2 Comp.3 Comp.4
Standard deviation 1.6221839 1.0039536 0.45568337 0.39108742
Proportion of Variance 0.6578701 0.2519807 0.05191183 0.03823734
Cumulative Proportion 0.6578701 0.9098508 0.96176266 1.00000000Loadings:Comp.1 Comp.2 Comp.3 Comp.4
工资性收入 0.582 0.314 0.749
家庭性收入 0.989 0.117
财产性收入 0.568 0.124 -0.806 -0.111
转移性收入 0.579 0.488 -0.651#Cumulative Proportion是方差累计贡献率
#可见在第二个主成分以及到达较高水平> prc2$loadings #查看principal对象的载荷阵和累计方差贡献率Loadings:PC1
工资性收入 0.944
家庭性收入 -0.102
财产性收入 0.921
转移性收入 0.939PC1
SS loadings 2.631
Proportion Var 0.658 #单个主成分的累计方差贡献率只有65.8%,不高
#这说明自提取一个主成分有点不太合适
#再用principal进行两个主成分的主成分分析
> prc2=principal(st.data2,nfactor=2)
> prc2$loadingsLoadings:RC1 RC2
工资性收入 0.945
家庭性收入 0.998
财产性收入 0.928
转移性收入 0.931 -0.136RC1 RC2
SS loadings 2.622 1.018
Proportion Var 0.655 0.254
Cumulative Var 0.655 0.910 #两个主成分的累计贡献率达到91%的较高水平
1.5 确定主成分个数(princomp)
princomp()的主成分分析会提取所有主成分,确定其主成分个数我们要从累计方差贡献率或碎石图确定。
对princomp对象使用summary()函数获取累计方差贡献率,Cumulative Proportion就是累计方差贡献率,通常取主成分个数使累计方差贡献率达到一个较高的百分数(如85%以上)。
确定princomp()的主成分个数也可以通过画碎石图确定,画princomp对象碎石图的函数为screeplot()
- screeplot()用法:screeplot(x,npcs=min(10,length(x$sdev)),type=c(“barplot”,”lines”),…)
x是princomp()的主成分分析结果
npcs是需要绘制的主成分个数,默认取10和x全部主成分个数之间的最小值
type指定绘图的类型,type=”barplot”时绘制直方图,type=”line”时绘制折线图
这个函数绘制的碎石图没有随机数据的平均特征值作为参考。我们可以通过下降程度或绘制特征值为1的水平线来判断。再特征值1的水平线上的散点个数,或者下降到一个较低水平的主成分之前的个数。
续上例:
#prc1是princomp对象
#上文以通过summary()函数已经到两个主成分时到达了较高的累计贡献率
> screeplot(prc1,type='line')
> abline(1,0,col='blue') #添加特征值1水平线
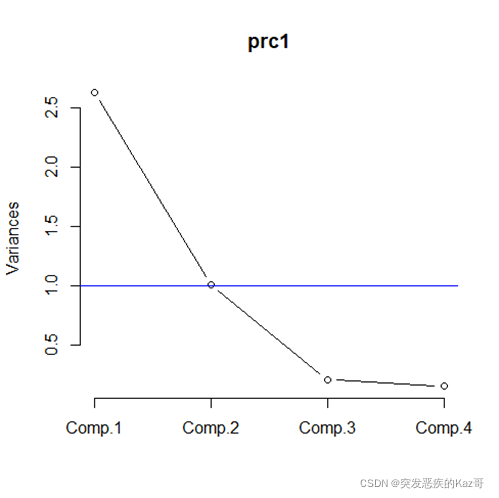
由图看,选取两个主成分是比较合适的
1.6 获取主成分得分以及进行综合评价
1.6.1 获取主成分得分
进行主成分分析的主要目的是对数据进行降维,确定好主成分个数后,可以通过scores组件获取原数据在各个主成分上的得分,并用主成分的得分代替原变量进行其他分析
主成分的得分就是各个样品主成分的值,主成分是标准化后的原始变量的线性组合,将每个样品标准化原始变量的值代入主成分的表达式里也可以获得主成分得分。
princomp对象会返回所有主成分的得分,我们只提取需要的前几个主成分得分即可
principal对象只返回m个主成分的得分,m记为我们确定的主成分个数
续上例:
> head(prc1$scores[,1:2])Comp.1 Comp.2
北京 4.58987415 0.3734869
天津 0.28101544 1.7953881
河北 -0.44296144 0.3489825
山西 -0.65163291 -0.5607793
内蒙古 -0.60834766 0.7533096
辽宁 -0.05120586 0.6893062
> head(prc2$scores)RC1 RC2
北京 2.80352044 0.1472879
天津 0.30740034 1.7405353
河北 -0.24107468 0.3619058
山西 -0.43690909 -0.5169196
内蒙古 -0.31009603 0.7647188
辽宁 0.02183515 0.6757876
1.6.2 进行综合评价
另外,我们可以利用主成分分析的得分对各个样品进行综合评价。主成分分析能从选定的指标体系中归纳出大部分信息,根据主成分提供的信息进行综合评价是一种可行的选择。
每个样品的综合评价得分是主成分得分的加权和,每个主成分的权重等于所属特征值除以m个特征根的和,这里m指所选主成分个数。
对princomp()和principal()两个函数主成分分析结果,进行综合评价的函数实现为如下,该函数同时对每个样品的综合评价得分进行了排序
evaluation=function(prin,data=NULL,nfactor=NULL){#当prin是princomp返回的主成分分析结果时,data和nfactor不可缺#data是标准化数据,用于获取特征值#nfactor是选择的主成分个数which=class(prin)[length(class(prin))]if(which=='princomp') {if(is.null(data)|is.null(nfactor)) {print('需输入原始数据和主成分个数');stop()}else{values=eigen(cor(data))$values[1:nfactor]w=values/sum(values);scores=prin$scores[,1:nfactor]}}#当prin是psych包内的principal函数返回的主成分分析结果时#特征值和主成分个数可以通过调用组件获取else if(which=='principal') {scores=prin$scores;nfactor=ncol(scores)values=prin$values[1:nfactor];w=values/sum(values)}eva.scores=scores%*%wfinal.eva=as.numeric(eva.scores)names(final.eva)=rownames(eva.scores)sort(final.eva,T)
}
续上例:
#对各个样品进行综合评价
> source(“evaluation.R”)
> evaluation(prc1,st.data2,2)上海 北京 浙江 江苏 福建 天津 广东 黑龙江 4.0900564 3.4221571 2.1177097 0.8304684 0.7029739 0.7004168 0.6631721 0.3083484 吉林 辽宁 山东 河北 内蒙古 海南 西藏 湖南 0.1738328 0.1538769 0.0554438 -0.2236347 -0.2312404 -0.3985562 -0.4122491 -0.4853280 宁夏 重庆 四川 湖北 江西 山西 河南 新疆
-0.5463658 -0.6151700 -0.6180057 -0.6203514 -0.6264414 -0.6264713 -0.7128601 -0.7134170 青海 安徽 云南 陕西 广西 甘肃 贵州
-0.7150107 -0.7755070 -0.8726009 -0.9127242 -0.9854052 -1.0570477 -1.0700698
> evaluation(prc2)上海 北京 浙江 天津 江苏 福建 黑龙江 2.08066022 2.06788405 1.59581510 0.70430316 0.60117700 0.58466717 0.37857458 广东 吉林 辽宁 山东 内蒙古 海南 河北 0.36999921 0.29433837 0.20294550 0.18165695 -0.01242897 -0.06809526 -0.07408087 湖北 新疆 湖南 江西 河南 四川 宁夏
-0.29972298 -0.32625842 -0.33136881 -0.38913165 -0.39577186 -0.42128328 -0.42511876 西藏 山西 重庆 安徽 青海 云南 广西
-0.44160741 -0.45906777 -0.46972445 -0.57218090 -0.58965530 -0.61776827 -0.69206531 陕西 甘肃 贵州
-0.78365886 -0.82534403 -0.86768814
二、因子分析
因子分析和主成分分析有点相似,但两种分析的出发点和结果都不同。主成分分析是试图寻找原变量的线性组合,使得这个组合的方差最大,使其携带的信息最多。因子分析是寻找对原变量都有影响的潜在变量,这种潜在变量往往难以量化,这个潜在变量称为公共因子,公共因子可以赋予一定的现实意义。
R中进行因子分析的函数是psych包内的fa()函数。psych包内的部分函数在上文的主成分分析中也有介绍。这个函数进行的为R型因子分析,Q型因子分析此处不做介绍。
2.1 KMO检验和Bartlett球形检验
在进行因子分析和主成分分析之前都需要进行KMO和Bartlett球形检验。这两个分析的介绍在上文的主成分分析已经给出,此处不赘述
例2:
对上一例题的数据进行因子分析
由上文进行主成分分析之前的kmo和Bartlett球形检验结果,可知该数据适合做因子分析
2.2 确定因子个数
上文介绍了psych包内的fa.parallel()函数,这个函数通过画原始数据和随机数据的碎石图来确定主成分或因子个数。我们令fa.parallel()的fa参数为”fa”,这样绘制的是因子的碎石图,用于确定因子个数
续上例:
> fa.parallel(st.data2,fm='pa',fa='fa')
Parallel analysis suggests that the number of factors = 1 and the number of components = NA
There were 32 warnings (use warnings() to see them)
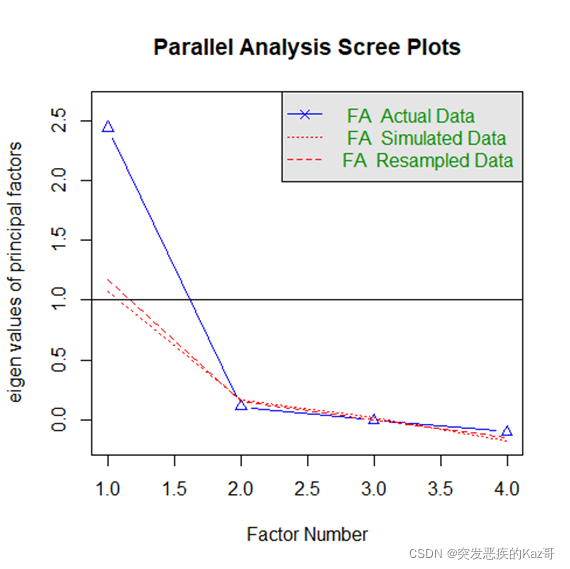
函数建议的因子个数为1个,从碎石图看也是1个比较合适。暂且先以1个因子进行因子分析
2.3 进行因子分析
进行因子分析需要用fa()函数
- fa()用法:fa(r,nfactors=1,n.bos=1,rotate=”oblimin”,fm=”minres”,…)
这些参数与主成分分析中的principal()函数的参数用法相同
r为标准化数据或相关阵,nfactors指定因子个数
n.bos指定样本量,当r是相关阵时需要指定
rotate是因子旋转方法,默认为”oblimin”斜交转轴法,我们常用的是”varimax”最大方差法或”none”不旋转
fm指定提取公共因子的方法,默认为”minres”极小残差法。此外还可以选择
“ml”——极大似然法;“pa”——主轴迭代法
“wls”——加权最小二乘法;”gls”——广义最小二乘法
fa()函数返回的分析结果有以下常用组件
communality——公共因子方差
values——特征根
loadings——载荷阵及因子方差贡献率
scores——因子得分
rot.mat——因子旋转矩阵
直接查看存储了因子分析结果的变量可以获取载荷阵、共同度等信息
续上例:
> FA1=fa(st.data2,nfactors=1,rotate='varimax',fm='pa')
> FA1$loadingsLoadings:PA1 #根据载荷阵可以知道因子与哪些原始变量关系密切
工资性收入 0.928
家庭性收入 #家庭性收入的载荷太小不显示
财产性收入 0.866 #这可能导致因子解释不够充分
转移性收入 0.910PA1
SS loadings 2.444
Proportion Var 0.611 #一个因子只解释了原始数据61.1%的信息
#尝试提取两个公共因子
> FA2=fa(st.data2,nfactors=2,rotate='varimax',fm='pa')
> FA2$loadingsLoadings:PA1 PA2
工资性收入 0.910
家庭性收入 0.396 #第二个公共因子对家庭性收入的载荷也不高
财产性收入 0.897
转移性收入 0.905 -0.291PA1 PA2
SS loadings 2.451 0.253
Proportion Var 0.613 0.063
Cumulative Var 0.613 0.676 #累计方差贡献率依然不高
#两个因子和一个因子对原始数据的解释程度相差不大
#这样只取一个因子会比较好
2.4 构建新的指标体系以及获取得分
通过载荷阵查看公共因子与原始变量的载荷,在一个公共因子上载荷较大的原始变量,它与这个公共因子的关系会比较密切。我们可以根据载荷高的变量对公共因子赋予一定的现实意义,以此构建新的指标体系
例如以高中各个学科成绩为原始变量的指标体系,提取两个公共因子,第一公共因子对数学、物理、化学的载荷较大,第二公共因子对语文、英语、历史、政治的载荷较大,那么我们可以将第一公共因子定义为理科能力,第二公共因子定义为文科能力,并将原始的指标体系简化为由理科能力和文科能力两个新指标组成
由于公共因子也是标准化原始数据的线性组合,根据载荷可以计算出每个样品的因子得分。对fa()函数返回的因子分析结果,我们也可以通过查看scores组件获得因子得分
续上例:
#以FA1为例
#因为只有一个因子,原始变量都是收入相关
#可以将公共因子定义为收入水平
> head(FA1$scores) #展示前6个样品的因子得分PA1
北京 2.520412846
天津 0.281677988
河北 -0.242683221
山西 -0.383635155
内蒙古 -0.347368818
辽宁 -0.009437811
三、相应分析
相应分析又称为对应分析,其是对两个定性变量的多水平进行相应研究,查看它们之前的内在联系。相应分析也是联系R型因子分析和Q型因子分析的桥梁
相应分析基于计数列联表数据,在R中进行简单相应分析,可以用MASS包中的corresp()函数,也可以用ca包中的ca()函数
以列联表出发进行简单相应分析的步骤很简单,只需三步。,一是对列联表进行独立性检验,二是用相应分析的函数进行相应分析,三是用画图函数画出相应分析图,将各个水平在相应分析图中表示出来,离得近的就是不同因素的比较类似的水平
3.1 构建列联表
有时候我们拿到的数据是每个观测的未分组数据,此时我们需要用table()函数对每个水平进行计数,构建列联表。
- table()用法:table(x,y,…)
x与y就是我们需要进行计数的变量,这两个变量通常需要先转化成因子
例3:
试对1991 U.S.GSS数据中的race(种族)和Happy(幸福)进行相应分析, 并说明它们两者的对应关系
| sex | race | region | happy | life | sibs |
| 2 | 1 | 1 | 1 | 1 | 1 |
| 2 | 1 | 1 | 2 | 1 | 2 |
| 1 | 1 | 1 | 1 | 0 | 2 |
| 2 | 1 | 1 | 9 | 2 | 2 |
| 2 | 2 | 1 | 2 | 1 | 4 |
注:以上仅为部份表格
> data3=read.csv('1991 U.S. General Social Survey.csv')
> data3=data3[,c(2,4)] #提取种族和幸福两个变量
> data3[,1]=as.factor(data3[,1])
> data3[,2]=as.factor(data3[,2])
> head(data3)race happy
1 1 1
2 1 2
3 1 1
4 1 9
5 2 2
6 2 2
> attach(data3)
> table3=table(race,happy) #做列联表
> table3happy
race 1 2 3 91 409 730 117 82 46 116 39 33 12 26 9 2
3.2 独立性检验
进行相应分析前需要对行列因子进行独立性检验。如果行列独立,说明这两个因素相互没有影响,也就没有做相应分析的必要了。在R中,进行独立性检验的函数主要是chisq.test(),用的是皮尔逊卡方检验。此外还可以用Deducer包内的likelihood.test进行似然比独立性检验
- chisq.test()用法:chisq.test(x,y=NULL,…)
x可以是矩阵、数据框、表,也可以是因子(factor)。当x是矩阵或数据框或表时,内容应该是计数数据,即x本质上应该是列联表,此时是对列联表的行列因子进行独立性卡方检验
y是因子(factor),当x是列联表时可以忽略这个参数。当x和y都是因子时,会对x和y的独立性进行检验。
- likelihood.test()用法:likelihood.test(x,y=NULL,…)
这个函数的用法与chisq.test()相同,不过需要注意的是Deducer包的加载需要Java环境,没有安装Java的电脑可能会加载失败
续上例:
> chisq.test(table3)Pearson's Chi-squared testdata: table3
X-squared = 32.237, df = 6, p-value = 1.47e-05Warning message:
In chisq.test(table3) : Chi-squared approximation may be incorrect
#P值<0.05,但是给出了卡方近似可能有误的警告,结果可能不可信
#更换检验函数,使用Deducer包内的likelihood.test()
#这个函数进行似然比独立检验
> library(Deducer)
> likelihood.test(table3)Log likelihood ratio (G-test) test of independence without correctiondata: table3
Log likelihood ratio statistic (G) = 27.146, X-squared df = 6, p-value = 0.0001359
#P值依然小于0.05,说明种族与幸福确实不独立
3.3 进行相应分析
进行简单相应分析可以使用MASS包内的corresp()函数,也可以使用ca包中的ca()函数,个人比较推荐使用ca()函数
- corresp()用法:corresp(x,nf=1,…)或corresp(x,y,nf=1,…)
第一种用法中,参数x是矩阵或数据框,这个函数没有对表类(table类)对象的用法
第二种用法中,x和y都是因子
nf指定提取公共因子数量,也就是绘图时的维度,只有当nf大于或等于2时才能绘制相应分析图,通常都会取nf=2
分析结果直接查看存储结果的变量即可
- ca()用法:ca(obj,…)
obj可以是数据框、矩阵、表,本质上是列联表
这个函数会算出相关矩阵的所有特征值,也就是提取出所有公共因子,我们可以根据特征值来看累计贡献率,以此确定公共因子数。但是这对我们画图没有影响,画图默认使用前两个公共因子
分析结果用summary()查看
续上例:
> CA1=corresp(race,happy,nf=2)
> CA2=ca(table3)
> CA1
First canonical correlation(s): 0.13874525 0.04472209 x scores:[,1] [,2]
1 -0.4450141 0.04605089
2 2.0997831 -1.42379260
3 2.7375930 4.73970128y scores:[,1] [,2]
1 -0.81133437 0.4891867
2 -0.08354719 -0.2131040
3 2.37903773 -1.0140261
9 4.55423642 9.5915978
> summary(CA2)Principal inertias (eigenvalues):dim value % cum% scree plot 1 0.019250 90.6 90.6 *********************** 2 0.002000 9.4 100.0 ** -------- ----- Total: 0.021250 100.0 Rows:name mass qlt inr k=1 cor ctr k=2 cor ctr
1 | 1 | 833 1000 150 | -62 999 165 | 2 1 2 |
2 | 2 | 134 1000 563 | 291 954 593 | -64 46 273 |
3 | 3 | 32 1000 288 | 380 763 242 | 212 237 726 |Columns:name mass qlt inr k=1 cor ctr k=2 cor ctr
1 | 1 | 308 1000 191 | -113 964 203 | 22 36 74 |
2 | 2 | 575 1000 6 | -12 597 4 | -10 403 26 |
3 | 3 | 109 1000 568 | 330 981 616 | -45 19 112 |
4 | 9 | 9 1000 235 | 632 685 178 | 429 315 788 |
#CA1返回的是因子得分,继续分析需要画相应分析图
#关心CA2的维度,发现第二个维度已经解释了100%的方差
3.4 画相应分析图
corresp对象的相应分析图可以用biplot()或plot()画出,ca对象的相应分析图只能用plot()画出,两个函数都是直接将相应分析结果放入第一个参数即可画出,不多做介绍
续上例:
> par(mfrow=c(1,2))
> biplot(CA1)
> plot(CA2)
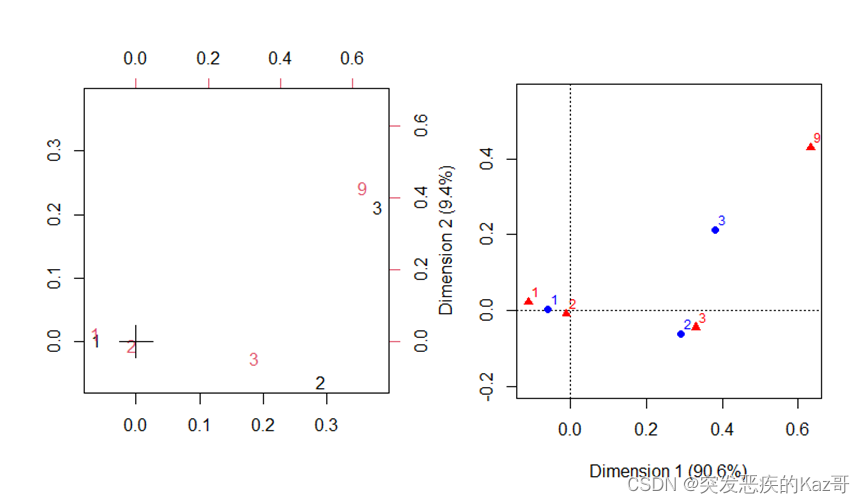
左图是corresp()函数对象的相应分析图
右图是ca()函数对象的相应分析图
红色的数字是幸福程度的水平,黑色或蓝色的数字表示不同的人种,两个相应分析图有些许不同,但是都相似1号人种的幸福程度与1、2很近;2号人种的幸福程度与3比较接近。左图的3号人种与幸福程度与9很接近,而右图的3号人种与幸福程度3和9的距离差不多,而且距离相对比较远,3号人种的幸福程度既较多是3也较多是9
四、小结
对各项分析所用到的函数和应用场景进行总结
| 主成分分析 | 因子分析 | ||
| 函数 | 应用场景 | 函数 | 应用场景 |
| kmo() | 开源获取,kmo检验 | kmo() | 开源获取,kmo检验 |
| cortest.bartlett() | 巴特利特球形检验,与kmo检验搭配检查信息重叠度 | cortest.bartlett() | 巴特利特球形检验,与kmo检验搭配检查信息重叠度 |
| princomp() | 主成分分析 | fa.parallel() | 画碎石图,确定因子个数 |
| principal() | psych包内的主成分分析 | fa() | 因子分析 |
| fa.parallel() | 画碎石图,提前确定主成分个数,对于principal对象 | ||
| screeplot() | 画碎石图,确定主成分个数,对于princomp对象 | ||
| evaluation() | 自编,综合评价 | ||
| 相应分析 | |
| 函数 | 应用场景 |
| table() | 做列联表 |
| chisq.test() | 独立性皮尔逊卡方检验 |
| likelihood.test() | 独立性似然比卡方检验 |
| corresp() | 简单相应分析(MASS) |
| ca() | 简单相应分析(ca) |
| plot() | 画相应分析图 |
| biplot() | 画相应分析图,对于corresp对象 |