有了泛癌的数据之后就可以进行各种分析了,当然这些都是在R语言的基础上进行的。如果你不会R语言,也可以通过各种各样的网页工具实现。
我们今天就简单展示下任意基因在泛癌图谱中的表达量情况。
TCGA,GTEx,TCGA+GTEx的泛癌数据都整理好了,大家可以自己通过easyTCGA包实现1行代码整理,也可以直接在公众号后台回复pancancer获取整理好的数据。详情请见:任意基因在泛癌中的表达量展示
GTEx
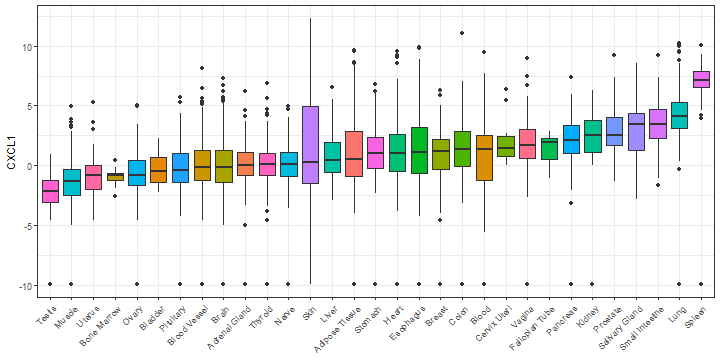
GTEx的展示比较简单,最常见的就是某个基因在所有组织中的表达量情况。
# 加载数据
load(file="output_pancancer_xena/GTEx_pancancer_mrna_pheno.rdata")
library(tidyverse)
## ── Attaching packages ─────────────────────────────────────── tidyverse 1.3.2 ──
## ✔ ggplot2 3.4.1 ✔ purrr 1.0.1
## ✔ tibble 3.2.1 ✔ dplyr 1.1.1
## ✔ tidyr 1.3.0 ✔ stringr 1.5.0
## ✔ readr 2.1.4 ✔ forcats 1.0.0
## ── Conflicts ────────────────────────────────────────── tidyverse_conflicts() ──
## ✖ dplyr::filter() masks stats::filter()
## ✖ dplyr::lag() masks stats::lag()# 简单看下,这几个泛癌数据的详细情况我都给大家有说明,看一下即可
head(colnames(gtex_mrna_pheno))
## [1] "sample_id" "primary_site" "MT-ATP8" "MT-ATP6" "MT-CO2"
## [6] "MT-CO3"
#table(gtex_mrna_pheno$primary_site)
length(table(gtex_mrna_pheno$primary_site))
## [1] 31
接下来以CXCL1这个基因为例进行展示。
gene <- "CXCL1"# 提取数据就是这么简单
plot_df <- gtex_mrna_pheno %>%select(1:2,all_of(gene))# 画图即可
ggplot2::ggplot(plot_df, aes(fct_reorder(primary_site,CXCL1),CXCL1))+ggplot2::geom_boxplot(aes(fill=primary_site))+ggplot2::labs(x=NULL)+ggplot2::theme_bw()+ggplot2::theme(legend.position = "none",axis.text.x = ggplot2::element_text(angle = 45,hjust = 1))
TCGA
单独使用TCGA泛癌的数据进行展示是花样最多的,你在pubmed中以pan cancer为关键词进行检索,基本上其中的Fig1都是类似的箱线图。
# tcga pancancer,前34列是临床信息
rm(list = ls())
load(file="output_pancancer_xena/TCGA_pancancer_mrna_clin.rdata")head(colnames(tcga_mrna_clin))
## [1] "sample_id" "patient_id"
## [3] "project" "age_at_initial_pathologic_diagnosis"
## [5] "gender" "race"
继续以CXCL1这个基因为例进行展示。
gene <- "CXCL1"plot_df <- tcga_mrna_clin %>%select(sample_id,project,all_of(gene)) %>%# 这个分组你可以任意指定,并不一定要tumor、normalmutate(sample_type=ifelse(as.numeric(substr(.$sample_id,14,15))<10,"tumor","normal"))#tcga pancancer中有很多癌种没有normal哦,要注意!
ggplot2::ggplot(plot_df,aes(project,CXCL1))+ggplot2::geom_boxplot(aes(fill=sample_type))+ggplot2::labs(x=NULL,y="expression")+ggplot2::theme_bw()+ggplot2::theme(legend.position = "top",axis.text.x = ggplot2::element_text(angle = 45,hjust = 1))+ggpubr::stat_compare_means(ggplot2::aes(group = sample_type,label = "p.format"),method = "kruskal.test")
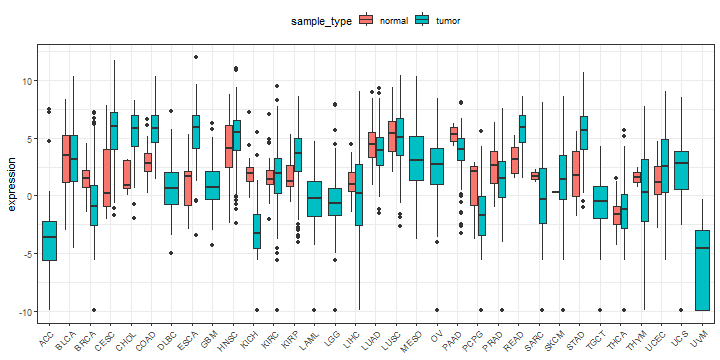
或者你也可以展示只在tumor样本中的表达量。
plot_df <- plot_df %>%filter(sample_type=="tumor")ggplot2::ggplot(plot_df,aes(fct_reorder(project,CXCL1),CXCL1))+ggplot2::geom_boxplot(aes(fill=project))+ggplot2::labs(x=NULL,y="expression")+ggplot2::theme_bw()+ggplot2::theme(legend.position = "none",axis.text.x = ggplot2::element_text(angle = 45,hjust = 1))
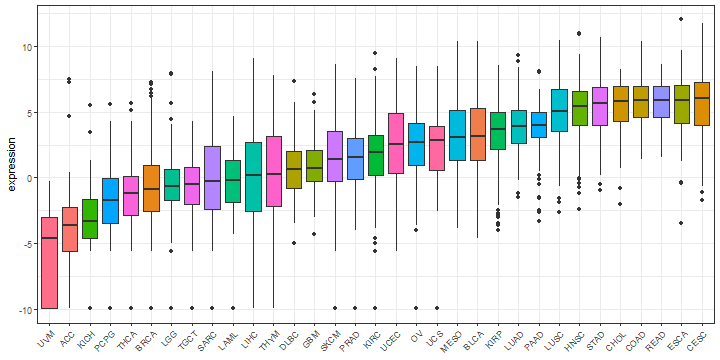
接下来是大家比较感兴趣的某个基因在泛癌配对样本中的表达。
首先我们把泛癌的表达矩阵(这里应该叫转置后的表达矩阵比较合适,一般我我们说表达矩阵就是指行是基因,列是样本的矩阵)按照project拆分,然后自定义一个可以提取配对样本的函数:
# 拆分
cancer_list <- split(tcga_mrna_clin,tcga_mrna_clin$project)# 自定义函数
get_paired_sample <- function(exprset){# get paired samplessample_group <- ifelse(as.numeric(substr(exprset$sample_id,14,15))<10,"tumor","normal")tmp <- data.frame(sample_group = sample_group, sample_id=exprset$sample_id,project=exprset$project)tmp_nor <- tmp[tmp$sample_group=="normal",]tmp_tum <- tmp[tmp$sample_group=="tumor",]#每一个normal都有配对的tumor吗?并不是keep <- intersect(substr(tmp_tum$sample_id,1,12),substr(tmp_nor$sample_id,1,12))tmp_tum <- tmp_tum[substr(tmp_tum$sample_id,1,12) %in% keep,]tmp_tum <- tmp_tum[!duplicated(substr(tmp_tum$sample_id,1,12)),]tmp_nor <- tmp_nor[substr(tmp_nor$sample_id,1,12) %in% keep,]tmp_nor <- tmp_nor[!duplicated(substr(tmp_nor$sample_id,1,12)),]tmp_pair <- rbind(tmp_tum,tmp_nor)
}
接下来就是把这个函数应用于33种癌症中,然后提取CXCL1这个基因的画图数据即可:
paired_samples <- do.call(rbind,lapply(cancer_list,get_paired_sample))plot_df <- paired_samples %>%left_join(tcga_mrna_clin[,c(gene,"sample_id")]) %>%mutate(sample_id=substr(sample_id,1,12))
## Joining with `by = join_by(sample_id)`
接下来画图就是基本功了,ggplot2搞定一切,下面这个interaction的用法在《R数据可视化手册》中有讲过,我强烈呼吁大家赶紧买本书看看吧!别再天天问图怎么画了!
ggplot(plot_df, aes(interaction(sample_group,project),CXCL1,color=sample_group))+ggplot2::geom_point(size=3,position = position_dodge(0.9))+ggplot2::geom_line(aes(group=interaction(sample_id,project)),color="grey70")+ggplot2::scale_color_manual(values = c("#028EA1","#F2AA9D"))+ggplot2::scale_x_discrete(labels = rep(unique(plot_df$project),each=2))+ggplot2::theme_bw()+ggplot2::theme(legend.position = "top",axis.text.x = element_text(angle = 45,hjust = 1))
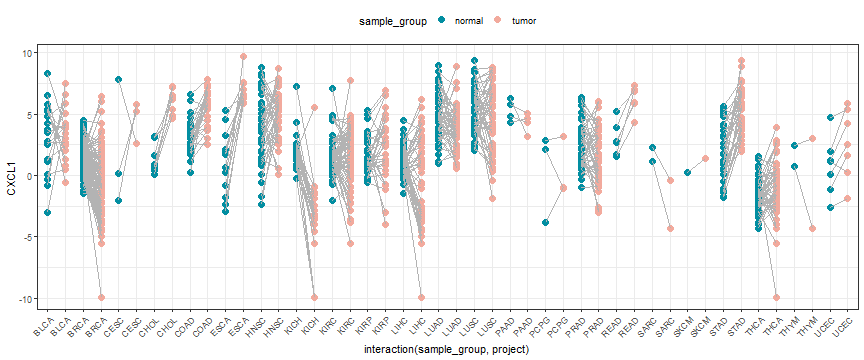
当然还有分面的画法:
#下面是分面
ggplot(plot_df, aes(interaction(sample_group,project),CXCL1,color=sample_group))+ggplot2::geom_point(size=3)+ggplot2::geom_line(aes(group=interaction(sample_id,project)),color="grey70")+ggplot2::scale_color_manual(values = c("#028EA1","#F2AA9D"))+ggplot2::scale_x_discrete(name = NULL)+ggplot2::facet_grid(~project,scales="free_x",switch = "x")+ggplot2::theme_bw()+ggplot2::theme(legend.position = "top",axis.text.x = element_blank(),strip.background = element_blank(),axis.ticks.x = element_blank(),panel.border = element_blank())
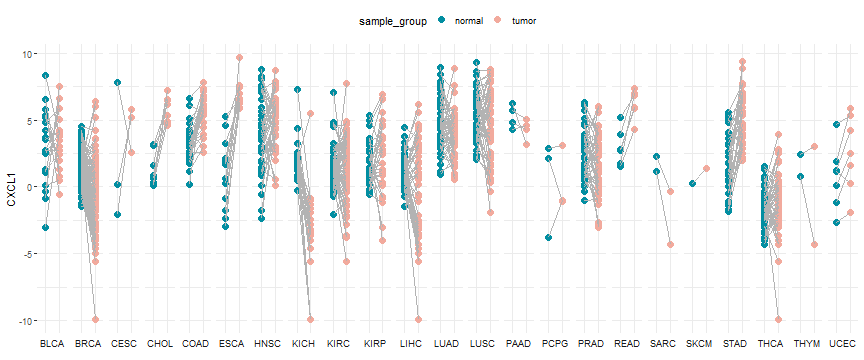
当然如果你看了书也搞不明白,也可以通过万能的网络解决一切,比如上面这种图,你可以通过关键词搜索:ggplot2 paired line multi groups, 实现方式非常多,任你选择。
TCGA+GTEx
TCGA+GTEx就没有配对展示了,除此之外都和TCGA的泛癌展示方式差不多。
rm(list = ls())
load(file="output_pancancer_xena/TCGA_GTEx_pancancer_mRNA_pheno.rdata")# 前4列是样本信息
head(colnames(tcga_gtex_mrna_pheno))
## [1] "sample_id" "sample_type" "project" "primary_site" "MT-ATP6"
## [6] "MT-CO2"
table(tcga_gtex_mrna_pheno$sample_type)
##
## GTEx_normal TCGA_normal TCGA_tumor
## 7568 712 9784
table(tcga_gtex_mrna_pheno$project)
##
## ACC BLCA BRCA CESC CHOL COAD DLBC ESCA GBM HNSC KICH KIRC KIRP LAML LGG LIHC
## 205 435 1390 319 45 637 491 848 1317 564 119 631 349 243 1674 531
## LUAD LUSC MESO OV PAAD PCPG PRAD READ SARC SKCM STAD TGCT THCA THYM UCEC UCS
## 862 836 87 515 350 185 648 410 264 1282 624 302 850 565 272 135
## UVM
## 79
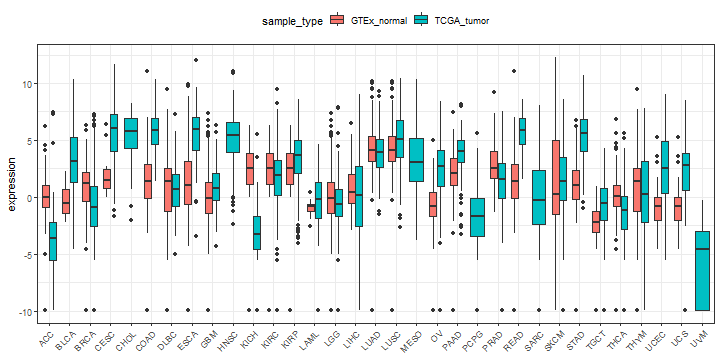
继续以CXCL1这个基因为例进行展示。
用的最多的肯定还是任意基因在不同组别中的表达:
gene <- "CXCL1"plot_df <- tcga_gtex_mrna_pheno %>%select(1:4,all_of(gene)) %>%filter(sample_type %in% c("GTEx_normal","TCGA_tumor"))ggplot(plot_df,aes(project,CXCL1))+geom_boxplot(aes(fill=sample_type))+theme(legend.position = "top")+ggplot2::labs(x=NULL,y="expression")+ggplot2::theme_bw()+ggplot2::theme(legend.position = "top",axis.text.x = ggplot2::element_text(angle = 45,hjust = 1))
扩展
其实任何类似于这个数据的格式都能像这样展示。
比如你可以通过ssGSEA对泛癌进行免疫浸润分析,这样每个样本都可以有一个得分,这样你就可以展示某个细胞在不同组别中的得分情况。
大家一定要多看文献,多积累不同的方法,以及一些好用的网站、图表等,说不定以后就用到了!
后面可能会安排几篇图表复现的推文,敬请期待。