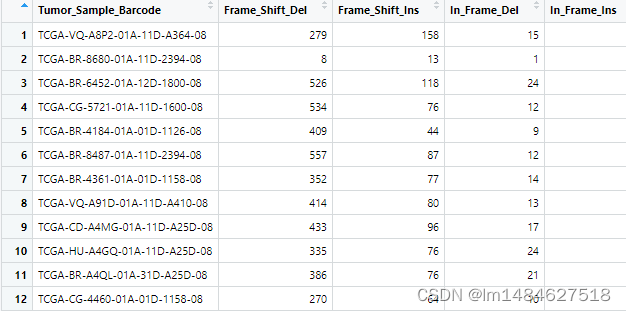
基因组变异检测SNPcalling(GATK)
- 第一步,将数据软连接到自己的工作路径下
- 第二步,用BWA index功能为ref文件做index,用Samtools软件为ref做index
- 第三步,用BWA软件做序列比对,得到序列比对的sam文件
- 第四步,sam文件转化成bam文件,并对bam文件进行进一步过滤(排序+去冗余)
- (optional)检查一下生成的bam文件质量是否合格
- 第五步,建立dict文件
- 第六步,得到变异文件
- 用samtools tview看这两个变异位点
- 第七步,筛选vcf文件,得到高质量的SNV
软件路径:
/opt/software/bwa-0.7.17/bwa
/opt/software/samtools-1.11/samtools
/opt/software/gatk-4.0.12.0/gatk
/opt/software/vcftools_0.1.13/bin/vcftools
数据路径:
/opt/data/E.coli_K12_MG1655.fa
/opt/data/SRR1770413_1.fastq
/opt/data/SRR1770413_2.fastq
工作路径:
##每一个登录账号下创建自己的工作目录
mkdir -p yourname/SNPCalling
cd /home/yourname/SNPCalling (路径需要修改)
第一步,将数据软连接到自己的工作路径下
ln -s /opt/data/E.coli_K12_MG1655.fa ./
ln -s /opt/data/SRR1770413_1.fastq ./
ln -s /opt/data/SRR1770413_2.fastq ./
第二步,用BWA index功能为ref文件做index,用Samtools软件为ref做index
/opt/software/bwa-0.7.17/bwa index E.coli_K12_MG1655.fa
/opt/software/samtools-1.11/samtools faidx E.coli_K12_MG1655.fa
第三步,用BWA软件做序列比对,得到序列比对的sam文件
/opt/software/bwa-0.7.17/bwa mem -M -t 1 -R '@RG\tID:Ecoli\tSM:Ecoli\tLB:Ecoli\tPL:ILLUMINA\tPU:run' E.coli_K12_MG1655.fa SRR1770413_1.fastq SRR1770413_2.fastq > SRR1770413.sam
第四步,sam文件转化成bam文件,并对bam文件进行进一步过滤(排序+去冗余)
java -Xmx8G -jar /opt/software/picard-tools/picard.jar SortSam I=SRR1770413.sam O=SRR1770413.bam SO=coordinate VALIDATION_STRINGENCY=LENIENT CREATE_INDEX=true TMP_DIR=./tmp.363296
java -Xmx8G -jar /opt/software/picard-tools/picard.jar MarkDuplicates I=SRR1770413.bam O=SRR1770413.dup.bam M=SRR1770413.dup.txt REMOVE_DUPLICATES=true VALIDATION_STRINGENCY=LENIENT CREATE_INDEX=true TMP_DIR=./tmp.690131
(optional)检查一下生成的bam文件质量是否合格
/opt/software/samtools-1.11/samtools flagstat SRR1770413.dup.bam > SRR1770413.dup.bam.stat
第五步,建立dict文件
java -jar /opt/software/picard-tools/CreateSequenceDictionary.jar R= E.coli_K12_MG1655.fa O= E.coli_K12_MG1655.dict
第六步,得到变异文件
/opt/software/gatk-4.0.12.0/gatk HaplotypeCaller -R E.coli_K12_MG1655.fa -I SRR1770413.dup.bam -O SRR1770413.dup.vcf
用samtools tview看这两个变异位点
/opt/software/samtools-1.11/samtools tview SRR1770413.dup.bam E.coli_K12_MG1655.fa
##可以按g跳转至NC_000913.3: 378700查看,或NC_000913.3:566530
第七步,筛选vcf文件,得到高质量的SNV
(筛选至少50%的个体成功被snp calling的位点,最低质量分数为30的位点,次要等位基因计数为3的位点,–recode标志告诉程序使用过滤器写入一个新的vcf文件,–recode-INFO-all保留旧vcf文件中的所有INFO标志。–out 输出的文件名称)
/opt/software/vcftools_0.1.13/bin/vcftools --vcf SRR1770413.dup.vcf --max-missing 0.5 --minDP 3 --minQ 30 --recode --recode-INFO-all --out filt
最终得到filt.recode.vcf这个文件。