推荐阅读:
造血干细胞扩增、转染以及基因编辑优化解决方案
T细胞培养技术进展及解决方案
PCR技术自从1985年由Mullis发明以来,被广泛的应用到核酸分子的检测当中。但显然,只能定性分析而无法定量,成为困扰科研工作者的一大难题。仅仅在5年之后,就有了一些初步的尝试,并取得了不错的效果。
Pang等人在采用了一种PCR后分子显像的方法来进行定量[1],将PCR的原料ATP用32P标记,掺入到产物中去,用自显影技术进行分析(Fig 1),其精度足以区分两倍浓度的差异。这种方法操作相对繁琐,且涉及放射性标记;另外,其检测方式仍稍显粗糙,仅考量条带的光密度值,属于半定量的范畴,同时还需要将扩增循环数控制在指数期(30个之内)。该方法尽管未得到广泛应用,但却迈出了突破性的一步。
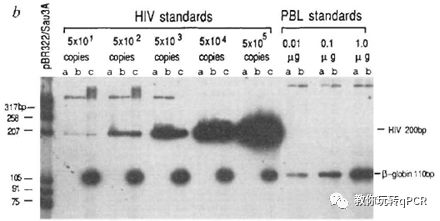
Fig 1. 基因组DNA及HIV-1 DNA的自显影检测
应用更广泛的一种定量方法是在1991年出现的,叫做Quantitative Competitive PCR(QC-PCR)[2]。其原理是样本中加入一种已知浓度的内参模板,即competitor,该competitor与样本中的target有着一致的扩增效率,这样扩增结束后(不必要求在指数期)C与T的比值就与扩增前是一致的,而扩增后的C:T可以通过电泳分析得到数值,从而推出扩增前target的数量(Fig 2)。该方法的精确度与灵敏度都得到了较大提升,应用于HIV-1的检测中,其灵敏度可达到100 copy per reaction[3]。但其缺点也是显而易见的,除了操作稍显繁琐,如何设计competitor是最大的难点。
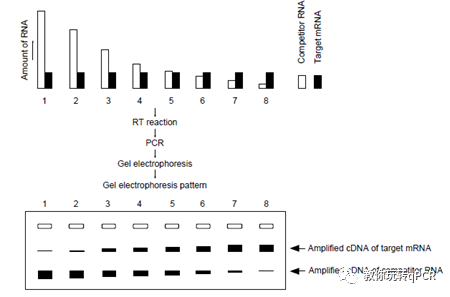
Fig 2. QC-PCR的原理示意图(Takara Competitive PCR Guide)
虽然前面的两种方法都不尽如人意,但为qPCR的诞生提供了正确的解决思路——如何在PCR产物浓度与初始模板浓度之间建立关系。1996年,Real Time Quantitative PCR的发表标志着qPCR的诞生[4]。它引入了荧光标记的水解探针,并在每个PCR循环的延伸结束后采集荧光信号,将到达指数扩增期所需的循环数(Ct值,后经标准化统一为Cq值)与模板的初始浓度建立了关系(Fig 3),这样就可以定量未知样品中target的数量(原理我会在下一节详细介绍)。

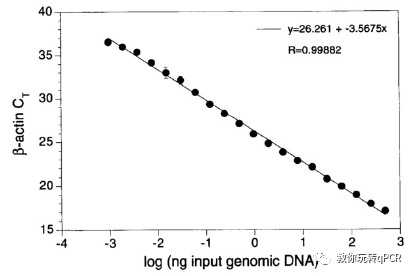
Fig 3. 荧光信号的实时检测和标准曲线的建立
该文章对此方法的精密度进行了详细的考察,表明对于不同的样本处理方法仍然是高度可重复的;文中还提到,相较于前两种方法,qPCR有三大优势:1、在密闭系统中进行检测,而无需PCR后开盖跑电泳等,减少了产物污染的可能性;2、支持管家基因作为calibrator,无需添加internal control,同时多重检测成为可能;3、检测时间短,样本通量高。该文章至今已被引用超7000次(GoogleScholar),其开创性和经典性毋庸置疑。
自这篇文章之后,各种技术革新型的文章如雨后春笋般冒出来。Philip S等创造性的使用探针技术做溶解曲线(Fig4),区分血色病基因的不同变体[5];Jacqueline等使用qPCR技术同时检测四种逆转录病毒核酸[6],多重检测日趋成熟。Yolanda等综述了qPCR在拷贝数决定、RNA表达和等位基因区分等方面的应用[7]。
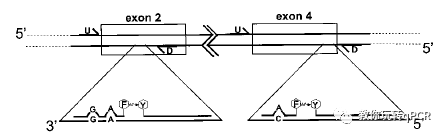
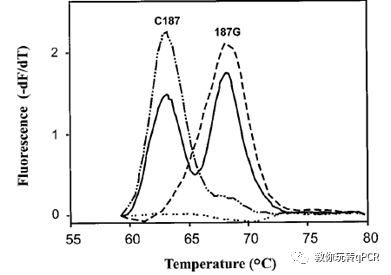
Fig 4. 荧光共振能量转移(FRET)技术用于溶解曲线分析
Michael等描述了将qPCR与抗原抗体结合相联系来检测微量的蛋白质的方法[8],将传统的Biotin-Streptavidin生物系统用PCR的方式替代(Fig 5),信号放大的作用更有优势,将蛋白质检测的灵敏度相比传统的ELISA提高了两到三个数量级。该文章虽然不是首创,但使得该方法更加系统和完善。Livak等于2001年提出了用我们熟悉的2-△△Cq来处理基因表达的数据[9],整个流程更加精炼简化;后来Pfaffl和Vandesompele基于不同的实验条件分别完善了计算方法[10][11],应用条件更加趋于真实情况。
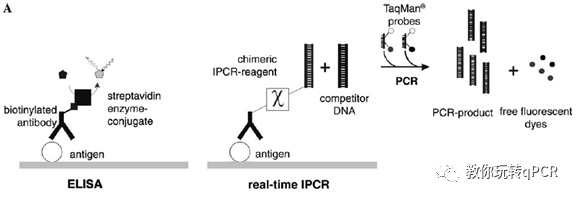
Fig 5. Real-time Immuno PCR与传统ELISA的原理上区别
直到最近,仍然有更多的以qPCR基础创新出的核酸定量技术发表。Yanan Du等描述了一种乳化PCR结合光谱检测的方式实现的核酸多重检测(EPFS System, see Fig 6)[12],对其灵敏度、特异性和重复性进行了详细的评估,并在转基因玉米中进行了测试。
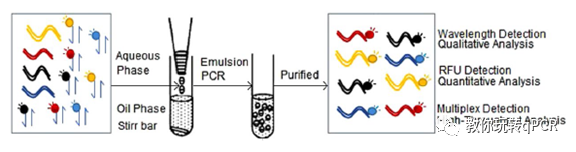
Fig 6. 荧光分子光谱的乳化PCR技术(EPFS)示意图
qPCR问世至今已经23年,对于科技日新月异的今天而言,已经算一门“老技术活”了。但是,我们仍然能够从这些老技术活中汲取营养,深耕细作或者另辟蹊径;老树开新花,说的就是这个意思了。未来,DNA荧光分子检测、微流控技术甚至流式细胞术的结合,必定会使qPCR绽放别样的风采。
参考文献
1. Pang S,Koyanagi Y, Miles S, et al. High levels of unintegrated HIV-1 DNA in braintissue of AIDS dementia patients[J]. Nature, 1990, 343(6253): 85.
2.Becker-Andre, M. Quantitative evaluation of mRNA levels. Method Molecular Cell Biology. 1991. 2:189-201
3. Piatak,Michael, et al. "High levels of HIV-1 in plasma during all stages ofinfection determined by competitive PCR." Science 259.5102 (1993):1749-1754.
4. Heid C A,Stevens J, Livak K J, et al. Real time quantitative PCR[J]. Genome research,1996, 6(10): 986-994.
5. Bernard,Philip S., et al. "Homogeneous multiplex genotyping of hemochromatosismutations with fluorescent hybridization probes." The American journal ofpathology 153.4 (1998): 1055-1061.
6. Vet,Jacqueline AM, et al. "Multiplex detection of four pathogenic retrovirusesusing molecular beacons." Proceedings of the National Academy of Sciences96.11 (1999): 6394-6399.
7. Lie Y S,Petropoulos C J. Advances in quantitative PCR technology: 5′nuclease assays[J]. Current Opinion in Biotechnology, 1998, 9(1): 43-48.
8. Adler M,Wacker R, Niemeyer C M. A real-time immuno-PCR assay for routine ultrasensitivequantification of proteins[J]. Biochemical and biophysical researchcommunications, 2003, 308(2): 240-250.
9. Livak K J,Schmittgen T D. Analysis of relative gene expression data using real-timequantitative PCR and the 2− ΔΔCT method[J]. methods, 2001, 25(4): 402-408.
10.Pfaffl, Michael W. "A new mathematicalmodel for relative quantification in real-time RT–PCR." Nucleic acidsresearch 29.9 (2001): e45-e45.
11. VandesompeleJ, De Preter K, Pattyn F, et al. Accurate normalization of real-timequantitative RT-PCR data by geometric averaging of multiple internal controlgenes[J]. Genome biology, 2002, 3(7): research0034. 1.
12. Du Y, ZhaoX, Zhao B, et al. A novel emulsion PCR method coupled with fluorescencespectrophotometry for simultaneous qualitative, quantitative andhigh-throughput detection of multiple DNA targets[J]. Scientific reports, 2019,9(1): 184