摘要
U-Net是基于卷积神经网络(CNN)体系结构设计而成的,由Olaf Ronneberger,Phillip Fischer和Thomas Brox于2015年首次提出应用于计算机视觉领域完成语义分割任务。(其发表U-Net的作者建议该模型能够很好的应用于生物医学图像分割)。而本文将从U-Net的应用领域、模型原理、具体应用三个方面具体概述U-Net神经网络的实现方式及应用技术。最后,本文基于keras神经网络框架设计并实现U-Net模型在EM神经元数据集上的 分割应用,其实现结果现实,U-Net在EM 堆栈神经元的分割任务上具有较好的实验结果。
1.计算机视觉任务概述
1.1 语义分割解析(Semantic Segmentation)
1.1.1 定义
图像语义分割(Semantic Segmentation)是图像处理和是机器视觉技术中关于图像理解的重要一环,也是 AI 领域中一个重要的分支。语义分割即是对图像中每一个像素点进行分类,确定每个点的类别(如属于背景、人或车等),从而进行区域划分。目前,语义分割已经被广泛应用于自动驾驶、无人机落点判定等场景中。
1.1.2 实例解释
图像分类
举个例子进行描述:
在图像领域,我们常见的任务是对图像的分类任务,该任务是较为粗粒度的来识别和理解图像。即是我们给定一张图像,我们期望模型输出该张图像所属的类别(离散标签),而在图像分类任务中,我们假设图像中只有一个(而不是多个)对象。描述如下图:

但是,语义分割则在图像分类任务上更为细化。其需要经历三个过程:图像定位、目标检测、语义分割(及实例分割)。其中,目标检测( Object detection)不仅需要提供图像中物体的类别,还需要提供物体的位置(bounding box)。语义分割( Semantic segmentation)需要预测出输入图像的每一个像素点属于哪一类的标签。实例分割( instance segmentation)在语义分割的基础上,还需要区分出同一类不同的个体。
图像定位
在与离散标签一起输出时(完成分类任务过程中),我们还期望模型能够准确定位图像中存在该物体的位置。这种定位通常使用边界框来实现,边界框可以通过一些关于图像边界的数值参数来识别。其如下图所示:

目标检测
与图像定位不同的是,目标检测需要判断图像中物体的类别,还判断指出提供图像中物体的具体位置(bounding box)。即是现在图像不再局限于只有一个对象,而是可以包含多个对象。任务是对图像中的所有对象进行分类和定位。这里再次使用边界框的概念进行定位。其如下图所示:

语义分割
语义分割通过对每个像素进行密集的预测、推断标签来实现细粒度的推理,从而使每个像素都被标记为其封闭对象矿石区域的类别。即是需要判断图像每个像素点的类别,进行精确分割。图像语义分割是像素级别的!因为我们要对图像中的每个像素进行预测,所以这个任务通常被称为密集预测。其如下图所示:

实例分割
实例分割比语义分割是更为细致的下游任务,其中与像素级的分类任务处理方式一样,我们希望模型分别对类的每个实例进行分类。例如,图中有 3 个人,严格来说是“Person”类的 3 个实例。所有这3个分别分类(以不同的颜色)。其如下图所示:

目标检测、语义分割、实例分割三个像素级别的分类任务,其区别如下图所示。

1.2 应用领域
1.2.1 自动驾驶汽车
自动驾驶是一项极为复杂的深度学习应用任务,其需要模型在不断变化的环境中进行感知、规划和执行相关任务(既是汽车避免路障等问题)。因为人生安全是最重要的,所以这项任务需要最为精准的模型运行效果。语义分割可提供有关道路上的障碍信息、检测路况标记和交通标志等信息。

1.2.2 生物医学影像诊断(精准医疗)
这就是本文将要实现的具体应用了。其实现的最终模型可以减少放射科医生的分析时间,为相关疾病的诊疗提供辅助和技术支持。

1.2.3 地理传感
语义分割问题也可以被视为分类问题(其最终的解决方式),其中每个像素被归类为一系列对象类别中的一个。因此,可用于卫星图像的土地使用制图。例如监测森林砍伐和城市化区域。道路和建筑物检测也是交通管理、城市规划和道路监测的重要研究课题等。

1.2.4 精准农业
农业机器人可以减少田间需要喷洒的除草剂次数和剂量,通过对作物和杂草进行语义分割,实时协助它们触发除草动作。

2. U-Net模型解释
2.1 U-Net模型概述
U-Net的U形结构如图所示。网络是一个经典的全卷积网络(即网络中没有全连接操作)。网络的输入是一张 572*572 的边缘经过镜像操作的图片(input image tile),关于“镜像操作“会在接下来进行详细分析,网络的左侧(红色虚线)是由卷积和Max Pooling构成的一系列降采样操作,论文中将这一部分叫做压缩路径(contracting path)。压缩路径由4个block组成,每个block使用了3个有效卷积和1个Max Pooling降采样,每次降采样之后Feature Map的个数乘2,因此有了图中所示的Feature Map尺寸变化。最终得到了尺寸为 32*32的Feature Map。
网络的右侧部分(绿色虚线)在论文中叫做扩展路径(expansive path)。同样由4个block组成,每个block开始之前通过反卷积将Feature Map的尺寸乘2,同时将其个数减半(最后一层略有不同),然后和左侧对称的压缩路径的Feature Map合并,由于左侧压缩路径和右侧扩展路径的Feature Map的尺寸不一样,U-Net是通过将压缩路径的Feature Map裁剪到和扩展路径相同尺寸的Feature Map进行归一化的(即图1中左侧虚线部分)。扩展路径的卷积操作依旧使用的是有效卷积操作,最终得到的Feature Map的尺寸是 388*388。由于该任务是一个二分类任务,所以网络有两个输出Feature Map。
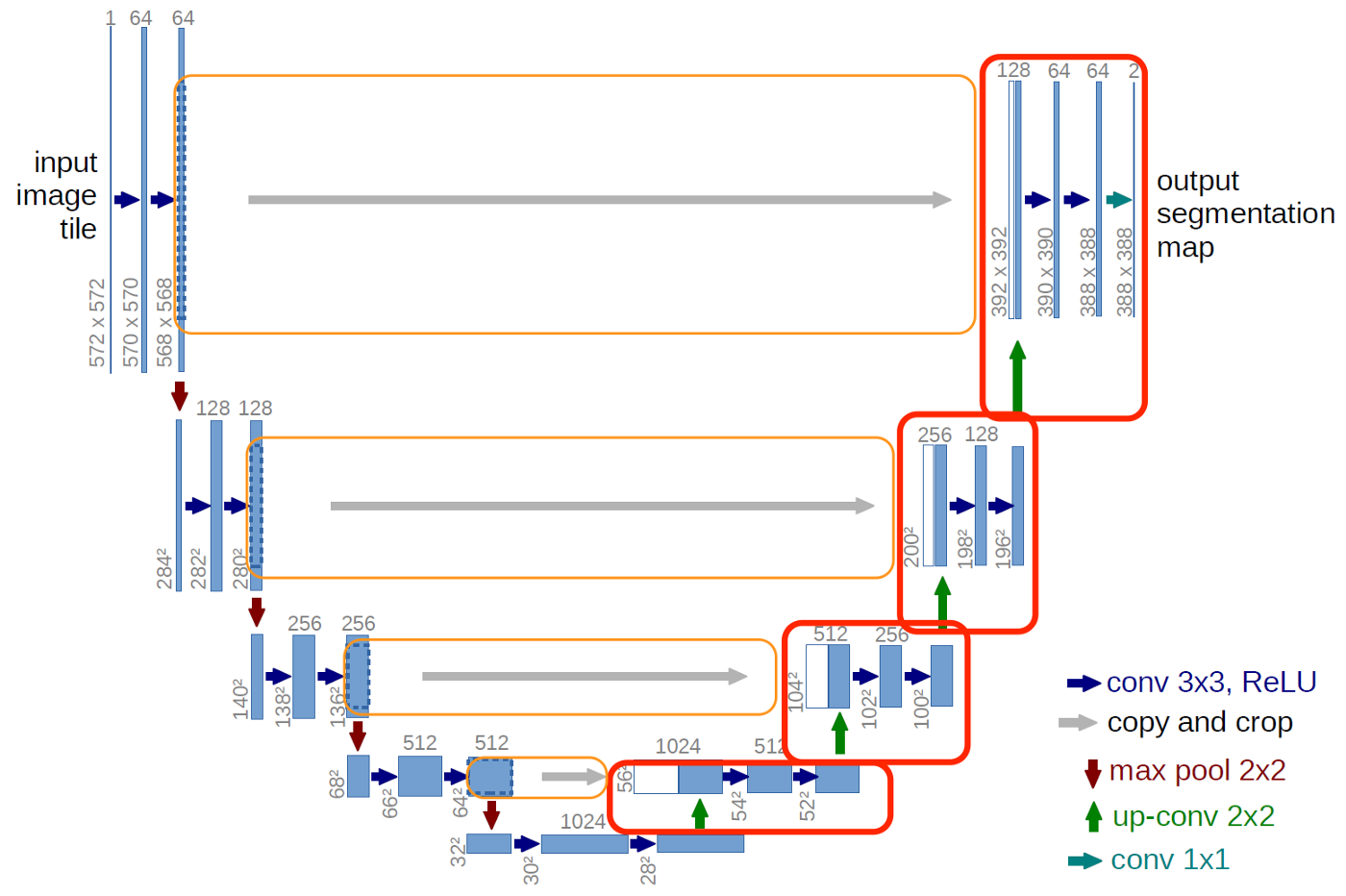
首先,数据集我们的原始图像的尺寸都是 512*512 的。为了能更好的处理图像的边界像素,U-Net使用了镜像操作(Overlay-tile Strategy)来解决该问题。镜像操作即是给输入图像加入一个对称的边(图2),那么边的宽度是多少呢?一个比较好的策略是通过感受野确定。因为有效卷积是会降低Feature Map分辨率的,但是我们希望 515*512的图像的边界点能够保留到最后一层Feature Map。所以我们需要通过加边的操作增加图像的分辨率,增加的尺寸即是感受野的大小,也就是说每条边界增加感受野的一半作为镜像边。
2.2 U-Net 和 autoencoder 架构的区别
U-Net模型与传统的图像分割方法(自动编码器体系结构)有所区别。传统的经典图像分割方法是最初输入信息的大小随着层数的增加其特征信息在不断减少,至此,自动编码器体系结构的编码器部分完成,开始解码器部分。而U-Net模型学习线性特征表示,其特征大小在逐渐增大,其最后的输出大小等于输入大小。
既是整个Auto-Encoder有两个吸引人的应用:1)利用前半部分做特征提取;2)利用后半部分做图像生成。

然而,U-Net结构和Auto-Encoder的传统结构十分相似,但是它独特的前传结构让网络能够capture到很多空间的信息,U-Net的直接前传可以保留很多空间信息,既其前半部分是降采样,下半部分是升采样。
2.3 U-Net模型代码详解
在这部分代码实现过程中一定要注意调整卷积核的大小和padding的大小,这样才可以在最后保证图片的尺寸恢复到和原来一样。
inputs = Input(input_size)conv1 = Conv2D(64, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(inputs)conv1 = Conv2D(64, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(conv1)pool1 = MaxPooling2D(pool_size=(2, 2))(conv1)conv2 = Conv2D(128, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(pool1)conv2 = Conv2D(128, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(conv2)pool2 = MaxPooling2D(pool_size=(2, 2))(conv2)conv3 = Conv2D(256, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(pool2)conv3 = Conv2D(256, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(conv3)pool3 = MaxPooling2D(pool_size=(2, 2))(conv3)conv4 = Conv2D(512, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(pool3)conv4 = Conv2D(512, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(conv4)drop4 = Dropout(0.5)(conv4)pool4 = MaxPooling2D(pool_size=(2, 2))(drop4)conv5 = Conv2D(1024, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(pool4)conv5 = Conv2D(1024, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(conv5)drop5 = Dropout(0.5)(conv5)up6 = Conv2D(512, 2, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(UpSampling2D(size = (2,2))(drop5))merge6 = concatenate([drop4,up6], axis = 3)conv6 = Conv2D(512, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(merge6)conv6 = Conv2D(512, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(conv6)up7 = Conv2D(256, 2, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(UpSampling2D(size = (2,2))(conv6))merge7 = concatenate([conv3,up7], axis = 3)conv7 = Conv2D(256, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(merge7)conv7 = Conv2D(256, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(conv7)up8 = Conv2D(128, 2, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(UpSampling2D(size = (2,2))(conv7))merge8 = concatenate([conv2,up8], axis = 3)conv8 = Conv2D(128, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(merge8)conv8 = Conv2D(128, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(conv8)up9 = Conv2D(64, 2, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(UpSampling2D(size = (2,2))(conv8))merge9 = concatenate([conv1,up9], axis = 3)conv9 = Conv2D(64, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(merge9)conv9 = Conv2D(64, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(conv9)conv9 = Conv2D(2, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(conv9)conv10 = Conv2D(1, 1, activation = 'sigmoid')(conv9)model = Model(input = inputs, output = conv10)from keras.utils.vis_utils import plot_modelplot_model(model, to_file='model1.png',show_shapes=True)3. 应用实现
3.1 数据集介绍
数据来自于 ISBI 挑战的数据集。该数据集包含30张训练图、30张对应的标签。30张测试图片。数据是来自果蝇一龄幼虫腹神经索 (VNC) 的串行部分透射电子显微镜 (ssTEM)的图像,其分辨率为4x4x50 nm /像素。
3.2 模型设计与实现
实验环境:python3.6.5;tensorflow==1.12;keras==2.2.4等;
上述2.3实现的 U-Net模型其参数过多,在自己电脑上运行过慢,为了快速建立模型并实现应用,我们设计了更小是U-Net模型,其实现后的结构如下所示:

其模型的训练采用如下代码进行实现(添加了数据增强):
ata_gen_args = dict(rotation_range=0.2,width_shift_range=0.05,height_shift_range=0.05,shear_range=0.05,zoom_range=0.05,horizontal_flip=True,fill_mode='nearest')
myGene = trainGenerator(2,'data/membrane/train','image','label',data_gen_args,save_to_dir = None)model = unet()
model_checkpoint = ModelCheckpoint('unet_membrane.hdf5', monitor='loss',verbose=1, save_best_only=True)
model.fit_generator(myGene,steps_per_epoch=3,epochs=5,callbacks=[model_checkpoint],validation_data=())
其实验结果的准确率曲线与模型训练次数的曲线如下所示:


从图中我们发现 U-Net模型在经过简化之后,其在细胞分割任务中已经达到了不错的训练效果结果,其准确率高达78%,为了进一步提高模型准确率,我们需要在模型设计环节设计完整的 U-Net模型,并加大模型的训练次数,这样就能使模型很轻松的达到95%左右。
3.3 结果展示
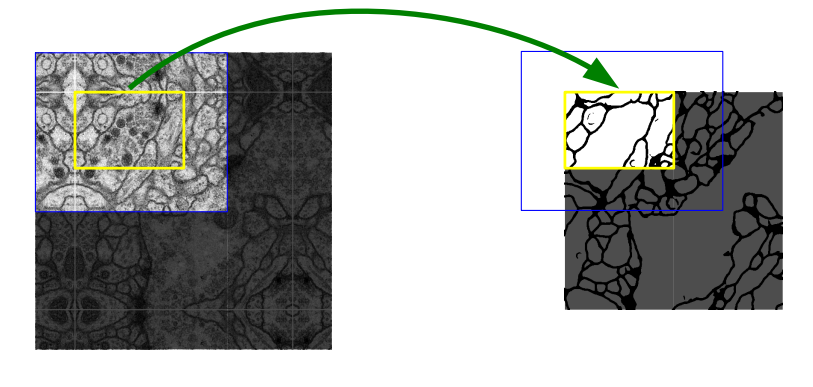
输入原始图像:

模型输出图像:


4.结论
本文主要介绍了U-net模型在细胞分割中的相关基本概念及一个具体应用,下一步,将进一步探究将每两个相邻的卷积层替换为残差结构,并在收缩路径和扩张路径中间加入并联在一起的位置注意力模块和通道注意力模块以进一步提高U-net模型的应用效果。