R|foestplot包绘制森林图forest plot
小编今天给大家分享的是foestplot包绘制组间差异比对图(森林图forest plot)相关方法。森林图(forestplot)常用于Meta分析,可用于表达统计指标的效应量和置信区间。
绘图示例
#安装包
#install.packages("forestplot")
#加载包
library(forestplot)
#输入文件
inputFile="input.txt"
#输出文件
outFile="forest.pdf"
#设置路径
setwd("G:\\RStudio\\38.forest")
#读取文件数据
rt=read.table(inputFile,header=T,sep="\t",row.names=1,check.names=F)
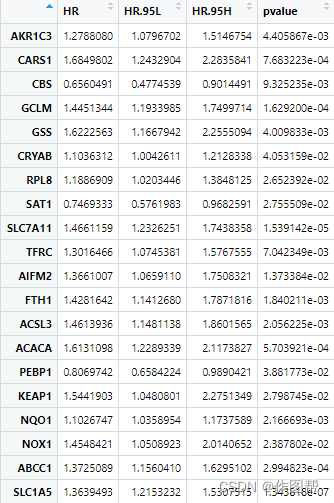
#绘制森林图函数
gene=rownames(rt)#读取基因列
hr=sprintf("%.3f",rt$"HR")#获取HR列取小数点后3位
hrLow=sprintf("%.3f",rt$"HR.95L")#获取95%置信区间取HR.95L列小数点后3位
hrHigh=sprintf("%.3f",rt$"HR.95H")#获取95%置信区间HR.95H列小数点后3位
pVal=ifelse(rt$pvalue<0.001, "<0.001", sprintf("%.3f", rt$pvalue)) #获取P值
Hazard.ratio=paste0(hr,"(",hrLow,"-",hrHigh,")")#合并为一个数据集
#输出图形
#pdf(file=outFile, width = 6, height =4.5)
n=nrow(rt)
nRow=n+1
ylim=c(1,nRow)
layout(matrix(c(1,2),nc=2),width=c(3,2))
#森林图左边的基因信息
xlim = c(0,3)par(mar=c(4,2,1.5,1.5))plot(1,xlim=xlim,ylim=ylim,type="n",axes=F,xlab="",ylab="")#axes=F表示禁止生成坐标轴text.cex=0.8 #放大0.8倍text(0,n:1,gene,adj=0,cex=text.cex)#显示基因列信息
text(1.5-0.5*0.2,n:1,pVal,adj=1,cex=text.cex);text(1.5-0.5*0.2,n+1,'pvalue',cex=text.cex,font=2,adj=1)#显示pvalue列信息text(3,n:1,Hazard.ratio,adj=1,cex=text.cex);text(3,n+1,'Hazard ratio',cex=text.cex,font=2,adj=1,)#显示计算出的Hazard ratio列信息

#绘制森林图
par(mar=c(4,1,1.5,1),mgp=c(2,0.5,0))
xlim = c(0,max(as.numeric(hrLow),as.numeric(hrHigh)))
plot(1,xlim=xlim,ylim=ylim,type="n",axes=F,ylab="",xaxs="i",xlab="Hazard ratio")#设置x轴的标题
arrows(as.numeric(hrLow),n:1,as.numeric(hrHigh),n:1,angle=90,code=3,length=0.03,col="darkblue",lwd=2.5)
abline(v=1,col="black",lty=2,lwd=2) #添加中线,设置中线的位置,颜色,类型,宽度
boxcolor = ifelse(as.numeric(hr) > 1, 'red', 'blue')#设置中线的取值
points(as.numeric(hr), n:1, pch = 15, col = boxcolor, cex=1.5)#pch表示点的样式,设置点的大小,颜色
axis(1)
#dev.off()

完整代码:
library(forestplot)
inputFile="input.txt"
outFile="forest.pdf"
setwd("E:\\forestplot")
rt=read.table(inputFile,header=T,sep="\t",row.names=1,check.names=F)
gene=rownames(rt)
hr=sprintf("%.3f",rt$"HR")
hrLow=sprintf("%.3f",rt$"HR.95L")
hrHigh=sprintf("%.3f",rt$"HR.95H")
pVal=ifelse(rt$pvalue<0.001, "<0.001", sprintf("%.3f", rt$pvalue))
Hazard.ratio=paste0(hr,"(",hrLow,"-",hrHigh,")")
pdf(file=outFile, width = 6, height =4.5)
n=nrow(rt)
nRow=n+1
ylim=c(1,nRow)
layout(matrix(c(1,2),nc=2),width=c(3,2))
xlim = c(0,3)par(mar=c(4,2,1.5,1.5))plot(1,xlim=xlim,ylim=ylim,type="n",axes=F,xlab="",ylab="")text.cex=0.8 text(0,n:1,gene,adj=0,cex=text.cex)
text(1.5-0.5*0.2,n:1,pVal,adj=1,cex=text.cex);text(1.5-0.5*0.2,n+1,'pvalue',cex=text.cex,font=2,adj=1)text(3,n:1,Hazard.ratio,adj=1,cex=text.cex);text(3,n+1,'Hazard ratio',cex=text.cex,font=2,adj=1,)
par(mar=c(4,1,1.5,1),mgp=c(2,0.5,0))
xlim = c(0,max(as.numeric(hrLow),as.numeric(hrHigh)))
plot(1,xlim=xlim,ylim=ylim,type="n",axes=F,ylab="",xaxs="i",xlab="Hazard ratio")
arrows(as.numeric(hrLow),n:1,as.numeric(hrHigh),n:1,angle=90,code=3,length=0.03,col="darkblue",lwd=2.5)
abline(v=1,col="black",lty=2,lwd=2)
boxcolor = ifelse(as.numeric(hr) > 1, 'red', 'blue')
points(as.numeric(hr), n:1, pch = 15, col = boxcolor, cex=1.5)
axis(1)
dev.off()

END
关注”作图帮“微信公众号,获取更多精彩