日前,Kaggle发布了CAFA 5 Protein Function Prediction蛋白质功能预测大赛。这是一个机器学习中的序列预测任务,需要你开发一个基于蛋白质氨基酸序列和其他数据的模型,预测一组蛋白质的功能。
该竞赛评估参与者对蛋白质序列的基因本体论(GeOntology, GO)术语的预测。
测试集分为三个子生物学:分子功能(MolecularFunction,MF)、生物过程(Biological Process,BP)和细胞成分(Cellular Component,CC)。参与者对每个子生物学分别进行评分。最后的性能指标是在三个子生物学上计算的最大F-measures的算术平均值。考虑到GO的层次结构,使用了加权精度和召回率。评估代码是公开的。

Baseline简析
对任何AI项目的建模过程如下(以往期为例):

part1: data preprocess()
1.1 从预训练的蛋白质功能预测模型(ProtBERT, T5等)中形成初始的embedding.

1.2 从train_terms生成标签,通过考虑蛋白质集中最常见的前k个GO项,为每个蛋白质生成长度为K的稀疏向量,用来指示K个GO项在蛋白质中的真实概率(0或1)

part2: build_transform()/ build_dataset()/ build_dataloader()
2.1 组合蛋白质ID以及对应的embedding到pytorch框架

part3: buiild_mode()
3.1 形式化建模为输入形状为(E,) 输出为(K, )的概率,此时可用任何分类模型进行探索实验,例如timm里面若干分类模型. 下面只是简单的CNN1D + MLP


part4: build_loss() & build_metric()
4.1 探索利用分类loss
4.2 利用F1-meature等指标进行验证

part5: train_one_epoch(), eval_one_epoch(), test_one_epoch()
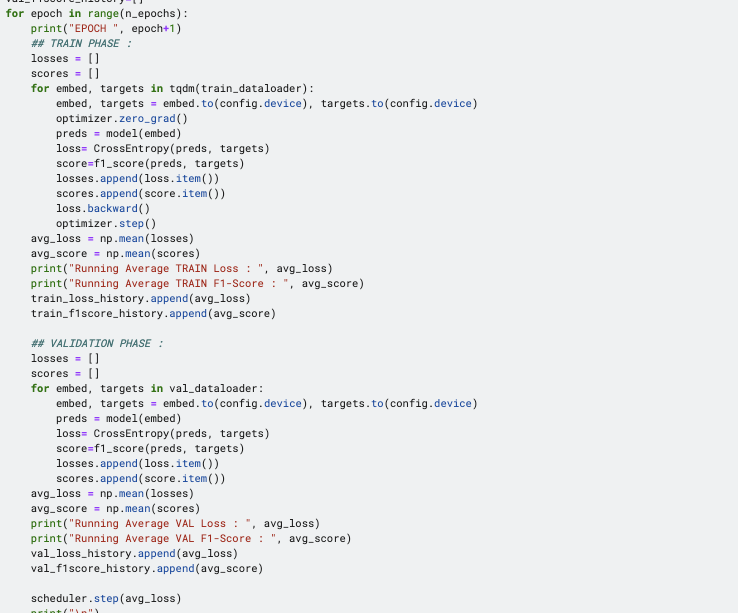
由于篇幅关系,此处只贴部分代码
关注下方【学姐带你玩AI】🚀🚀🚀
回复“蛋白质”领取完整baseline
码字不易,欢迎大家点赞评论收藏!