写在前面
【这图怎么画】系列的图都来自VIP群里同学的提问。推文只是对图片的复现,不代表作者对图片展现形式的认同。欢迎同学们在群里分享有意思的图片。
本期图片
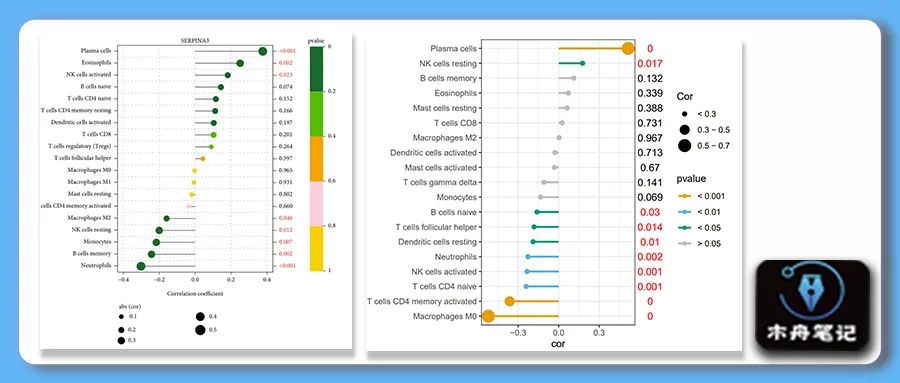
❝Title:Integrated Analysis Identifies Four Genes as Novel Diagnostic Biomarkers Which Correlate with Immune Infiltration in Preeclampsia
Doi:https://doi.org/10.1155/2022/2373694
❞
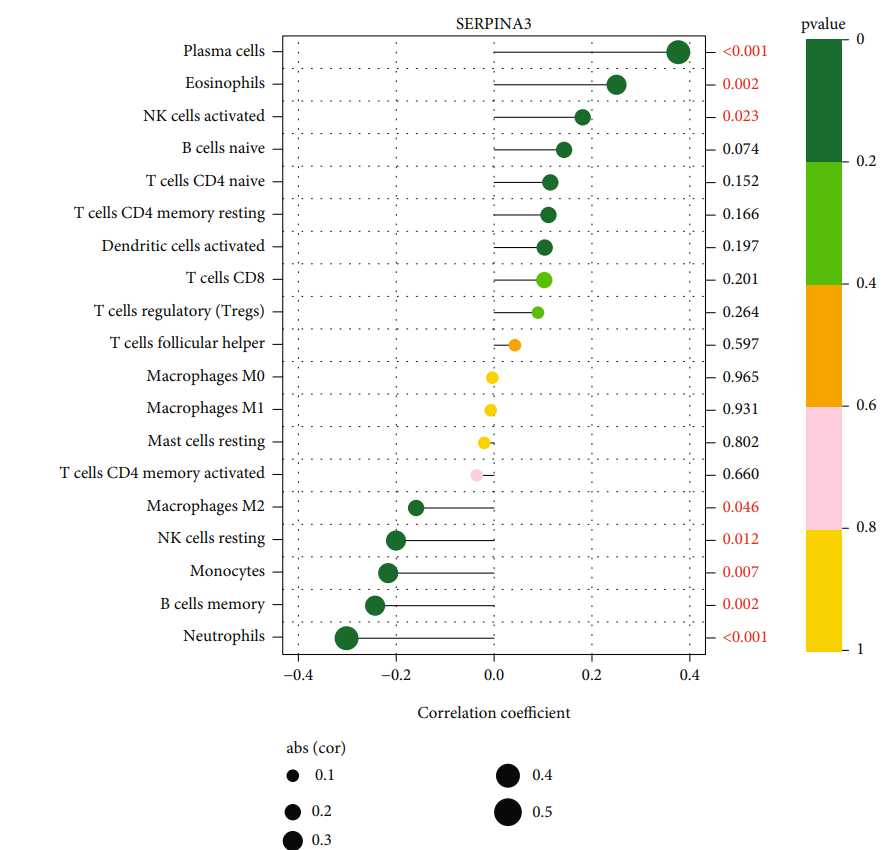
这是一个相关分析结果的展示图。这里作者分别用色阶条以及数值展现了pvalue,用点的大小表示相关系数绝对值。
复现结果
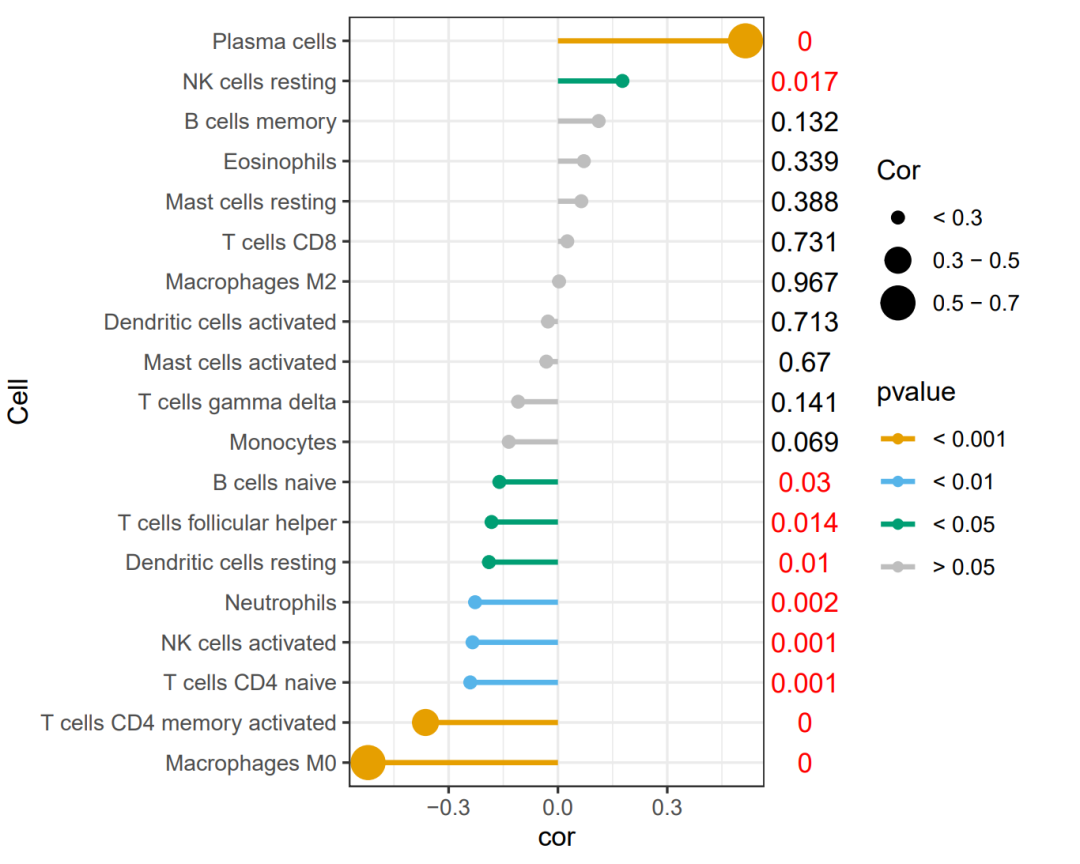
❝稍微调整了一下,原图把p值分层好多段,尤其是>0.05的部分,我觉得意义不大。
❞
示例数据和代码领取
点赞、在看 本文,分享至朋友圈集赞25个并保留30分钟,截图发至微信mzbj0002领取。
「木舟笔记2022年度VIP可免费领取」。
木舟笔记2022年度VIP企划
「权益:」
「2022」年度木舟笔记所有推文示例数据及代码(「在VIP群里实时更新」)。
资源合集 木舟笔记「科研交流群」。
「半价」购买
跟着Cell学作图系列合集(免费教程+代码领取)|跟着Cell学作图系列合集。
「收费:」
「99¥/人」。可添加微信:mzbj0002 转账,或直接在文末打赏。

绘图
dat = read.csv("cor.csv")
head(dat)
# 对相关系数和p值转换为分类变量
dat$cor1 <- cut(abs(dat$cor),# 绝对值breaks = c(0, 0.3, 0.5, 0.7, 0.9, 1),labels = c("< 0.3","0.3 - 0.5","0.5 - 0.7","0.7 - 0.9","> 0.9"),right=FALSE) # right=FALSE表示表示区间为左闭右开
dat$pvalue1 <- cut(dat$pvalue,breaks = c(0, 0.001, 0.01, 0.05, 1),labels = c("< 0.001","< 0.01","< 0.05","> 0.05"),right=FALSE)
# 排序
dat = dat[order(dat$cor),]
dat$Cell = factor(dat$Cell, levels = dat$Cell)
p = ggplot(dat, aes(x = cor, y = Cell, color = pvalue1)) +scale_color_manual(name="pvalue",values = c("#E69F00", "#56B4E9", "#009E73", "gray"))+geom_segment(aes(x = 0, y = Cell, xend = cor, yend = Cell),size = 1) +geom_point(aes(size = cor1))+theme_bw()+labs(size = "Cor")
p## 添加p值
dat$pvalue2 <- cut(dat$pvalue,breaks = c(0, 0.05,1),labels = c("< 0.05","> 0.05"),right=FALSE)
p1 = ggplot()+geom_text(dat,mapping = aes(x = 0, y = Cell, color = pvalue2, label = round(pvalue,3)))+scale_color_manual(name="",values = c("red", "black"))+theme_void()+guides(color=F)p1
library(patchwork)
p|p1ggsave("lollipop.pdf",width = 8,height = 5)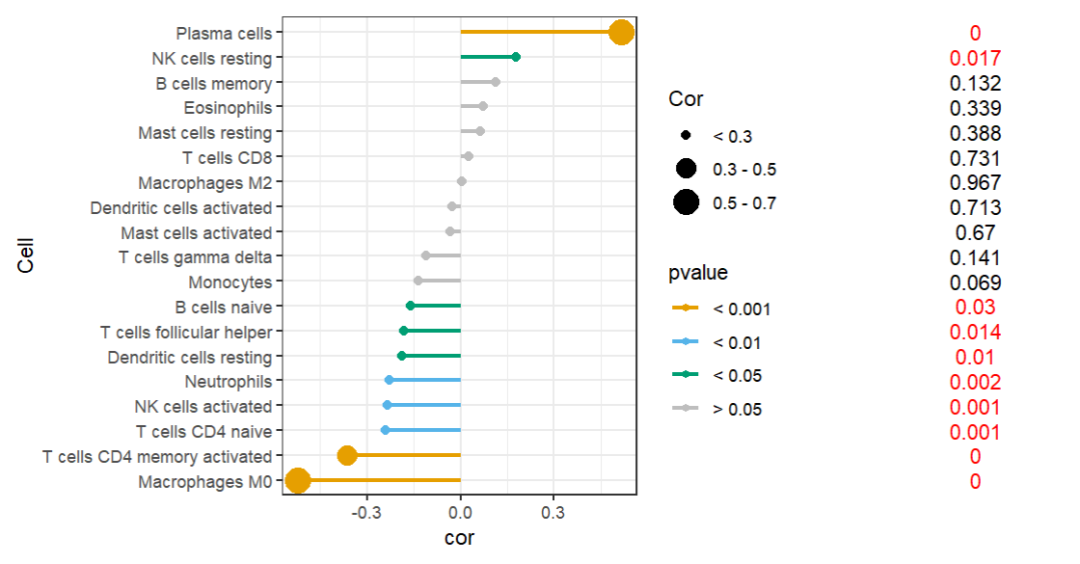
后续在AI里调整一下位置即可~当然用基础函数或其他办法也能够直接画出来,这里我就不想了,有更好的办法的同学可以私信我。
往期内容
(免费教程+代码领取)|跟着Cell学作图系列合集
Q&A | 如何在论文中画出漂亮的插图?
跟着 Cell 学作图 | 桑葚图(ggalluvial)
R实战 | Lasso回归模型建立及变量筛选
跟着 NC 学作图 | 互作网络图进阶(蛋白+富集通路)(Cytoscape)
R实战 | 给聚类加个圈圈(ggunchull)
R实战 | NGS数据时间序列分析(maSigPro)
跟着 Cell 学作图 | 韦恩图(ggVennDiagram)