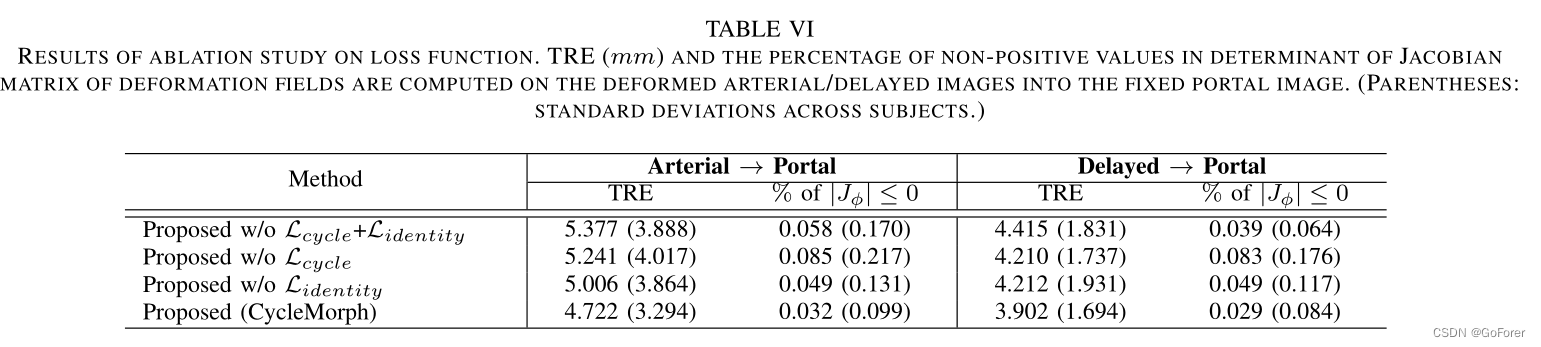
第三代测序技术在微生物研究中的应用
曹晨霞, 韩琬, 张和平
摘要: 1977年Sanger 发明的双末端终止法开启了测序之旅,而测序技术在30多年内不断革新。 每种新技术的出现都有超过前代产品的独特之处,但也会不可避免的存在自身局限性,关键在于掌握每种技术的优缺点并加以合理应用。第三代测序技术是一种集高通量、快速度、长读长及低成本等多种优点于一身的新型测序技术,它的出现为基因组学、转录组学及DNA甲基化等研究 注入了新活力。本文在介绍基本技术原理的基础上,着重概述了第三代测序技术在微生物研究中 的应用,从而揭示了其广泛的应用前景。
关键词: 第三代测序技术 微生物 基因组学 DNA 甲基化
Application of third generation sequencing technology to microbial research
CAO Chen-Xia, HAN Wan, ZHANG He-Ping
Abstract: The dideoxy chain-termination method, which was invented in 1977 by Sanger, has opened the door of DNA sequencing. The sequencing technology has continuously advanced during the past 30 years. Each new technology has unique advantages over the previous generation, but it also has its own limitations. The key is to recognize well both the advantages and disadvantages of each technology so that it can be used reasonably. The third generation sequencing technology is a new technology with many advantages, such as high throughput, fast speed, long read and low cost. Additionally, its emergence has advanced the study of genomics, transcriptomics, epigenomics and so on. In this review, the principle of the third generation sequencing technology is introduced, followed by a summary of its application to microbial research, and its potential uses in other areas.
Key words: The third generation sequencing Microorganism Genomics DNA methylation
DNA测序技术在短短三十几年内不断革新,每种新技术都有超过前代产品的独特之处,但在各具优势的同时又都不可避免的存在一定局限性。 1977年Sanger的双脱氧链核苷酸终止法及Gilbert的化学裂解法并行被称为第一代测序技术[1-2]。第一代测序技术操作简单、读取序列长、准确率高。基于此测序技术,1990年开启了人类基因组计划,并于1995年完成了第一个细菌基因组测序[3]。但一代测序技术缺点是成本高、速度慢、通量低,因此限制了其被大规模商业化使用。第二代测序技术以2005年的Roche GS-FLX 454、Illumina Solexa和ABI SOLiD测序平台为代表,此技术拥有较高通量,可最大缺点是读长短(仅30-450 bp),且扩增过程容易产生引入外源基因而错配的现象。因此随着大数据时代的到来,逐渐出现以Helicos Biosciences公司的单分子DNA测序(True single molecular sequencing,tSMS)、Pacific Bioscience公司的单分子实时测序(Single molecule real time sequencing,SMRT)以及Oxford Nanopore的纳米孔单分子技术为代表的第三代测序技术[4]。
第三代测序技术是一种集高通量、快速度、长读长及低成本等多种优点于一身的新型测序技术。它最大特点是无需进行PCR扩增,可直接读取目标序列,因此假阳性率大大减少,同时避免了碱基替换及偏置等常见PCR错误的发生。就精准度来说,第三代测序技术与第二代测序技术相比并不具有优势,错误率通常在15%左右[5]。但随着测序深度的加大及使用更正软件可达到99.9%的准确率[6],因此第三代测序技术具有广泛的应用前景。本文将介绍目前具代表性的第三代测序技术并着重阐述其在微生物研究中的应用。
1 第三代测序技术基本原理1.1 SMRT技术
SMRT技术是PacBio公司的新型DNA测序技术,其核心在于零模式波导技术(Zero-mode waveguide technology,简称ZMW)[7]。ZMWs实质是一些直径为100 nm、厚度为70 nm的微小纳米孔,此空间正好可容纳一个DNA聚合酶分子,从而使得在此位置可观察到合成DNA链过程(图 1A)。由于成千上万个纳米孔同时作用,因此我们可重复观察到此现象。事实上,此时的DNA聚合酶才是整个测序过程的引擎。DNA聚合酶附着在ZMW孔的底部,身上携带有荧光标记的碱基,每个碱基上有不同颜色的荧光染料。聚合酶以单个DNA分子为模板,当DNA聚合酶读取模板结合不同的碱基时就会发出不同颜色的荧光信号,此时检测器就可由颜色来判别碱基种类。当反应完成后,荧光标记被聚合酶裂解而弥散到孔外,由此完成测序工作(图 1B)。长期以来单分子测序技术最大的瓶颈是在测序过程中生物材料会引起相当大的背景噪音,而SMRT的零模波导技术首次攻克了这一难题[7]。
| 图 1 SMRT测序技术[4]Figure 1 Principle of single-molecule,real-time DNA sequencing[4] |
| 图选项 |
1.2 Oxford Nanopore纳米孔单分子技术
Oxford Nanopore技术不是采用以往“边合成边测序”的方法,而是采用“边解链边测序”的方法。核酸外切酶与α-溶血素纳米孔相耦合是此测序平台的核心[8]。纳米孔外包被有脂质双分子层,在其两端各有一对电极(图 2A)。脂质双分子层两侧为不同的盐浓度,其主要作用是满足外切酶的活性条件。外切酶被共价结合在纳米孔的入口处,当单链DNA模板通过纳米孔时,外切酶会“捕捉”到DNA分子并将碱基剪切下来,使其依次单个通过纳米孔 (图 2B)。已检测过的碱基被很快清除,因此不会出现重复测序现象。Oxford Nanopore技术关键在于控制碱基穿过纳米孔的速度。纳米孔长度仅为 5 nm,因此为保证可监测到每个碱基,要求速度保持在1核苷酸/ms。已证实采用环糊精配接器与 α-溶血素纳米孔共价结合可有效降低其通过速 率[9]。因此当单个碱基通过接有环糊精配接器的纳米孔时,电流会受到干扰,从而根据不同的电流特征来判断相应的核苷酸种类[10]。
| 图 2 纳米孔单分子测序[8,11]Figure 2 Oxford Nanopore technology[8,11] |
| 图选项 |
1.3 tSMS技术
tSMS技术需先对待测DNA样品进行裂解和变性处理以获得多条DNA单链,在其3′末端多聚腺苷酸化,使其带有Poly(A)尾,末端腺苷酸用Cy5荧光染料标记。同时要在末端进行阻断,防止其在测序过程中延伸(图 3A-F)。带有Poly(T)尾的寡聚核苷酸共价结合在玻璃盖片上,其作用是捕获模板,并作为延伸时的引物。这些玻璃盖片被随机放在流动槽里,当二者结合后,CCD相机记录杂交模板所处的位置,建立边合成边测序位点,同时解除Cy5荧光标记(如图 3中的位点1、2和3)。随后与DNA聚合酶和荧光标记的核苷酸(4种碱基之一)相混合,反应完成后洗脱掉未反应的dNTP及DNA聚合酶,最后通过CCD相机在激光作用下读取杂交模板信息(图 3G-N)。当标记解除后,加入下一种核苷酸及DNA聚合酶,新的碱基可被结合,依此反复循环,从而确定碱基序列(图 3中表格)[4, 12]。
| 图 3 Helicos true-single molecule sequencing (tSMS)技术测序样本的制备、碱基加入、清洗及荧光位点切除等示意图12]Figure 3 Helicos true-single molecule sequencing (tSMS) sample preparation and image series illustrating template-specific base addition, successful rinsing, and successful linker cleavage12] |
| 图选项 |
2 第三代测序技术在微生物研究中的应用
微生物在我们的生活中无处不在,据估计地球上微生物细胞达1030个,仅人体中就有100万亿,约是人体自身细胞个数的10倍[13-14]。自然界中虽存在数百万种原核生物,但大多数不可被培养。而细菌微生物在人体中可发挥重要作用,具有调控消化、内分泌系统及免疫系统等功能[15]。随着第三代测序技术的出现,有望实现从基因水平及分子结构掌握微生物发挥功能作用的机理,这对我们今后的科研工作及健康管理具有一定指导意义。
2.1 基因组学研究
2.1.1 全基因组测序:在无参考序列情况下,凭借生物信息学分析方法直接对物种序列进行拼接、组装,最终获得该物种的基因组图谱,称为全基因组测序或从头测序(De novo sequencing)[16]。全基因组测序有助于我们深入了解物种的基因组成及分子进化,目前由第三代测序技术完成的完整基因组测序有很多。Chan等利用三代SMRT技术报告了首个分离自腌制生鱼片的奈氏西地西致病菌SSMD04的完整序列。此前该菌总是分离自多种微生物共存的状态,且其免疫功能严重低下,因此该菌的致病性一直以来不是很清楚。而此次奈氏西地西菌SSMD04全基因组的获得有望增进对其多样基因及功能的认识,最终对其致病性有更深入的了解[17]。同样,Wibberg等利用第三代测序技术完成了对类产碱假单胞菌CECT5344的完整基因组测序[18],由于该菌可消化吸收氰化物并转变为无毒成分而欲将其应用到氰化物污染的治理中。此次类产碱假单胞菌CECT5344的完整测序使我们对其基因组成有了充分认识,同时得到了该菌氰化物同化、氰化物抗性及基因簇等重要信息,预示着它在未来污染物的生物降解及生物塑料的生产中可发挥重要作用。
2.1.2 基因组重测序及比较基因组学:基因组重测序是在已知参考序列的情况下,对同一物种的不同个体进行测序,最终在个体或群体间做出差异性分析(如SNPs、InDels、SVs、CNV及群体多态性分析等),从而获得整个生物群体的遗传特征[16]。经进一步序列比对及构建系统发育树可衍生到比较基因组学和演化基因组学。2011年在德国爆发的腹泻和肠溶血性综合征引起人们极度重视,它是由 一种产志贺毒素的大肠杆菌(O104:H4)所致。Rasko等利用第三代SMRT技术对引起此次疾病暴发的菌株进行了测序,并对来自非洲的7种O104:H4肠致病型大肠杆菌以及属于其它血清型肠致病型大肠杆菌的4株参考菌株进行了从头测序。通过分析基因组数据,显示引起此次德国疾病暴发的致病菌与其它O104:H4菌株不同,是大肠杆菌属的另一分支,因其携带有噬菌体编码的志贺毒素2和耐抗生素基因而导致其危害巨大[19]。甲烷氧化菌是一类可在火山喷发地带等极端条件下生存的疣微菌门微生物,可利用甲烷生成自身所需能量[20]。Anvar等利用PacBio SMRT技术完成了甲烷氧化菌Methylacidiphilum fumariolicum SolV的完整测序,并基于基因及其功能子系统注释信息,与其它甲烷氧化菌尤其是M.Infernorum V4基因组进行了比较,最终构建了该菌的系统发育树,从而使人们对甲烷氧化菌的演化机制有了更清楚的认识[21]。
2.1.3 宏基因组学: 由于大多数微生物不可培养,近年来基于“应用现代基因组学技术直接研究自然状态下某一有机群落的全部微生物而无需分离单一菌株”的宏基因组学逐步显现出巨大优势,以其革命性的方法克服了大多数微生物的不可培养性,并取得了丰硕成果。在一项对动物肠道微生物抗生素抗性的研究中,以奶牛粪便为宏基因组建立DNA文库,以期筛选出对抗生素β-酰胺类、氯霉素类、氨基苷类以及四环素类的抗性基因。Wichmann等利用PacBio测序技术对整个宏基因组进行测序分析,发现了变形菌门、拟杆菌门以及壁厚菌门等中存在多类抗生素抗性基因,且不同菌类中存在一些相同的抗生素抗性基因,表明这些抗生素抗性基因很可能在不同菌类间发生了水平化转移(包括致病菌)[22]。这意味着抗生素抗性基因极有可能从农业生态系统转移到临床、食物系统,进而影响到人类健康。而此次发现也为后续抗生素抗性基因的进一步深入研究提供了重要依据。
2.1.4 第三代测序技术在基因组学研究中的优势:目前报道了许多由前代测序技术未能解决而最终由第三代测序技术完成的对目标菌株的完整测序工作。第三代测序技术为什么会有如此巨大的优势?现可总结为以下3点:(1) 高CG含量的测序。高CG含量通常都是CG富集序列(CG多次重复),因此扩增困难、容易混淆,且对测序量有一定要求。而第三代测序技术无需扩增环节,因此大大提高了高CG含量测序的准确率。(2) 重复序列等复杂结构。染色体、质粒具重复序列或环状、发夹环等复杂结构,给测序带来困难,而第三代测序技术可利用跨越、循环等测序技巧克服此类困难。(3) 更正软件。第三代测序技术虽在精准度上不具有优势,如10 kb左右长度的序列在单向、连续通过SMRT测序仪时精准度仅为82.1%-84.4%,而使用软件更正后可获得>99.99%的精准度[6, 19]。此类软件有pacbioCA、LSC、p-errormolule、PBcR等[5, 23-24]。此外,随着测序深度的增加,测序错误率可被控制在极低范围之内。
2.2 甲基化研究
DNA的修饰作用无处不在,广泛存在于噬菌体、原核生物、真核生物中。这些修饰作用可引发机体多种功能,如人体中的基因沉默、大肠杆菌中复制起点的确定等[25-26]。真核生物中,DNA的修饰作用直接影响细胞的多样性及整个生物的发展进程,一旦出错可导致多种疾病的发生[27-28]。而DNA甲基化是一种重要的修饰作用,在生命活动中起重要角色,可调节染色体的复制、转录过程,并可通过相位变化来调控生物的多样性[26, 29]。DNA甲基化尤其在细菌微生物中起关键作用,可调控基因表达及细胞循环周期,同时具抗突变、维护自身基因组等功能[30]。
DNA甲基化具多样性。限制性修饰系统(Restriction-modification,RM)中存在多种甲基转移酶,细菌微生物通过这些甲基转移酶来实现甲基化作用,而大多数甲基转移酶具高度专一性[31]。DNA甲基化具多样性的具体原因有:(1) 甲基化识别位点数量多。每种甲基转移酶都有其特定的序列识别位点,如RM系统中M基因形成的复杂基因产物决定I型甲基转移酶的识别序列,而人胃部致病菌幽门螺杆菌中含大量的M基因,且每株菌都具一套独特的M基因,因此可呈现出多种甲基转移酶的识别序列,由此表明幽门螺杆菌的多样性甲基化可能[32-33]。 (2) 酶本身结构发生改变。由于RM系统常常处于动态和变化中,其中I型和III型RM系统中的靶标识别区域(Target recognition domain,TRD)本身发生改变,从而导致被识别DNA序列的多样性[34]。如幽门螺杆菌及许多其它细菌的TRD可通过重组侧面重复序列而在TRD1和TRD2区域间发生移动(Domain movement),因此会相应地出现一套不同的识别序列。此外,TRD1和TRD2之间的等位基因重组、点突变以及串联重复序列中拷贝数的改变都是导致幽门螺杆菌甲基化多样性的影响因素[35]。
SMRT第三代测序技术可快速、高效地识别DNA甲基化。其基本原理是依据DNA聚合酶不同的作用时间而产生不同的脉冲间隔来判断甲基化位点。Flusberg等在2010年首次使用SMRT技术准确判定出N6-甲基腺嘌呤(m6A)、5-甲基胞嘧啶(m5C)和5-羟甲基胞嘧啶(5hmC)[36]。Fang等利用SMRT技术成功检测出致病性大肠杆菌基因组中49 311个 6-甲基腺嘌呤(m6A)和1 407个5-甲基胞嘧啶(m5C)的残留量并获得了大量甲基化位点信息,并对每个修饰位点的甲基化几率作出评估[37]。目前也可利用第三代纳米测序技术,由待检物在环糊精内停留时间是胞嘧啶停留时间的2倍而确定此待检物为甲基化的胞嘧啶,且准确率可达到99%[38]。
虽然DNA甲基化在细菌及古菌界普遍存在,但相应的功能还未被广泛、深入的研究。限制性修饰系统最主要的功能是保护宿主免受外源DNA侵入,但目前不少研究报道甲基化作用可影响转录水平的调控,进而影响到基因表达。Fang等发现在限制性修饰系统中若缺失由噬菌体编码的甲基转移酶可引起整个转录过程的变化及基因的扩增,这表明核酸的限制性修饰作用远远超出了仅仅是保护宿主免受外源DNA侵入的作用[37]。此外,Furuta等也进一步证实,若缺失I型RM系统中S基因(甲基化特异性决定基因)的甲基化可引起转录组改变,最终导致细胞表型发生变化,从而设想此机制可对适应性进化做出贡献[39]。
笔者参与益生菌Lactobacillus casei Zhang (L. casei Zhang)和Lactobacillus plantarum P-8 (L. plantarum P-8)甲基化组的破译工作。经生物信息学分析,预测L.casei Zhang里存在2种与II型RM系统相关的甲基转移酶(属于N12类型的m6A甲基转移酶)。这2种甲基转移酶的基因标识分别为LCAZH_2054和LCAZH_2056,且由LCAZH_2054编码的蛋白质增加有一个具催化活性的m6A特异性保守位点(IPR002052)。基于以上预测,Zhang等利用SMRT技术对L.casei Zhang菌株的甲基化水平进行了鉴定,其中1个活性m6A甲基转移酶在L. casei Zhang中得到证实。进一步分析,将5′-ACRCm6AG-3′确定为L.casei Zhang菌株m6A的识别序列,由此可知在L. cas ei Zhang菌株中存在 1 906个甲基化位点,且几乎所有(>99.8%)甲基化位点与此序列相匹配。然而在L.plantarum P-8菌株中,虽预测存在I型、II型RM系统相关成分,但经SMRT测序分析表明L.plantarum P-8中并无具活性的甲基转移酶[40]。之前还没有对乳杆菌属甲基化的相关报道,而此次对这2株菌的甲基化研究是否可代表其整个物种水平还有待考究。相信随着第三代测序技术的深入发展,DNA甲基化位点信息及其生物学功能作用会进一步得到揭示。
2.3 第三代测序技术的其它应用
转录组学是研究基因结构和功能的基础和切入点。通过转录组测序,我们可获得转录位点、可替换剪切、突变位点以及转录本的多种表达等重要信息[16]。但对RNA的传统研究大多需先将RNA先转化为cDNA,然后再对DNA链进行相关分析,即是一种间接的RNA分析方法。不言而喻,此方法出现的错误率较高。主要原因是此过程中模板的转换及2种反转录酶(即依赖于RNA的DNA聚合酶和依赖于DNA的DNA聚合酶)的作用使得基因表达的错误率增大[41-42]。这些因素阻碍了对RNA的准确分析,因此迫切需要一种新方法来解决此问题。tSMS第三代单分子测序技术成功实现了对RNA的直接测序,无需转化cDNA及扩增过程,从而在很大程度上提高了序列分析的精准度,且具有较高通量。Ozsolak等利用此技术完成了对啤酒酵母RNA的测序,其基本原理就是利用包被Poly(dT)寡核苷酸探针的表面来捕获小片段RNA的Poly(A)尾的边合成边测序过程[43]。16S rRNA基因是目前识别环境微生物群落的重要依据,采用SMRT技术有望实现对16S rRNA基因的全长测序,这有助于我们更深入地了解微生物在生态方面的功能,并对整个细菌属的功能作用作出评价[44]。
正如对DNA测序一样,也可利用纳米孔单分子技术对蛋白质进行测序。纳米孔单分子技术对蛋白质测序一直存有两大挑战,其一是测序前必需打开蛋白质的高级结构从而使蛋白质得以穿过纳米孔传感器;其二是尽管多肽链上电荷分布不均匀,但要求多肽链在通过纳米孔感应区域时必需保证能够进行单向迁移。Nivala等利用酶驱动法成功解决了这一技术瓶颈,即用Escherichia coli解叠酶ClpX作为驱动分子,利用ATP降解蛋白质并驱动肽链片段通过纳米孔,最终在此过程中根据不同的离子流特征辨析出蛋白质片段的氨基酸序列[45]。由此表明,纳米孔单分子技术也可用来对蛋白质序列进行相关研究。然而,虽蛋白质可通过纳米孔,但如何快速打开蛋白质的高级结构并使其大量通过纳米孔的技术还尚未成熟,相关的蛋白质移位控制技术还有待提高。
3 展望
第三代测序技术无疑推动了生物、医疗、化学、计算机等多领域的发展。科研者们可实现低成本、短时间对物种的认识,从而合理地设计实验方案,加快物种的研究进程;相信人类个人基因组档案的建立已为时不远,届时可实现对遗传性疾病的诊断和治疗,并用于人类医疗保健的指导。每代测序技术都有其优缺点,新测序技术的出现并不否定原有的测序技术,关键在于掌握各种技术的优缺点,根据实际问题选用合理的测序手段。如可利用第二代测序技术的高精准度结合第三代测序技术的长读长优势来应对今后繁重的测序任务,且这种“2+3”的模式目前已被很有效的应用。
二、三代测序数据的整合通常需2个步骤,即利用二代测序数据对三代测序数据进行更正及更正序列的从头组装。当然这其中也存在一些困难和挑战,具体为:(1) 这种“杂交”的测序方法通常要求制备至少2个不同的测序文库,这对人力和物力的消耗都是一个不小的挑战;(2) 基因组组装实质上是利用一些组装算法对序列进行基因组的重新构建,而通常这些组装算法对文库及基因组大小有一定要求,如短的叠连群中的错误就可能“逃脱”而未被更正,太大或太小的基因组都可能超出其作用范围;或者不同的组装算法对不同区域,如外显子、内含子等的组装都有其不同的局限性。因此,为了更好地实施这一方案,未来我们需开发更精确、更广适用性的组装算法,提升硬件和软件实力,并在技术选取和相对成本上做出权衡。
我国于2009年开启了第三代测序技术的研究,主要的研发机构有中国科学院北京基因组研究所联合中国科学院半导体研究所、深圳华因康基因科技有限公司及无锡艾吉因生物信息技术有限公司。但基本属于起步阶段,主要还是依靠吸收引进,因此还需国人共同努力,加大投入,加强合作,提升创新意识,争取早日有所突破。虽然测序技术的发展带来了些许成就,但我们仍面临许多挑战。相对于整个物种来说,已被测序的物种还只是“冰山一角”,但带来的海量数据对后续生物信息学分析及有用信息的提取都具有一定挑战,因此还需我们继续努力。由于其建立时间较短,目前还未被大规模商业化使用。但相信在不久的将来,第三代测序技术会被不断普及到多个领域,可能成为未来实验室分析及临床诊断的常规检测手段。
参考文献
| [1] | Sanger F, Coulson AR. A rapid method for determining sequences in DNA by primed synthesis with DNA polymerase[J]. Journal of Molecular Biology , 1975, 94 (3) : 441–446. DOI:10.1016/0022-2836(75)90213-2 |
| [2] | Langhorst J, Elsenbruch S, Koelzer J, et al. Noninvasive markers in the assessment of intestinal inflammation in inflammatory bowel diseases: performance of fecal lactoferrin, calprotectin, and PMN-elastase, CRP, and clinical indices noninvasive assessment of inflammation in IBD[J]. The American Journal of Gastroenterology , 2008, 103 (1) : 162–169. DOI:10.1111/j.1572-0241.2007.01556.x |
| [3] | Collins FS, Morgan M, Patrinos A. The Human Genome Project: lessons from large-scale biology[J]. Science , 2003, 300 (5617) : 286–290. DOI:10.1126/science.1084564 |
| [4] | Treffer R, Deckert V. Recent advances in single-molecule sequencing[J]. Current Opinion in Biotechnology , 2010, 21 (1) : 4–11.DOI:10.1016/j.copbio.2010.02.009 |
| [5] | Liao YC, Lin SH, Lin HH. Completing bacterial genome assemblies: strategy and performance comparisons[J]. Scientific Reports , 2015, 5 : 8747. DOI:10.1038/srep08747 |
| [6] | Shin SC, Ahn DH, Kim SJ, et al. Advantages of single-molecule real-time sequencing in high-GC content genomes[J]. PLoS One , 2013, 8 (7) : e68824. DOI:10.1371/journal.pone.0068824 |
| [7] | McCarthy A. Third generation DNA sequencing: pacific biosciences' single molecule real time technology[J]. Chemistry & Biology , 2010, 17 (7) : 675–676. |
| [8] | Munroe DJ, Harris TJR. Third-generation sequencing fireworks at Marco Island[J]. Nature Biotechnology , 2010, 28 (5) : 426–428. DOI:10.1038/nbt0510-426 |
| [9] | Banerjee A, Mikhailova E, Cheley S, et al. Molecular bases of cyclodextrin adapter interactions with engineered protein nanopores[J]. Proceedings of the National Academy of Sciences of the United States of America , 2010, 107 (18) : 8165–8170.DOI:10.1073/pnas.0914229107 |
| [10] | Clarke J, Wu HC, Jayasinghe L, et al. Continuous base identification for single-molecule nanopore DNA sequencing[J]. Nature Nanotechnology , 2009, 4 (4) : 265–270. DOI:10.1038/nnano.2009.12 |
| [11] | Schadt EE, Turner S, Kasarskis A. A window into third-generation sequencing[J]. Human Molecular Genetics , 2010, 19 (R2) : R227–R240. DOI:10.1093/hmg/ddq416 |
| [12] | Harris TD, Buzby PR, Babcock H, et al. Single-molecule DNA sequencing of a viral genome[J]. Science , 2008, 320 (5872) : 106–109. DOI:10.1126/science.1150427 |
| [13] | Turnbaugh PJ, Gordon JI. An invitation to the marriage of metagenomics and metabolomics[J]. Cell , 2008, 134 (5) : 708–713.DOI:10.1016/j.cell.2008.08.025 |
| [14] | Savage DC. Microbial ecology of the gastrointestinal tract[J]. Annual Review of Microbiology , 1977, 31 (1) : 107–133.DOI:10.1146/annurev.mi.31.100177.000543 |
| [15] | Saad N, Delattre C, Urdaci M, et al. An overview of the last advances in probiotic and prebiotic field[J]. LWT-Food Science and Technology , 2013, 50 (1) : 1–16. |
| [16] | Zhu QL, Liu S, Gao P, et al. High-throughput sequencing technology and its application[J]. Journal of Northeast Agricultural University (English Edition) , 2014, 21 (3) : 84–96. DOI:10.1016/S1006-8104(14)60073-8 |
| [17] | Chan KG, Tan KH, Yin WF, et al. Complete genome sequence of Cedecea neteri strain SSMD04, a bacterium isolated from pickled mackerel sashimi[J]. Genome Announcements , 2014, 2 (6) : e01339–e01414. |
| [18] | Wibberg D, Luque-Almagro VM, Ige?o MI, et al. Complete genome sequence of the cyanide-degrading bacterium Pseudomonas pseudoalcaligenes CECT5344[J]. Journal of Biotechnology , 2014, 175 : 67–68. DOI:10.1016/j.jbiotec.2014.02.004 |
| [19] | Rasko DA, Webster DR, Sahl JW, et al. Origins of the E. coli strain causing an outbreak of hemolytic-uremic syndrome in Germany[J]. The New England Journal of Medicine , 2011, 365 (8) : 709–717. DOI:10.1056/NEJMoa1106920 |
| [20] | Hanson RS, Hanson TE. Methanotrophic bacteria[J]. Microbiological Reviews , 1996, 60 (2) : 439–471. |
| [21] | Anvar SY, Frank J, Pol A, et al. The genomic landscape of the verrucomicrobial methanotroph Methylacidiphilum fumariolicum SolV[J]. BMC Genomics , 2014, 15 (1) : 914. DOI:10.1186/1471-2164-15-914 |
| [22] | Wichmann F, Udikovic-Kolic N, Andrew S, et al. Diverse antibiotic resistance genes in dairy cow manure[J]. MBio , 2014, 5 (2) : e01017. |
| [23] | Koren S, Schatz MC, Walenz BP, et al. Hybrid error correction and de novo assembly of single-molecule sequencing reads[J]. Nature Biotechnology , 2012, 30 (7) : 693–700. DOI:10.1038/nbt.2280 |
| [24] | Au KF, Underwood JG, Lee L, et al. Improving PacBio long read accuracy by short read alignment[J]. PLoS One , 2012, 7 (10) : e46679. DOI:10.1371/journal.pone.0046679 |
| [25] | Wion D, Casadesús J. N6-methyl-adenine: an epigenetic signal for DNA-protein interactions[J]. Nature Reviews Microbiology , 2006, 4 (3) : 183–192. DOI:10.1038/nrmicro1350 |
| [26] | Marinus MG, Casadesus J. Roles of DNA adenine methylation in host-pathogen interactions: mismatch repair, transcriptional regulation, and more[J]. FEMS Microbiology Reviews , 2009, 33 (3) : 488–503. DOI:10.1111/j.1574-6976.2008.00159.x |
| [27] | Kong A, Steinthorsdottir V, Masson G, et al. Parental origin of sequence variants associated with complex diseases[J]. Nature , 2009, 462 (7275) : 868–874. DOI:10.1038/nature08625 |
| [28] | Deng J, Shoemaker R, Xie B, et al. Targeted bisulfite sequencing reveals changes in DNA methylation associated with nuclear reprogramming[J]. Nature Biotechnology , 2009, 27 (4) : 353–360. DOI:10.1038/nbt.1530 |
| [29] | Casadesús J, Low D. Epigenetic gene regulation in the bacterial world[J]. Microbiology and Molecular Biology Reviews , 2006, 70 (3) : 830–856. DOI:10.1128/MMBR.00016-06 |
| [30] | Low DA, Casadesús J. Clocks and switches: bacterial gene regulation by DNA adenine methylation[J]. Current Opinion in Microbiology , 2008, 11 (2) : 106–112. DOI:10.1016/j.mib.2008.02.012 |
| [31] | Suzuki MM, Bird A. DNA methylation landscapes: provocative insights from epigenomics[J]. Nature Reviews Genetics , 2008, 9 (6) : 465–476. DOI:10.1038/nrg2341 |
| [32] | Murray NE. Type I restriction systems: sophisticated molecular machines (a legacy of Bertani and Weigle)[J]. Microbiology and Molecular Biology Reviews , 2000, 64 (2) : 412–434. DOI:10.1128/MMBR.64.2.412-434.2000 |
| [33] | Vale FF, Mégraud F, Vítor JMB. Geographic distribution of methyltransferases of Helicobacter pylori: evidence of human host population isolation and migration[J]. BMC Microbiology , 2009, 9 : 193. DOI:10.1186/1471-2180-9-193 |
| [34] | Furuta Y, Kobayashi I. Movement of DNA sequence recognition domains between non-orthologous proteins[J]. Nucleic Acids Research , 2012, 40 (18) : 9218–9232. DOI:10.1093/nar/gks681 |
| [35] | Furuta Y, Kobayashi I. Mobility of DNA sequence recognition domains in DNA methyltransferases suggests epigenetics-driven adaptive evolution[J]. Mobile Genetic Elements , 2012, 2 (6) : 292–296. DOI:10.4161/mge.23371 |
| [36] | Flusberg BA, Webster DR, Lee JH, et al. Direct detection of DNA methylation during single-molecule, real-time sequencing[J]. Nature Methods , 2010, 7 (6) : 461–465. DOI:10.1038/nmeth.1459 |
| [37] | Fang G, Munera D, Friedman DI, et al. Genome-wide mapping of methylated adenine residues in pathogenic Escherichia coli using single-molecule real-time sequencing[J]. Nature Biotechnology , 2012, 30 (12) : 1232–1239. DOI:10.1038/nbt.2432 |
| [38] | Li MS, Zhao M. The mechanisms of the third generation sequencing technology[J]. Progress in Modern Biomedicine , 2012, 12 (10) : 1980–1982. (in chinese) 李明爽, 赵敏. 第三代测序基本原理[J]. 现代生物医学进展 , 2012, 12 (10) : 1980–1982. |
| [39] | Furuta Y, Namba-Fukuyo H, Shibata TF, et al. Methylome diversification through changes in DNA methyltransferase sequence specificity[J]. PLoS Genetics , 2014, 10 (4) : e1004272. DOI:10.1371/journal.pgen.1004272 |
| [40] | Zhang WY, Sun ZH, Menghe B, et al. Short communication: Single molecule, real-time sequencing technology revealed species- and strain-specific methylation patterns of 2 Lactobacillus strains[J]. Journal of Dairy Science , 2015, 98 (5) : 3020–3024.DOI:10.3168/jds.2014-9272 |
| [41] | Cocquet J, Chong A, Zhang GL, et al. Reverse transcriptase template switching and false alternative transcripts[J]. Genomics , 2006, 88 (1) : 127–131. DOI:10.1016/j.ygeno.2005.12.013 |
| [42] | Roy SW, Irimia M. When good transcripts go bad: artifactual RT-PCR ‘splicing’ and genome analysis[J]. BioEssays , 2008, 30 (6) : 601–605. DOI:10.1002/bies.v30:6 |
| [43] | Ozsolak F, Platt AR, Jones DR, et al. Direct RNA sequencing[J]. Nature , 2009, 461 (7265) : 814–818. DOI:10.1038/nature08390 |
| [44] | Mosher JJ, Bowman B, Bernberg EL, et al. Improved performance of the PacBio SMRT technology for 16S rDNA sequencing[J]. Journal of Microbiological Methods , 2014, 104 : 59–60. DOI:10.1016/j.mimet.2014.06.012 |
| [45] | Nivala J, Marks DB, Akeson M. Unfoldase-mediated protein translocation through an α-hemolysin nanopore[J]. Nature Biotechnology , 2013, 31 (3) : 247–250. DOI:10.1038/nbt.2503 |



![[转]杂谈如何绕过WAF(Web应用防火墙)](http://static.wooyun.org/201402/2014022614071092308.png)