参考:
https://github.com/evolutionaryscale/esm
通过GPT模型原理,输入蛋白质序列等模态输出预测的蛋白质序列及结构
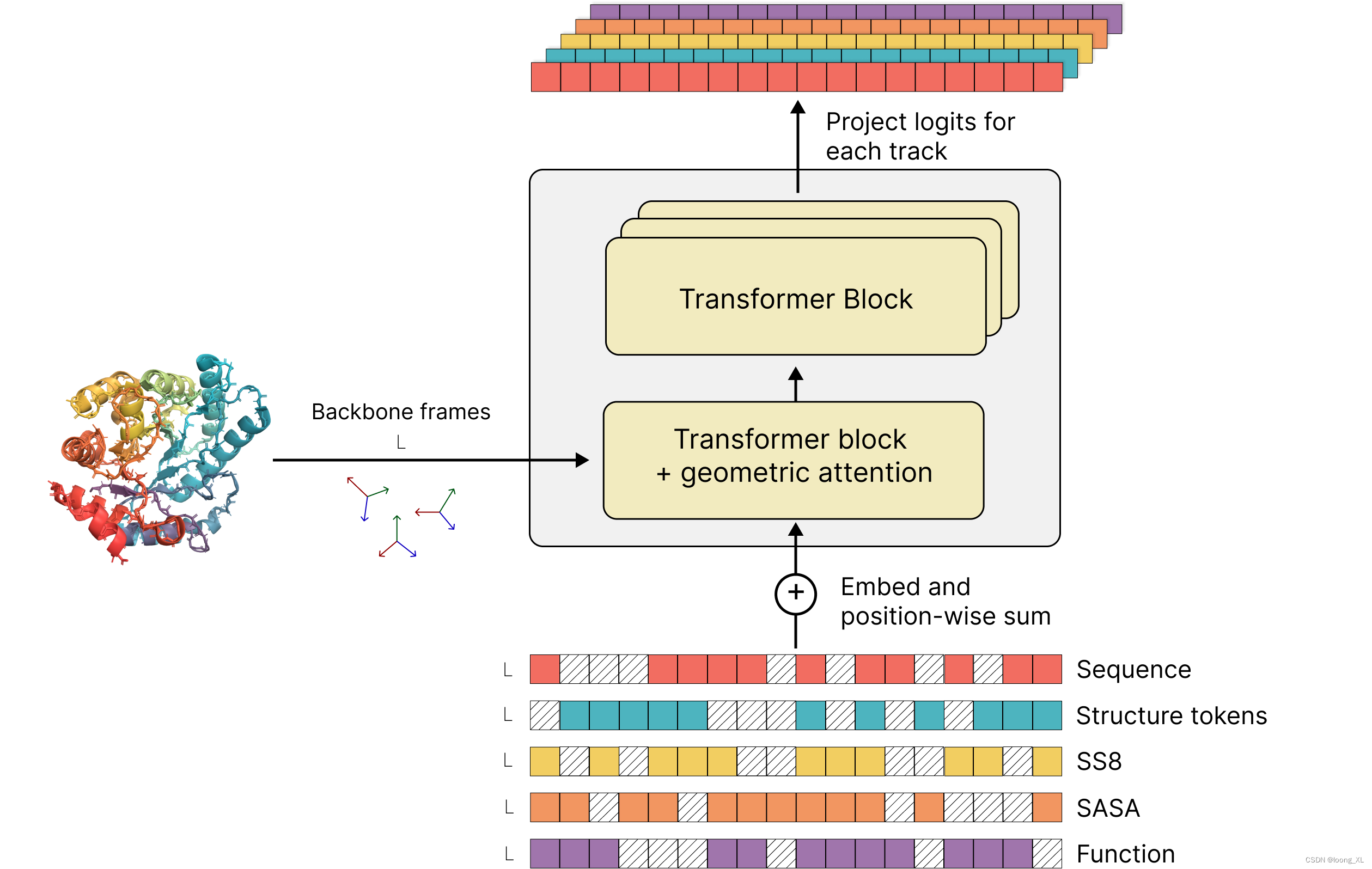
使用
参考:https://colab.research.google.com/github/evolutionaryscale/esm/blob/main/examples/generate.ipynb#scrollTo=Ta7VVnJLy7Wd
安装:
pip install esm
huggingface下载模型这个需要token:
https://huggingface.co/settings/tokens
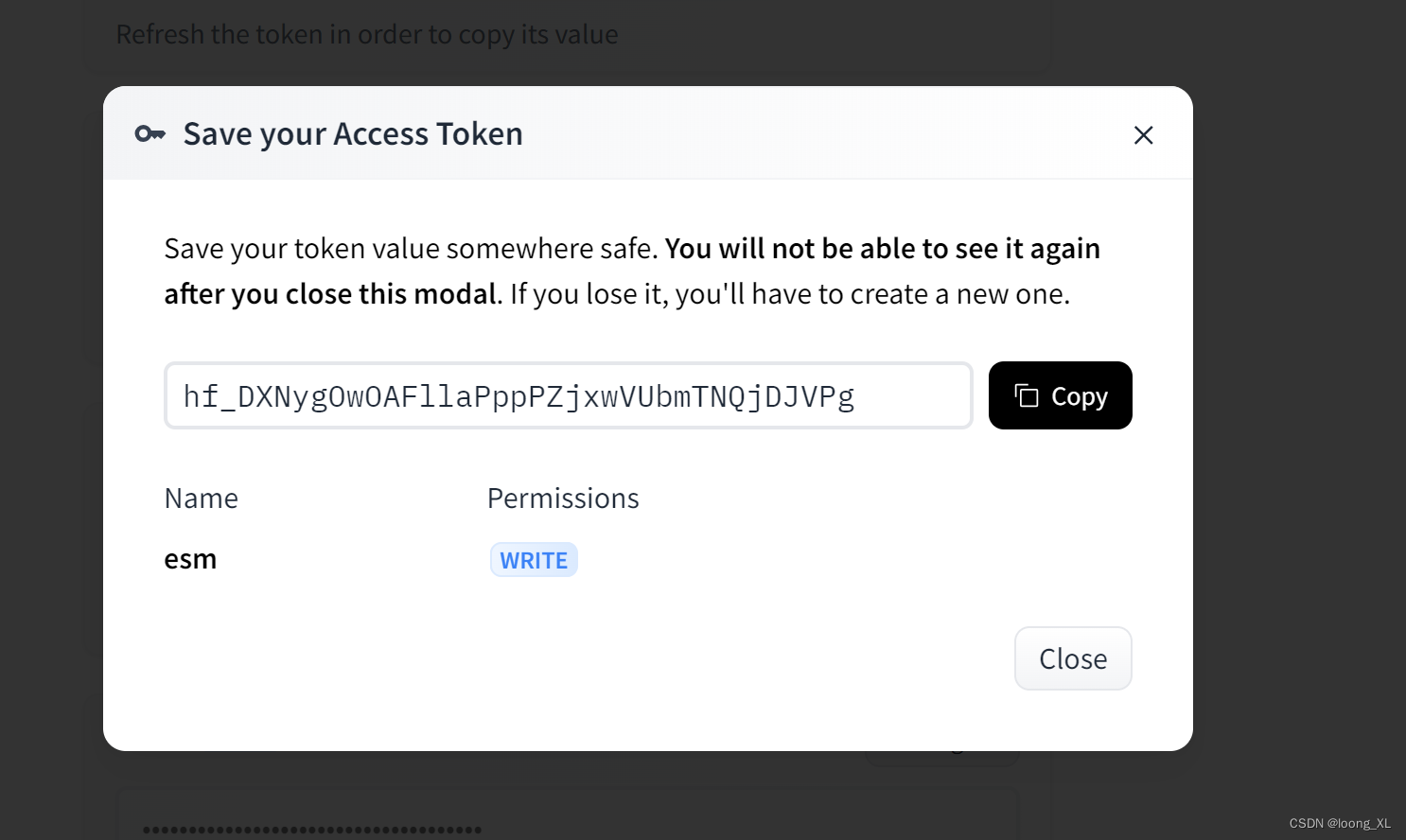
from huggingface_hub import login
from esm.models.esm3 import ESM3
from esm.sdk.api import ESM3InferenceClient, ESMProtein, GenerationConfig# This will prompt you to get an API key from huggingface hub, make one with
# "Read" or "Write" permission and copy it back here.
login()# This will download the model weights and instantiate the model on your machine.
model: ESM3InferenceClient = ESM3.from_pretrained("esm3_sm_open_v1").to("cuda") # or "cpu"# Generate a completion for a partial Carbonic Anhydrase (2vvb)
prompt = "___________________________________________________DQATSLRILNNGHAFNVEFDDSQDKAVLKGGPLDGTYRLIQFHFHWGSLDGQGSEHTVDKKKYAAELHLVHWNTKYGDFGKAVQQPDGLAVLGIFLKVGSAKPGLQKVVDVLDSIKTKGKSADFTNFDPRGLLPESLDYWTYPGSLTTPP___________________________________________________________"
protein = ESMProtein(sequence=prompt)
# Generate the sequence, then the structure. This will iteratively unmask the sequence track.
protein = model.generate(protein, GenerationConfig(track="sequence", num_steps=8, temperature=0.7))
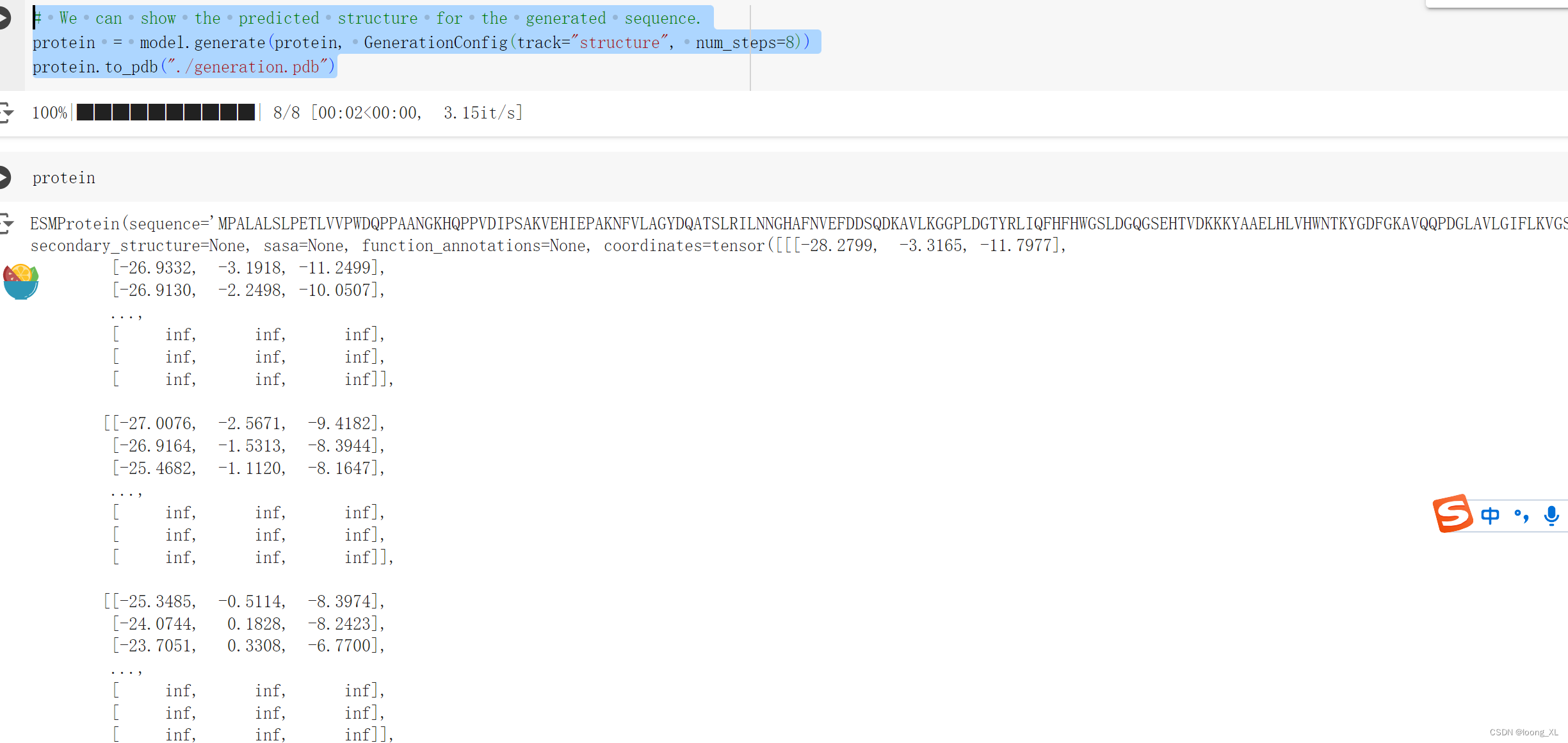
# We can show the predicted structure for the generated sequence.
protein = model.generate(protein, GenerationConfig(track="structure", num_steps=8))
protein.to_pdb("./generation.pdb")
# Then we can do a round trip design by inverse folding the sequence and recomputing the structure
protein.sequence = None
protein = model.generate(protein, GenerationConfig(track="sequence", num_steps=8))
protein.structure = None
protein = model.generate(protein, GenerationConfig(track="structure", num_steps=8))
protein.to_pdb("./round_tripped.pdb")
输入prompt一小段蛋白质序列,模型延迟生成_未知位置的序列
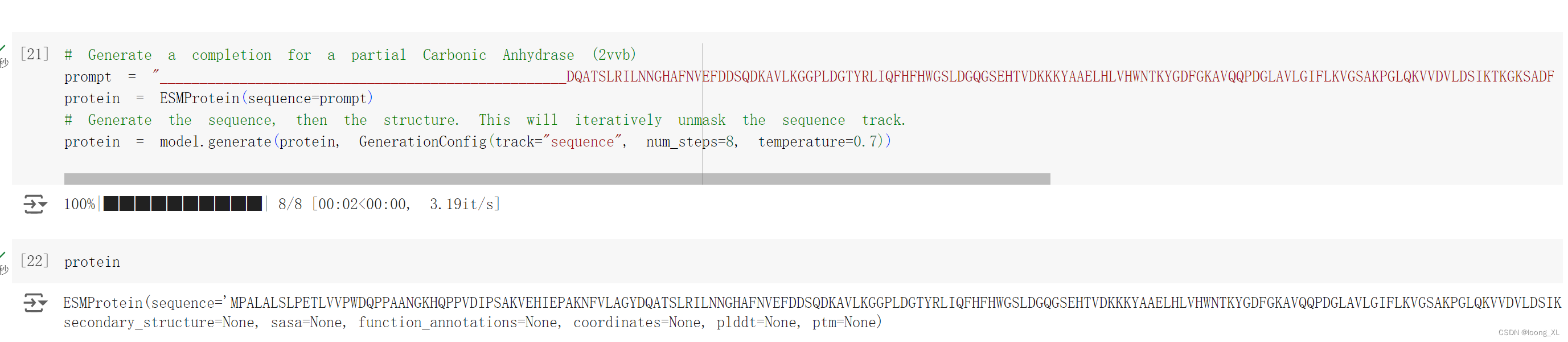
比如这两端各有3个_需要生成6个token填充_
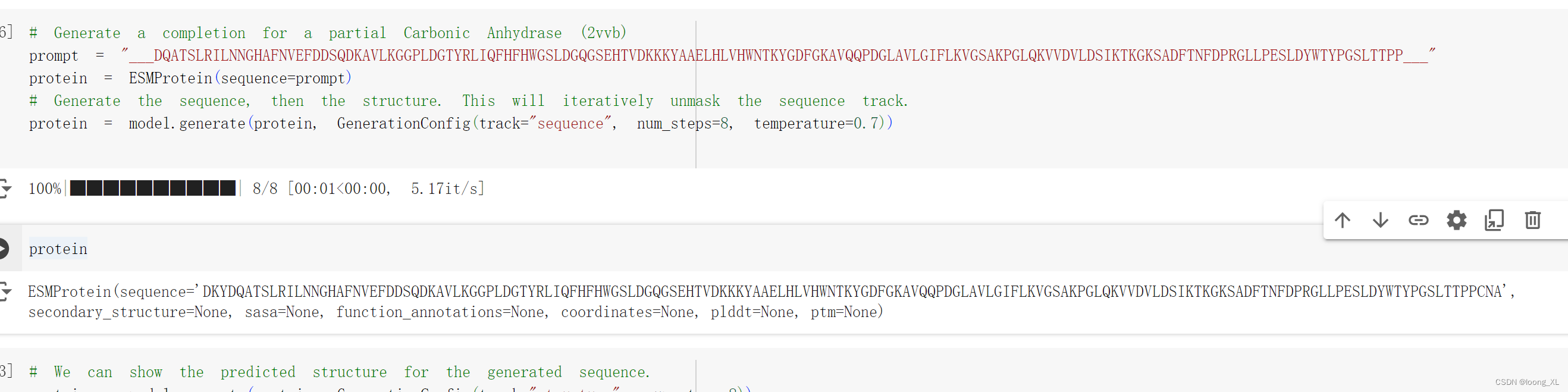
再把生成的序列预测结构:
# We can show the predicted structure for the generated sequence.
protein = model.generate(protein, GenerationConfig(track="structure", num_steps=8))
protein.to_pdb("./generation.pdb")
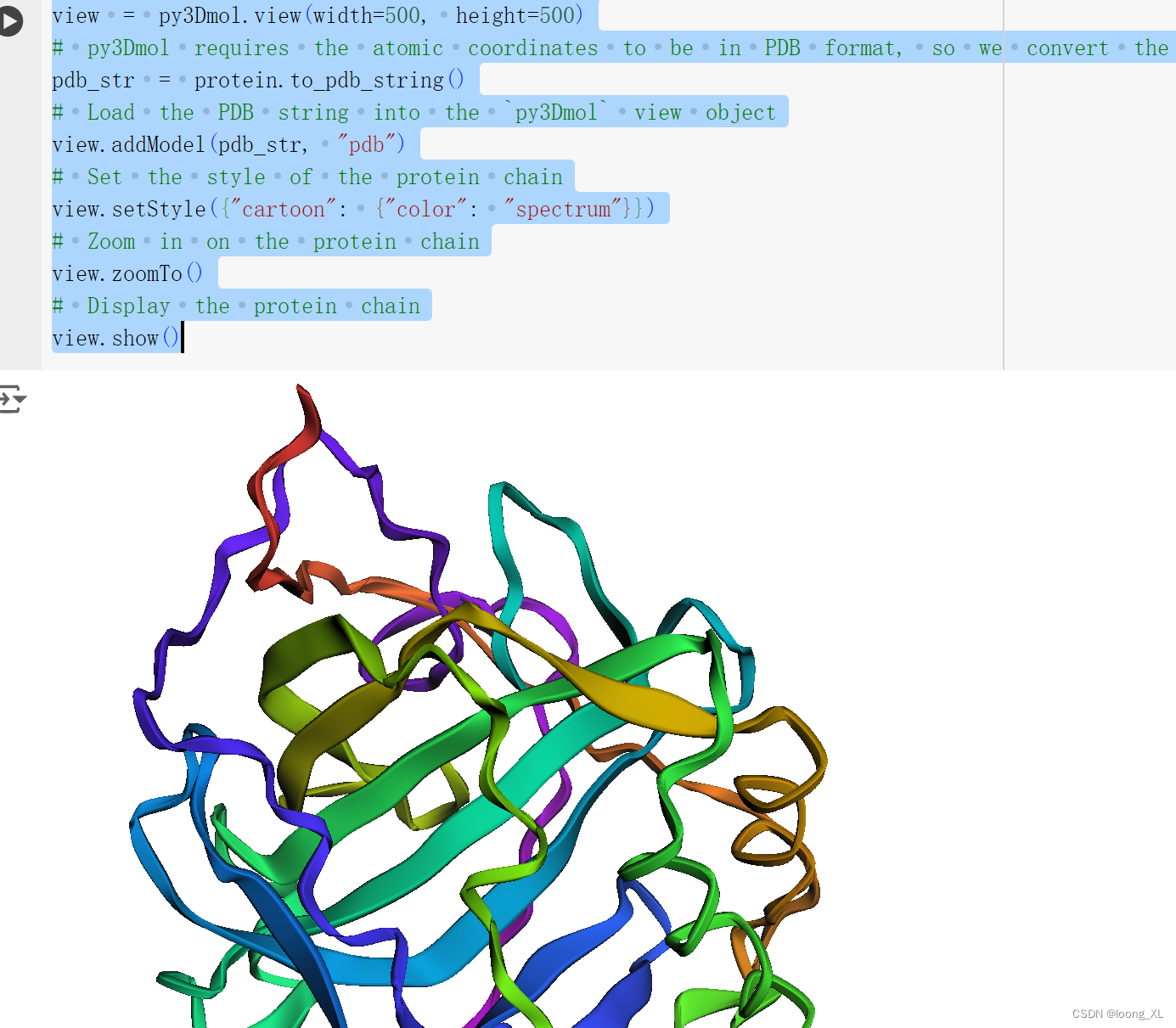
蛋白质结构pymol展示:
!pip install py3Dmol
import py3Dmol# First we can create a `py3Dmol` view object
view = py3Dmol.view(width=500, height=500)
# py3Dmol requires the atomic coordinates to be in PDB format, so we convert the `ProteinChain` object to a PDB string
pdb_str = protein.to_pdb_string()
# Load the PDB string into the `py3Dmol` view object
view.addModel(pdb_str, "pdb")
# Set the style of the protein chain
view.setStyle({"cartoon": {"color": "spectrum"}})
# Zoom in on the protein chain
view.zoomTo()
# Display the protein chain
view.show()