文章目录
- 一、基本知识
- 1.1、各向同性(isotropic) + 各向异性(anisotropic)
- 1.2、像素尺寸 + 像素间距 + 像素分辨率
- 1.3、点扩散函数(PSF)
- 1.3.1、高斯函数 + 二维高斯PSF
- 1.3.2、二维高斯PSF:代码生成
- 一、Isotropic Reconstruction
- 1.1、研究背景
- 1.1.1、当前现状 —— 荧光显微镜图像:(横向)各向同性 + (轴向)各向异性
- 1.1.2、主要原因 —— Z轴各向异性 + Z轴采样率低
- 1.1.3、解决方法 —— 基于CARE的各向同性重建
- 1.1.3.1、CARE:研究背景
- 1.1.3.2、CARE:是一种数据生成策略
- 1.1.3.3、最新文章
- 1.2、网络模型(IsoNet)
- 1.2.1、算法流程(原理)
- 1.2.2、数据生成(图解)
- 1.2.3、网络架构
- 1.3、实验数据
- 1.3.1、效果展示
- 1.3.2、不同PSF下的实验结果:高斯PSF + 共聚焦PSF + 光片PSF
- 1.3.3、设置参数:dowmSample + PSF
- 1.3.4、经验结论
- 1.4、成像原理
- 1.4.1、理想的显微镜3D图像:g(x,y,z) -> f(x,y,z)
- 1.4.2、模拟的显微镜2D图像:g(x,y) -> p(x,y) -> g(x,z)
- 二、环境配置
- 2.1、创建虚拟环境
- 2.2、配置相关环境
- 2.3、配置Tensorflow
- 2.3.1、配置Tensorflow(CPU)
- 2.3.1、配置Tensorflow(GPU)
- 2.3.1.1、ImportError: Matplotlib requires numpy>=1.20; you have 1.19.5
- 2.3.1.2、Debug:Could not locate zlibwapi.dll. Please make sure it is in your library path!
一、基本知识
1.1、各向同性(isotropic) + 各向异性(anisotropic)
| 图像在不同方向上 | 特征 | |
|---|---|---|
各向同性 | 相似 | 呈现圆形、没有明显的方向性 |
各向异性 | 不同 | 呈现椭圆形、沿某个方向拉伸 |
1.2、像素尺寸 + 像素间距 + 像素分辨率
图形化理解:请看该篇文章【3.2.2、图解 - 度量metric】
像素尺寸(Size):表示长宽高对应的像素个数像素间距(spacing ):表示两个像素的物理间距(又叫光学分辨率)像素分辨率(Physical extent):表示单个像素的物理尺寸(又叫采样率、采样步长)
- 相机成像过程:通过镜头捕捉光线,光线通过透镜(放大镜)进入摄像机内部,击中感光元件(如:CMOS),转化成电信号,并经过处理转换为数字图像,最终显示在屏幕上,呈现出我们所看到的图像。
- 现实情况:由于电脑的内存限制,轴向Z的采样率往往低于横向XY,最终导致轴向各向异性(横向各向同性)。
1.3、点扩散函数(PSF)
点扩散函数(Point Spread Function,PSF) —— PSF可以是各向同性,也可以是各向异性,具体取决于成像系统和条件。
- PSF(x, z) = 6 x 6(此时,x 和 z 各向同性)
- PSF(x, z) = 6 x 3(此时,x 和 z 各向异性)
1.3.1、高斯函数 + 二维高斯PSF
高斯点扩散函数(PSF):描述光学系统中点源成像效应的一种常见模型。它基于高斯分布的数学形式,表示理想点源在成像过程中的扩散。
1.3.2、二维高斯PSF:代码生成

python">import numpy as np
import matplotlib.pyplot as pltdef gaussian_psf(sigma_x, sigma_z):_X, _Z = np.meshgrid(np.arange(-sigma_x, sigma_x+1), np.arange(-sigma_z, sigma_z+1), indexing='ij') # 生成坐标网格psf_array = np.exp(-(_X ** 2 / (2 * sigma_x ** 2)) - (_Z ** 2 / (2 * (sigma_z - sigma_x) ** 2))) # 二维的高斯分布函数# psf_array = 1 / (2 * np.pi * sigma_x * sigma_z) * np.exp(-(_X ** 2 / (2 * sigma_x ** 2)) - (_Z ** 2 / (2 * (sigma_z - sigma_x) ** 2))) # 二维的高斯分布函数return psf_array####################################
# 各向异性PSF : sigma_x != sigma_z
# 各向同性PSF : sigma_x == sigma_z
####################################
isotropic_psf = gaussian_psf(sigma_x=30, sigma_z=120) # 各向异性PSFplt.imshow(isotropic_psf)
plt.title('Gaussian PSF (anisotropic)')
plt.xlabel('Z Axis')
plt.ylabel('X Axis')
plt.colorbar(label='Intensity')
plt.show()一、Isotropic Reconstruction
论文地址:Isotropic Reconstruction of 3D Fluorescence Microscopy Images Using Convolutional Neural Networks
中文翻译:使用卷积神经网络对 3D 荧光显微镜图像进行各向同性重建
❤️ ❤️ 使用文档:https://csbdeep.bioimagecomputing.com/doc/index.html
❤️ github源码:https://github.com/CSBDeep/CSBDeep
❤️ 训练与测试数据下载:
(1)黑腹果蝇的轴向切片:Isotropic_Drosophila.tar.gz(3.3GB)
(2)发育中的斑马鱼眼睛的轴向切片(细胞核-洋红色标记,核膜-绿色标记):Isotropic_Retina.tar.gz(5.1GB)
(3)小鼠肝脏组织的轴向切片:Isotropic_Liver.tar.gz(889MB)
1.1、研究背景
1.1.1、当前现状 —— 荧光显微镜图像:(横向)各向同性 + (轴向)各向异性
在3D图像中:X轴(横向)、Y轴(纵向)、Z轴(轴向)
1.1.2、主要原因 —— Z轴各向异性 + Z轴采样率低
(1)由于PSF固有的轴向 z 伸长,导致沿该轴的像素分辨率降低2至4倍。
(2)由于显微镜采用机械逐平面采集方式,轴向采样减少了,导致整体的像素分辨率降低4至8倍。
1.1.3、解决方法 —— 基于CARE的各向同性重建
❤️ 荧光显微镜没有地面实况GT数据,导致深度学习无法应用。其本质上需要构建一个理想的、物理上不可能的显微镜。
- (母篇):CARE是一种数据生成策略,论文中分别展示了其在四个领域的应用效果。
- (子篇):Isotropic Reconstruction是CARE在各向同性重建的一种应用
- 其实现了将光学采集到的各向异性图像恢复为各向同性图像,而不需要额外的地面实况GT训练数据。
1.1.3.1、CARE:研究背景
荧光显微镜是生命科学中不可或缺的工具,用于研究细胞、组织和发育中生物体的时空动态。
- (1)光片显微镜、结构照明显微镜和超分辨率显微镜等,实现对细胞内的生物进行高分辨率成像。
- (2)图像质量受到多种因素的制约,如:光学设备的空间分辨率,时间分辨率、成像速度、成像深度、荧光团密度、漂白和光毒性。
- (3)制约因素无法同时优化,必须进行权衡。如:减少曝光时间来牺牲信噪比 (SNR),以提高成像速度。
- (4)权衡可以通过优化显微镜硬件来解决,但存在无法轻易克服的物理限制。因此,提高显微图像质量变得越来越重要。
- (5)图像恢复算法可以突破设计空间的的极限,恢复单独成像无法获得的重要生物信息。如:
超分辨率显微镜,反卷积,表面投影算法、去噪算法,且不同算法需要一定的使用条件。- (6)深度学习通过在大量经过充分注释的示例数据上,训练多层人工神经网络,可以完成特定数据的复杂任务。但是,缺乏足够的训练数据且不可能手动生成它们。
1.1.3.2、CARE:是一种数据生成策略
发表期刊:Nature Communications.2018
论文地址:Content-aware image restoration(CARE): pushing the limits of fluorescence microscopy
中文翻译:(内容感知图像恢复)突破荧光显微镜的极限
CARE(2个创新 + 4个应用):
(1)CARE核心:是一种数据生成策略 —— 通过XY面模拟Z面,得到Z面的GT数据(荧光图像没有Z面的GT数据)。
(2)CARE模型:提出了与 U‑net 拓扑架构类似的 IsoNet‑2 模型。
(3)CARE应用:基于上述两个创新,分别应用于四个领域。
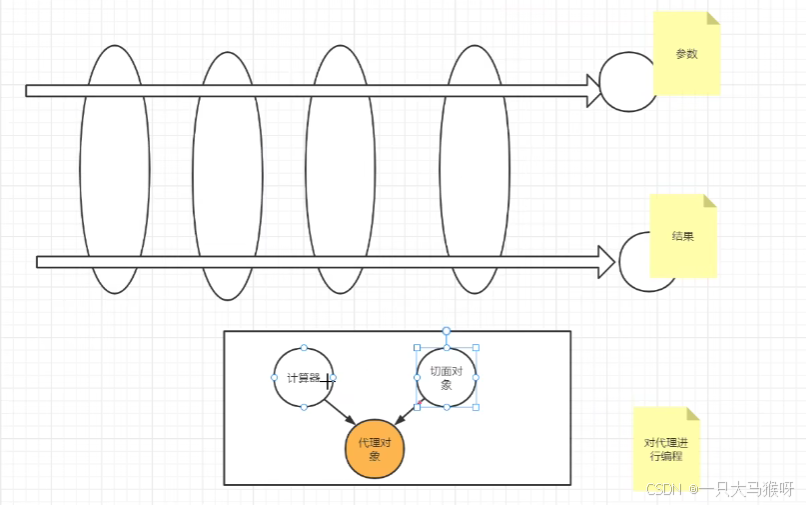
❤️ CARE(论文中示例 - 图解)
❤️ CSBDeep工具箱:一种用于显微镜图像恢复和分析的深度学习工具各向同性重建(Isotropic Reconstruction)
论文:《Isotropic reconstruction of 3D fluorescence microscopy images using convolutional neural networks》分割(DenoiSeg)
论文:《DenoiSeg: Joint Denoising and Segmentation》去噪(Noise2Void)
论文:《Noise2Void - Learning Denoising from Single Noisy Images》细胞检测(StarDist)
(官网)开发的所有图像恢复方法都提供了Python、FIJI和KNIME的免费开源软件。
1.1.3.3、最新文章
背景:基于 CARE 的各向同性重建需要一定的先验知识。如:根据光学分辨率计算输入图像(各向异性)的理论参数(各向同性):下采样 + PSF。
- CARE数据生成:通过PSF和下采样因子反向计算(模拟)各向异性的 XY 数据集
- cycleGAN:使用生成网络G和F替代CARE数据生成
采用循环一致生成对抗网络(cycleGAN)的无监督学习方法,通过估计模糊 PSF 并对其进行反卷积(自动计算而无需手动计算),实现各向同性重建。
发表期刊:Nature Communications.2022
论文地址:Deep learning enables reference-free isotropic super-resolution for volumetric fluorescence microscopy
中文翻译:深度学习为体积荧光显微镜提供无参考各向同性超分辨率
开源地址:Neuroclear
代码复现:未成功
发表期刊:Light: Science & Applications.2023(nature子刊)
论文地址:Deep self-learning enables fast, high-fidelity isotropic resolution restoration for volumetric fluorescence microscopy
中文翻译:Self-Net 管道以及使用合成和半合成数据验证其各向同性恢复性能。
开源地址:Self_Net
代码复现:没有细读
1.2、网络模型(IsoNet)
1.2.1、算法流程(原理)
a. 光学显微镜采集的 3D 图像
- 主要特征:在(横向 lateral) xy表现为各向同性,在(轴向 axial)xz / yz 表现为各向异性。
- 主要原因:
Z轴采样率低、PSF各向异性b. 提出了一种与 U‑net 拓扑架构类似的 IsoNet‑2 模型
(1)数据生成:提取(各向同性)
横向平面gxy = g(x, y),然后与PSF进行卷积运算 + Sσ横向下采样,最后构建(各向异性)横向平面pxy = p(x, y)
- PSF生成:
gaussian_anisotropic_psf(sigma_x, sigma_z)- 数据增强:
create_patches(raw_data, patch_size, n_patches_per_image, transforms)
- 对每个输入图像进行随机切割。裁剪数量 = n_patches_per_image;裁剪尺寸 = patch_size;PSF变换 = transforms;
- 最终得到图像对(gxyn ,pxyn)n∈N
(2)模型训练:采用PSNR (Peak Signal-to-Noise Ratio) 损失函数,对
图像对(gxy, pxy)进行逆映射学习(pxy -> gxy)与模型训练;
- 逆映射学习:通过可获得的(各向同性)横向平面pxy,模拟(各向异性)横向平面gxy,然后学习两者之间的映射关系;
(3)数据测试:对(具有模糊及下采样特性)(各向异性)
轴向平面pxz进行轴向上采样,并输入预训练模型,得到(各向同性)轴向平面~gxy(即gxz),使轴向平面 Z 恢复各向同性。
1.2.2、数据生成(图解)
1.2.3、网络架构
基于 2D U-Net 的各向同性重建(补充图13 、补充表3、补充注释2)
- 补充图13:详细的网络架构
- 补充表3:数据和训练的详细信息(深度学习的超参数)
- 补充注释2:对原理的解读,并没有介绍调参经验。
补充资料:补充图1-30、补充表1-3 和补充注释1-5
1.3、实验数据
1.3.1、效果展示
提出了两种不同的模型学习策略IsoNet-1和IsoNet-2
给出了两种不同的训练数据生成方法(full、split)
IsoNet-1:基于超分模型
- 网络架构:
C(64, 9, 9) - C(32, 5, 5) - C(1, 5, 5) - C(1, 1, 1)- 提出者《Learning a deep convolutional network for image super-resolution.》
IsoNet-2:基于分割模型
- 网络架构:
C(16, 7, 7) - M(2, 2) - C(32, 7, 7) - M(2, 2) - C(64, 7, 7) - U(2, 2) - C(16, 7, 7) - C(1, 1, 1)- 类似于《U-net: convolutional networks for biomedical image segmentation.》
荧光图像在多种模型下的结果比较:
行:(3D细胞核数据、3D膜数据、3D组合数据)的轴向切片
列:前三列
(1)GT(理论GT - 各向同性):其与各向同性PSF卷积来生成各向同性地面实图像
(2)GT isotropic(现实GT - 各向同性):微观数据的模糊图像。
(3)blurred input (对输入图像进行模糊处理):用于所有测试方法前六列(测试结果)
(4)deconv 反卷积(Richardson-Lucy,RL)
(5)SRCNN 超分辨率重建
(6 / 7)IsoNet-1分别使用完整(full) + 分割(split)的PSF
(8 / 9)IsoNet-2分别使用完整(full) + 分割(split)的PSF备注:full 得到的结果更像 GT 、split 得到的结果更像 GT isotropic
下图是源码中给定数据的测试结果:
1.3.2、不同PSF下的实验结果:高斯PSF + 共聚焦PSF + 光片PSF
PSNR (Peak Signal-to-Noise Ratio):是一种用于衡量图像或视频质量的指标。PSNR值越高,表示质量损失越小。
在3 种不同类型的数据集下,基于GT和模拟GT的PSNR计算结果。
- 数据集:
(Nuclei:原子核)、(Membranes:膜)、(Nuclei + Memb.:细胞核与细胞膜的合成图)
- 粗体值:表示PSNR表现最好
- 括号外:表示PSNR
- 括号内:表示标准差 (n = 10)
不同PSF类型的参数设置:
高斯PSF(Gaussian PSF)—— (σxy / σz = 2 / 8)
- 适用领域:生物荧光显微镜成像,尤其是在Z轴方向上具有一定的深度。
- 特点:光强呈现高斯分布。即中间最亮并逐渐向两侧减弱。
- σxy(横向 - 标准差): 控制高斯分布在 xy 平面上的宽度。
- σz(纵向 - 标准差): 控制高斯分布在 z 轴上的宽度。
共聚焦PSF(Confocal PSF)—— (NA = 1.1)
- 适用领域:共聚焦显微镜成像
- 特点:(1)共聚焦PSF主要在焦点处具有较小的主峰值,焦点附近有较小的侧翼。(2)具有较小的深度聚焦区域,对焦点附近的细节有较好的分辨率。
- 数值孔径(NA): 显微镜中物镜的参数。表示光学系统的光采集能力和分辨能力的量度。
光片PSF(light-sheet PSF)—— (NA detect = 0.8、NA illum = 0.1)
- 适用领域:光片显微镜成像
- 特点:具有较小的数值孔径,产生相对较宽的主要峰值。
- 检测系统的数值孔径(NA detect)
- 照明系统的数值孔径(NA illum)
1.3.3、设置参数:dowmSample + PSF
计算参数(软件设置参数 + 相机镜头参数)
dowmSample—— 必须大于等于1PSF—— 由于是逆映射学习,故PSF = X : Z
- 若设置的PSF参数,未能使得X轴与Z轴合并,则效果等同于超分(如:SRCNN)
- 若取消PSF,效果等同于超分(如:SRCNN)
1.3.4、经验结论
- 结论1:倍率越高,ISO各向同性重建效果越不明显。
- 原因如下:倍率越高,(横向)光学分辨率与(轴向)光学分辨率的比值越小,则PSF比值越小
- 结论2:ISO去噪效果非常好,但存在 " 微弱信号丢失 " 问题。
- 原因如下:在模型预测的图像前处理中,ISO采用自定义公式合并 u1 和 u2:
(负值置零)mean = lambda u1, u2: np.sqrt(np.maximum(mean1, 0) * np.maximum(mean2, 0))。该公式的核心是建立在percentile归一化上。
- 在percentile中,当pmin=2时,去噪效果非常好,但存在微弱信号丢失问题。
- 图像分为噪声(-0.06) +
微弱信号(-0.03)+ 正常信号(正值)- 在percentile中,当pmin=0时,解决了微弱信号丢失问题,但去噪效果同时消失。
- 图像分为噪声(-0.06) +
微弱信号(正值)+ 正常信号(正值)- 结论3:提取信号区域用于数据生成,提高训练数据集的质量。
- 问题定位:ISO采用随机裁剪,根据裁剪数量(n_patches_per_image) + 裁剪尺寸(patch_size)
- 解决方法:采用迭代裁剪(数量不固定),根据大津阈值获取背景值并用于计算图像信噪比,然后根据信噪比例(如:0.35)提取信号区域。
1.4、成像原理
1.4.1、理想的显微镜3D图像:g(x,y,z) -> f(x,y,z)
1.4.2、模拟的显微镜2D图像:g(x,y) -> p(x,y) -> g(x,z)
二、环境配置
2.1、创建虚拟环境
【Python虚拟环境】创建 + 激活 + 查看 + 退出 + 复制 + 删除
2.2、配置相关环境
python">pip install python=3.9
pip install matplotlib
pip install tifffile
pip install scipy
pip install tqdm
2.3、配置Tensorflow
2.3.1、配置Tensorflow(CPU)
ModuleNotFoundError: No module named 'tensorflow'
解决方案:pip install tensorflow
2.3.1、配置Tensorflow(GPU)
【深度学习环境配置】Anaconda +Pycharm + CUDA +cuDNN + Pytorch + Opencv(资源已上传)
解决方案:
pip install tensorflow_gpu-2.5.0-cp39-cp39-win_amd64.whl
报错提示:ModuleNotFoundError: No module named 'tensorflow'
2.3.1.1、ImportError: Matplotlib requires numpy>=1.20; you have 1.19.5
安装指定版本numpy(将自动卸载当前版本):
pip install numpy==1.22.0
备注1:虽然 iso 没有显示最高可支持的numpy版本,但若是numpy版本太高将报错提示。
- (numpy == 1.26.1)报错提示:
AttributeError: module 'numpy' has no attribute 'object'.- (numpy == 1.20.0)报错提示:
ImportError: Matplotlib requires numpy>=1.21; you have 1.20.0- (numpy == 1.22.0)没有问题(最终测试结果)
2.3.1.2、Debug:Could not locate zlibwapi.dll. Please make sure it is in your library path!
解决问题:Could not locate zlibwapi.dll. Please make sure it is in your library path!