TITLE:Environmental DNA reveals seasonal shifts andpotential interactions in a marine community
译名:环境DNA揭示了海洋群落的季节变化和潜在的相互作用
期刊:nature communications
日期:2020年1月
下载链接:
https://www.nature.com/articles/s41467-019-14105-1
一.简述
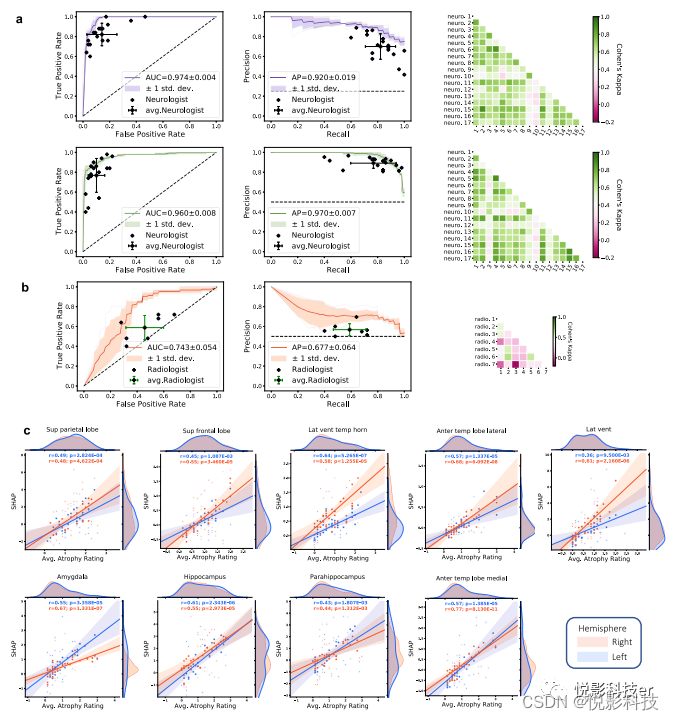
环境 DNA (eDNA) 分析允许同时检查多个营养水平和生命领域的生物,提供有关与生态系统变化相关的复杂生物相互作用的关键信息。在这里,我们使用 eDNA 的多位点扩增子测序来调查来自加利福尼亚州蒙特雷湾的 18 个月(2015-2016 年)时间序列海水样本的生物多样性。生成的数据集包含从微生物到哺乳动物的 663 个分类群(在科或更高分类等级)。我们推断了群落组成的变化,揭示了类群之间推定的相互作用,并确定了这些群落与环境特性之间的相关性。社区网络分析提供了所有生命领域中预期的捕食者-猎物关系、营养联系和季节性变化的证据。
二.结果概述
eDNA 方法的发展为了解海洋生物多样性提供了一个独特的视角,可以同时检查多个营养水平和生命领域的生物,从而提供有关与生态系统变化相关的复杂生物相互作用的关键信息。
海洋生物群可以有效地聚集成群落(子网络),这些群落的组成和丰度会随着时间的推移而波动。我们确定了六个子网络,它们代表不同的群落及其随时间的变化。春季/夏季类群(橙色、黄色、绿色子网络)之间丰富度的 eDNA 观察结果与秋季/冬季类群(灰色、蓝色、黑色)明显不同。所有子网都与环境变量相关,分类单元丰富度的季节性反映了与冬季较低的叶绿素 a 水平(灰色子网)和秋季较高的海面温度(蓝色子网)的相关性。
Balaenopteridae(座头鲸)和红螺菌科(变形菌)分别是蓝色和灰色子网络中连接最紧密的分类群。鉴于最相关的分类群对环境变量高度敏感,我们假设这些分类群可能代表不同生态系统状态的潜在指标,类似于 Banerjee 等人从微生物网络预测的关键物种。这些分类群可用作生态系统规模事件(如厄尔尼诺周期、上升流或人为干扰)后整个生物群落的转变的预警指标,这些事件很可能在较长时间内被 eDNA 检测到。
eDNA 条形码的效用,创建扩展了有据可查的捕食者-猎物相互作用的假设。假定的相互作用网络突出了分类群与其环境的波动,这可能是以前没有注意到的,因此可能是识别新生物和群落组合的有力方法,这些生物和群落组合可能表明环境变化和先锋保护策略。
三.部分结果展示
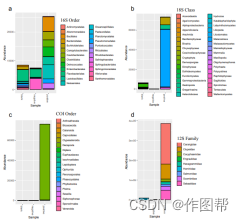
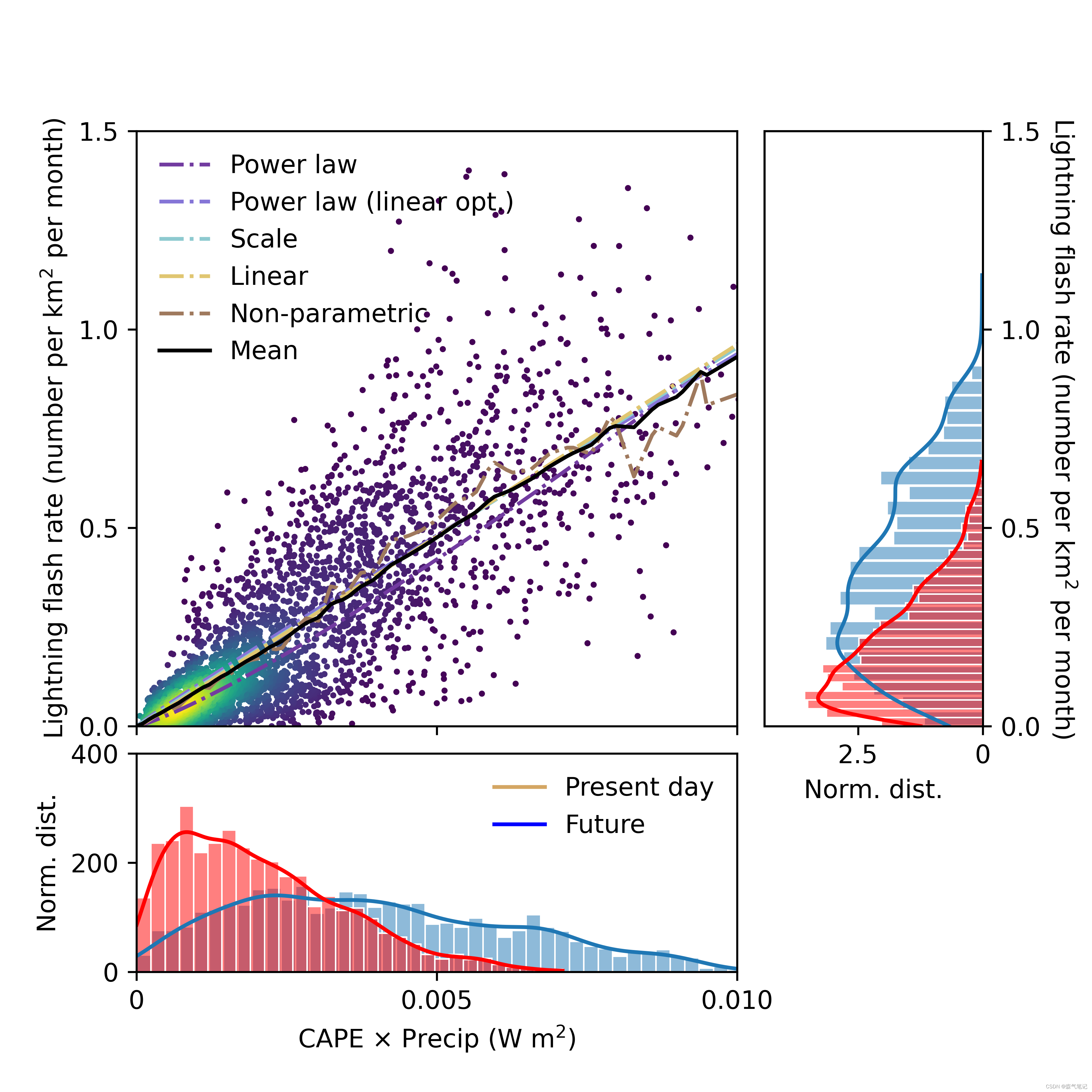
图1.空白、阴性和阳性对照。 在 16S (a)、18S (b)、COI © 和 12S (d) 的空白(DNA 提取和 PCR 空白)、阴性(无菌水)和阳性(人工群落)中检测到的分类群的序列计数。
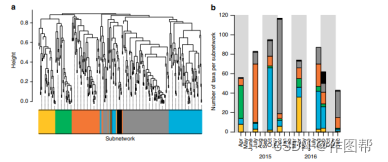
图2.与环境变量相关的 eDNA 数据的加权基因相关网络分析 (WGCNA)。a 对所有分类群的丰度指数变化进行聚类的树状图,颜色对应于不同的子网。b 从图 1a 中观察到每个子网络的分类群丰富度。
图3.子网与环境的相关性。所有子网络,都与本研究中所有测量的环境参数相关。蓝色(冬季丰富度最高)和灰色(秋季丰富度最高)是唯一分别与温度和叶绿素 a 显着相关的子网络。
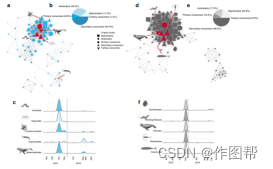
图4.随着时间的推移,选定分类群的网络可视化和扩增子指数丰度。2015 年和 2016 年秋季(蓝色)子网(a-c)和 2015 年 12 月(灰色)子网(d-f)的网络可视化。
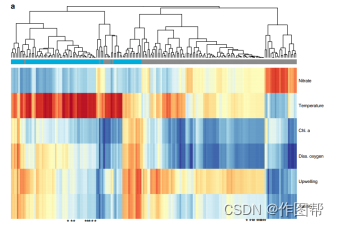
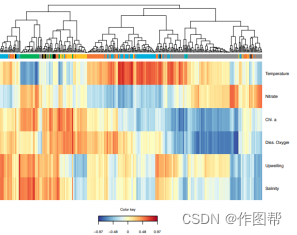
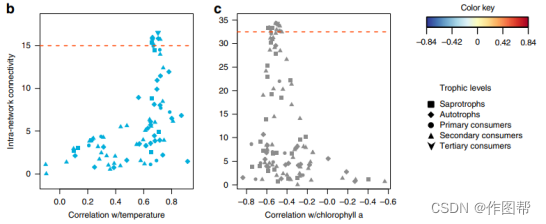
图5.分类单元和环境变量之间的相关性分析。分类单元和环境变量之间的偏最小二乘分析,以及分类单元与环境的关联性和相关性图。a.偏最小二乘分析图显示子网络与环境变量(行)中所有分类群(列)的相关性, 星号代表每个子网络中连接最多的前 10% 的分类群(在 (b) 和 © 中的红色水平线上方)。b, c 蓝色 (b) 和灰色 © 子网络中每个分类单元的连通性或中心性的相关性分别与温度和叶绿素 a 相关。
END