近几天看到论文里面有T分布随机近邻嵌入(T-distributed stochastic neighbor embedding, T-SNE)这种可视化方法,以前好像也看到过,但没有系统了解过,现有时间正好实践记录一下。
1. T-SNE简介
T-SNE是一种降维方法,降维?PCA(Principal component analysis)也可以降维,T-SNE有什么特点呢?T-SNE是非线性的,而PCA是线性的。T-SNE非线性降维的思路是将高维特征投射到低维空间,使得原本在高维空间距离较远的数据点在低维空间同样距离较远,而原本在高维空间距离较近的数据点在低维空间同样距离较近。低维空间一般设为2维或者3维,方便可视化。
然而,T-SNE很少用于降维,论文中看到的基本是用来可视化的,因为一般需要降维的数据都具有线性相关性,故PCA用的比较多。T-SNE可视化原始数据的分布,度量原始数据信息的相关性;T-SNE可视化神经网络提取的特征,评估不同模型的优劣性。本质上,近邻嵌入寻找保留了样本邻居关系在新维度上的较低数据表示。
T-SNE理论详解
2. T-SNE的Python实践
Python中实现T-SNE采用sklearn.manifold模块中的TSNE类。
TSNE类有几个重要参数:
(1)n_components:嵌入空间的尺寸,即降维维度,其默认值为2。
(2)perplexity:困惑度,与流形学习算法中使用的最近邻的数量有关,通常情况下,大数据集需要大困惑度,考虑选择5到50之间的值,不同的值可能导致显著不同的结果,perplexity的默认值为30。
(3)learning_rate:T-SNE的学习率通常在[10.0,1000.0]范围内。如果学习率太高,数据可能看起来像一个“球”,任何一点与它最近的邻居的距离都差不多;如果学习率太低,大多数点可能看起来被压缩在密集的云中,几乎没有离群值;如果代价函数陷入一个糟糕的局部最小值,增加学习率可能会有所帮助,learning_rate的默认值为200。
(4)n_iter:优化的最大迭代次数,至少应该是250,其默认值为1000。
用鸢尾花数据来试试T-SNE:设置降维到2
import matplotlib.pyplot as plt
from mpl_toolkits.mplot3d import Axes3D
from sklearn.manifold import TSNE
from sklearn.datasets import load_irisiris = load_iris()
X = iris.data
y = iris.targettsne = TSNE(n_components=2, perplexity=15).fit_transform(X)
aa = tsne[:, 0]
bb = tsne[:, 1]color = ['limegreen', 'cornflowerblue', 'orange']plt.figure(dpi=300)
for i in range(tsne.shape[0]):plt.scatter(aa[i], bb[i], facecolor=color[y[i]], alpha=0.7)
plt.savefig('./tsne_2.jpg')
plt.show()
输出为:
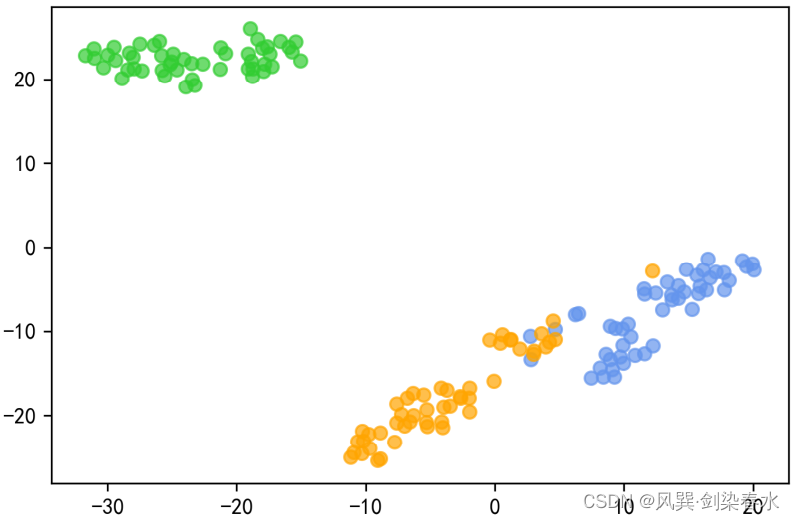
值得注意的是:T-SNE每次运行会得到不同的可视化图像,但不同类别之间的距离是相似的
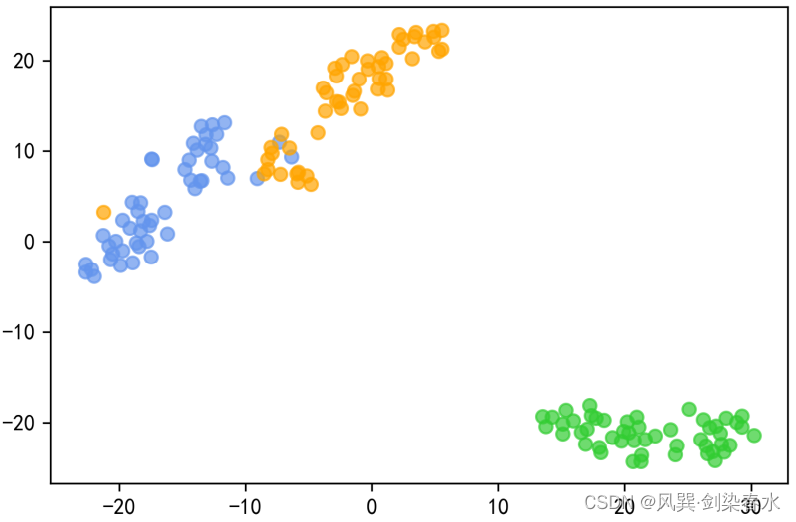
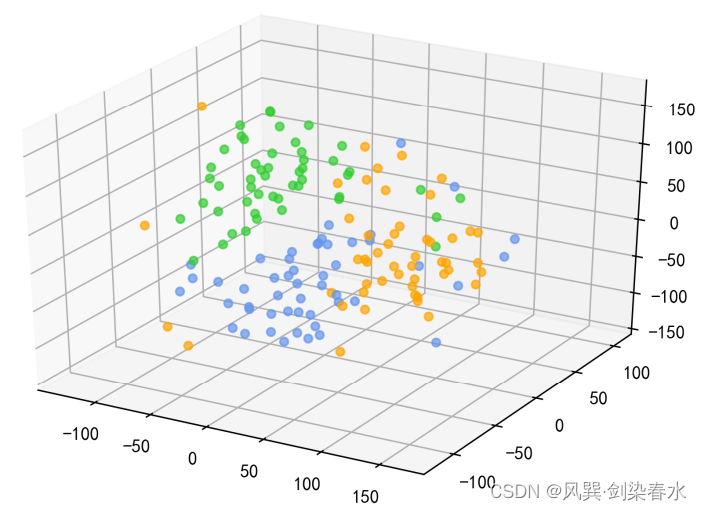
同样的,降低到3维也可以进行可视化
import matplotlib.pyplot as plt
from mpl_toolkits.mplot3d import Axes3D
from sklearn.manifold import TSNE
from sklearn.datasets import load_irisiris = load_iris()
X = iris.data
y = iris.targettsne = TSNE(n_components=3, perplexity=15).fit_transform(X)
aa = tsne[:, 0]
bb = tsne[:, 1]
cc = tsne[:, 2]color = ['limegreen', 'cornflowerblue', 'orange']fig = plt.figure(dpi=300)
ax = Axes3D(fig)
for i in range(tsne.shape[0]):ax.scatter(aa[i], bb[i], cc[i], facecolor=color[y[i]], alpha=0.7)
plt.show()