文章目录
- 一、介绍
- 二、原理及步骤
- 三、三维基因组检测技术比较
- 1、C技术
- 3C(一对一)
- 4C(一对多)
- 5C(多对多)
- Hi-C(全部互作)
- 2、基于免疫沉淀技术
- ChIP-loop
- ChIA-PET
- 四、总结
一、介绍
Hi-C 技术源于基因组捕获技术(Chromosome conformation capture,3C),用于分析染色质三维空间结构的一种测序方法。1
关于什么是三维基因组,可以参考:一文读懂三维基因组
用途:
- 量化在三维空间中基因组的染色质间交联(cross-linked chromatin)
- 解析全基因组互作模式,如启动子和增强子互作
- 构建三维空间结构模型,如研究基因组三维结构特征:compartment,TAD,loop等
- 构建全基因组互作图谱
- 辅助提升基因组组装
- 构建基因组单体型图谱
二、原理及步骤
1、甲醛固定
先加入甲醛将基因组中参与染色质互作作用的蛋白质凝固
一般将活体样本在室温用 1-3%的甲醛处理 10-30min,但是此步骤会减少限制内切酶对DNA序列的消化效率,需要严格控制。2
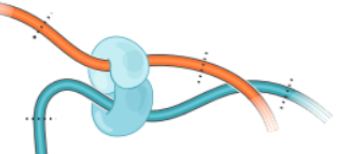
2、酶切序列
用限制性内切酶切割基因组,打断后的片段大小会影响测序分辨率,一般有两种酶可供选择:6bp 的限制性内切酶,4bp 的限制性内切酶。后者具有更高的分辨率。EcoR1 或 HindIII 用于每4000bp切割一次基因组,在人类基因组中产生约100万个片段。2
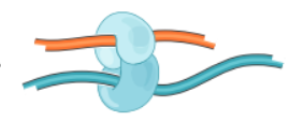
3、末端修复
得到的片段具有平末端或粘性末端,然后将末端补平修复,加入生物素。
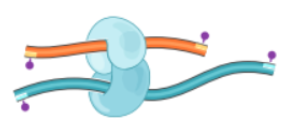
4、连接及解交联
使用 T4 DNA连接酶连接互作片段,形成环状。将连接DNA片段的蛋白质消化掉,得到交联片段。
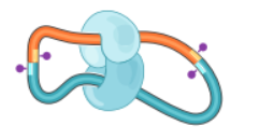
5、序列打断
使用超声波或其他方式,再次打断片段。
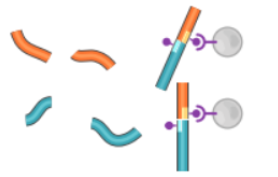
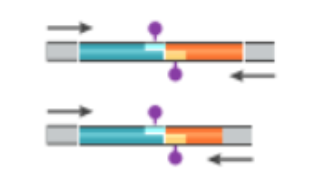
6、上机测序
用磁珠将带生物素的捕获,制作文库,上机测序。
三、三维基因组检测技术比较
下图展示了目前主流三维基因组测序技术3:
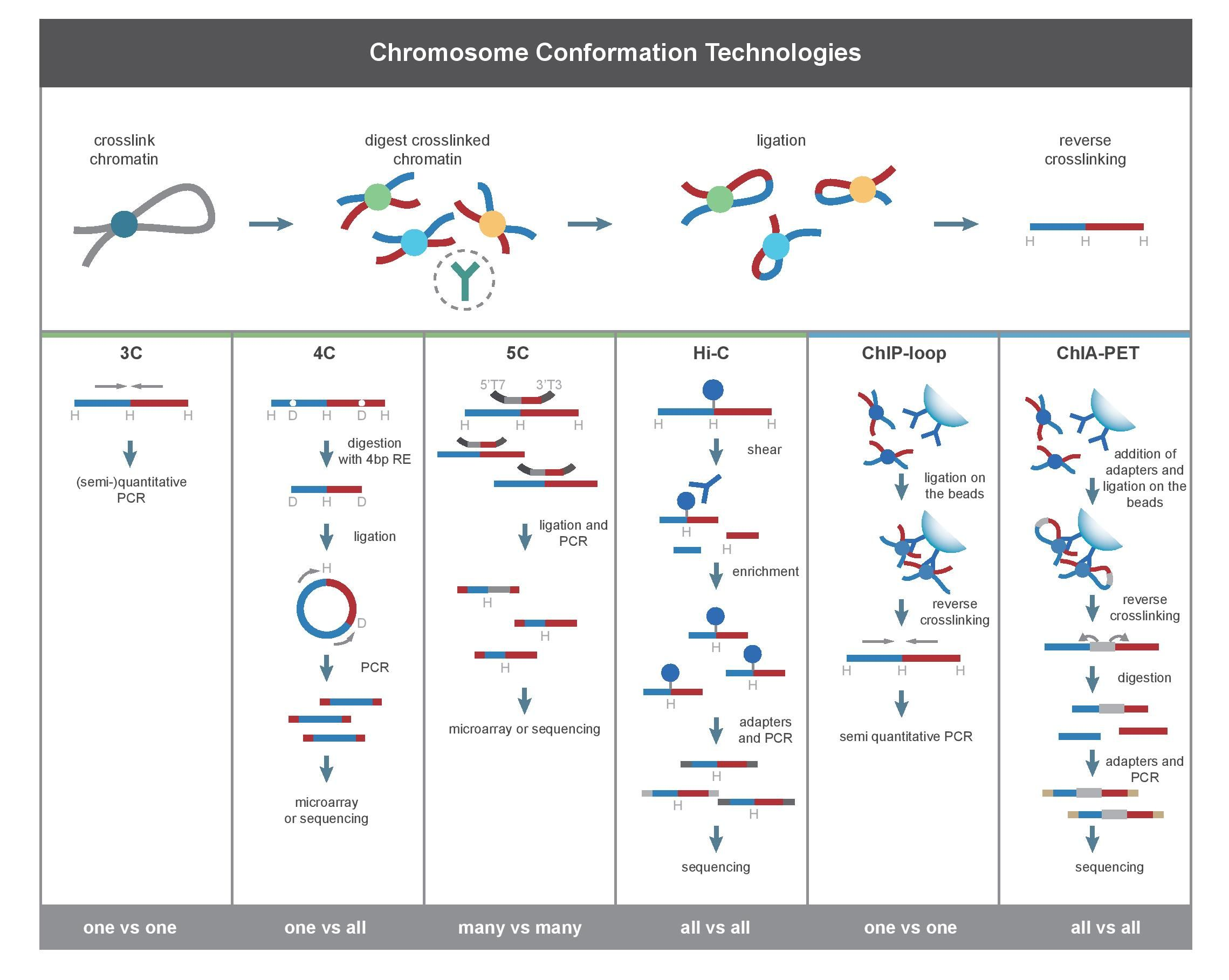
1、C技术
3C(一对一)
基因组捕获技术(Chromosome conformation capture,3C)是最早研究三维基因组的技术,需要提前知道互相作用区域,才能量化一对基因组基因座之间的互相作用。4
4C(一对多)
染色体构象捕获芯片(Chromosome conformation capture-on-chip,4C ),可以捕获一个基因区域其他区域间的互相作用。该技术不需要知道作用区域的先验知识就可以使用。5
5C(多对多)
染色体构象捕获碳拷贝(Chromosome conformation capture carbon copy,5C) ,可以检测某段区域内所有的互作,但是该区域一般<1 Mb。覆盖度的问题也就造成该技术不适用于全基因组测序。6
Hi-C(全部互作)
高通量基因组捕获技术,基本解决了上述技术的缺点,可以实现全基因组覆盖检测全部未知互作区域。
2、基于免疫沉淀技术
ChIP-loop
该技术将 3C 与 ChIP-seq 结合,可以检测目的蛋白质介导的两个目的基因区域互作。7
ChIA-PET
该技术将 HiC 与 ChIP-seq 结合,可以检测目的蛋白质的所有互相作用.8
四、总结
| 互作 | 覆盖 | 作用 | |
|---|---|---|---|
| 3C | 单对单 | < 1 Mb | 检测已知基因组基因区域之间的互相作用 |
| 4C | 一对多 | 全基因组 | 确定某基因组区域与其他区域的互相作用 |
| 5C | 多对多 | < 1Mb | 确定染色体特定区域内的全部高级结构 |
| Hi-C | 全部互作 | 全基因组 | 检测全基因组范围内的全部高级结构 |
| ChIP-loop | 一对一 | < 1 Mb | 检测目的蛋白质介导的两个目的基因区域互作 |
| ChIA-PET | 全部互作 | 全基因组 | 检测目的蛋白质的所有互相作用 |
de Wit E, de Laat W (January 2012). “A decade of 3C technologies: insights into nuclear organization” ↩︎
Naumova N, Smith EM, Zhan Y, Dekker J (November 2012). Analysis of long-range chromatin interactions using Chromosome Conformation Capture ↩︎ ↩︎
Li, G., Cai, L., Chang, H., Hong, P., Zhou, Q., Kulakova, E. V, … Ruan, Y. (2014). Chromatin Interaction Analysis with Paired-End Tag (ChIA-PET) sequencing technology and application. ↩︎
Dekker J, Rippe K, Dekker M, Kleckner N (February 2002). “Capturing chromosome conformation”. Science. ↩︎
Zhao, Zhihu. et al. (2006). “Circular chromosome conformation capture (4C) uncovers extensive networks of epigenetically regulated intra- and interchromosomal interactions”. Nature Genetics. ↩︎
Dostie J, Richmond TA, Arnaout RA, Selzer RR, Lee WL, Honan TA, et al. (October 2006). “Chromosome Conformation Capture Carbon Copy (5C): a massively parallel solution for mapping interactions between genomic elements”. Genome Research. ↩︎
Horike S, Cai S, Miyano M, Cheng JF, Kohwi-Shigematsu T (January 2005). “Loss of silent-chromatin looping and impaired imprinting of DLX5 in Rett syndrome”. Nature Genetics. ↩︎
Fullwood MJ, Liu MH, Pan YF, Liu J, Xu H, Mohamed YB, et al. (November 2009). “An oestrogen-receptor-alpha-bound human chromatin interactome”]. Nature. ↩︎


![BUU[SCTF2019]Who is he](https://img-blog.csdnimg.cn/a03cd67b6e9a40b4a8183cbc3d6a199e.png?x-oss-process=image/watermark,type_d3F5LXplbmhlaQ,shadow_50,text_Q1NETiBAMWVucw==,size_20,color_FFFFFF,t_70,g_se,x_16)