胞嘧啶甲基化是DNA中广泛存在的一种修饰,发挥重要的作用。在酵母新型隐球菌(Cryptococcus neoformans),CG甲基化发生在富含转座子的重复序列中,而且需要DNA甲基转移酶Dnmt5。科学家发现Dnmt5在体内和体外都表现出精确的甲基化维持特异性,并且在体内显示出与后生动物维持甲基化的Dnmt1酶利用相似的辅酶因子。值得注意的是,系统发育和功能研究显示,祖先物种在50 到150 百万年前丢失了重头甲基化酶DnmtX基因。文中作者分析了自远古DnmtX丢失以来甲基化是如何维持的。实验和比较研究揭示了在酵母新型隐球菌甲基化模式如何得以有效复制,复制过程中极少出现甲基化的随机缺失和获得,以及自然选择的作用。作者认为表观遗传通过一种类似于达尔文基因组进化的方式,已经被繁殖了不少于5000万年。
DNA胞嘧啶五碳甲基化(5mC)存在于各种生命体。可能因为它的基因组防御重要功能,所有已知的脊椎动物和陆生植物DNA都有这种修饰。5mC初始因各种重头甲基化转移酶(DNMTs)作用于非甲基化胞嘧啶而产生。在回文序列,如CG和CHG中,两条链的胞嘧啶都被甲基化。这种初始修饰形态在DNA复制过程中被维持甲基化酶“记住”,并对复制产生的半甲基化链发挥功能。哺乳动物DNA甲基化系统就是一个类似分工的例子。Dnmt 3a/b和Dnmt3L复合物行使重头甲基化功能,一但DNA甲基化在生殖细胞和胚胎发育过程中建立,Dnmt1酶将联合UHRF1进行维持。UHRF1可以识别H3K9me和半甲基化5mC的蛋白质。这种重头甲基化和维持甲基化的分工模式同样存在于陆生植物,尽管Dnmt3在被子植物中丢失且被其它酶替代。DNA甲基化对发育至关重要,在转座因子沉默、染色体稳定性、单等位基因表达和基因沉默等方面发挥关键作用。此外,Dnmt3a是急性髓性白血病常见的突变驱动基因,Dnmt3b突变导致常染色体隐性遗传病免疫缺陷、着丝粒区不稳定、面部异常等。
虽然后鞭毛生物基因组几乎都至少编码两种甲基化转移酶同系物,但个别物种只编码一个,人类病原真菌新型隐球菌就是其一,它是一种担子酵母菌类,在转座子富集的着丝粒和亚着丝粒区域具有对称的CG甲基化。该甲基化修饰依赖于DMT5基因编码的胞嘧啶甲基转移酶Dnmt5,该基因为新型隐球菌感染适应性所必须。Dnmt5蛋白家族的特征是具有一个N-末端染色质域(CD),其后依次为胞嘧啶甲基转移酶催化域、环指结构和SNF2型腺苷三磷酸酶(ATPase)相关的域组成(图1A)。这种推测甲基化转移酶在真菌和绿藻中广泛存在,其中一些物种CG甲基化已被证实可影响核小体定位。5mC在新型隐球菌中聚集在富含转座元件的重复序列上,这可能与真菌类隐球菌Cryptococcus deuterogattii发挥类似的转座子沉默功能。DMT5基因失活突变伴随Cryptococcus deuterogattii的5mC丢失。以下为作者探索Cryptococcus neoformans菌Dnmt5描述。
1.新型隐球菌5mC促进因子
已报到在新型隐球菌中,5mC修饰区域同时伴随H3K9me修饰。由此,作者通过将已纯化的含有CD结构域的片段与商业化集成人不同组蛋白修饰芯片结合,测试Dnmt5的CD结构域能否识别这一组蛋白修饰标记。结果H3K9me和H3K27me修饰肽均产生信号。然而,由于C. neoformans与人组蛋白H3的27位赖氨酸周围序列差异,作者进一步通过荧光偏振评估了染色质域与C. neoformans组蛋白H3序列的结合能力(图1B)。H3K9me的结合强度比H3K27me结合强度大幅增强显示H3K9me为CD的主要结合配体。
为进一步评估H3K9me是否影响5mC,作者敲除C. neoformans中H3K9仅有的甲基转移酶基因Clr4,用甲基化敏感酶(HpyCH4IV;AˆCGT)消化基因组DNA,然后Southern杂交测定5mC。基于已发表的野生型新型隐球菌5mC图谱,作者设计了一个唯一识别着丝粒13的特异性探针(probe U)(图1C)。此外,还设计了另一个识别重复着丝粒序列的探针(probe R),实现一次性分析多个着丝粒5mC位点的分布。在探针检测区域,clr4敲除(clr4Δ)菌株的5mC水平显著降低,而dmt5敲除(dmt5Δ)菌株或DNMT催化结构域或ATPase结构域催化残基突变的菌株没有产生5mC修饰(图1D)。染色质免疫沉淀测序(ChIP-seq)分析FLAG-Dnmt5发现其募集到H3K9me结构域显著减少。相反,Dnmt5染色质域的一个废除其结合H3K9me(W87A,Y90A)的突变对5mC的影响很小,这表明存在其它H3K9me依赖性因子影响5mC水平(图1E)。异染色质蛋白1(HP1)家族包括多种保守蛋白,可通过与H3K9me结合,参与异染色质形成,并促使5mC甲基化。在C. neoformans中,作者根据裂殖酵母同源基因特征,鉴定了一个HP1种间同源基因,命名为Swi6。虽然swi6敲除对5mC影响较轻,但swi6 敲除和dmt5-W87A,Y90A共突变比clr4敲除降低了5mC的水平(图1E)。免疫沉淀结合质谱和免疫共沉淀检测显示Swi6和Dnmt5物理结合,表明H3K9me通过Dnmt5的CD结构域和HP1蛋白两种方式招募Dnmt5促使5mC形成。
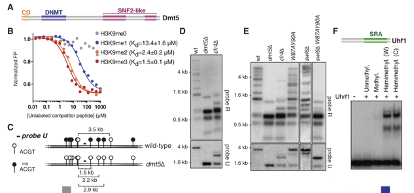
图1 A-F
缺失H3K9me的细胞仍残留5mC让作者进一步探索DNA甲基化的其它相关因子。C. neoformans具有一种类似于Uhrf1的蛋白质,该蛋白质包含SRA结构域(图1F),但缺失人种间同源基因的H3K9me读取Tudor结构域和RING E3连接酶结构域。运用非变性凝胶电泳,作者检测了C. neoformans重组Uhrf1与6个CG位点分别进行无甲基化、半甲基化和对称甲基化标记的DNA结合的能力。结果显示Uhrf1选择性地结合半甲基化DNA(图1F)。Uhrf1(uhf1敲除)缺失对体内5mC的影响几乎检测不到,但当与Clr4共缺失(clr4敲除加uhf1敲除)时,捕获的5mC分布略有改变(图1G)。
为了在全基因组范围内分析这些基因型,作者通过全基因组甲基化测序检测了野生型、dmt5敲除、clr4敲除、uhf1敲除和clr4及uhf1共敲除菌株DNA甲基化分布(图1H)。对比clr4缺失,clr4和uhf1双缺失菌株DNA的CG二核苷酸对称甲基化从31%减少到11%(图1I和1J)。分析表明,Uhrf1和Clr4通过两条平行的途径促使了5mC的形成(图1J)。
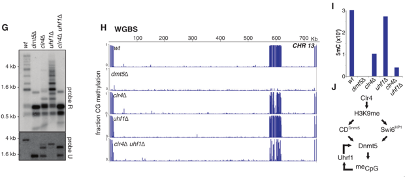
图1 G-J
2. 纯化的Dnmt5显示出精确的维持甲基化的特异性
在C. neoformans中,Dnmt5与Uhrf1同系物行使功能,并可以独自识别H3K9me,这种特征让作者联想到哺乳动物甲基化系统的维持。作者因此检测Dnmt5是否具有维持酶期望的底物特异性。通过酿酒酵母表达并纯化全长Dnmt5。通过S-[3H-甲基]-腺苷甲硫氨酸供体和合成的20bp双链DNA寡核苷酸底物进行甲基转移酶分析,这些底物含有6个CG位点,进行未甲基化,半甲基化,或者所有CG位点对称甲基化修饰。与缺乏Dnmt5的对照相比,在ATP缺失情况下,Dnmt5对任何底物未显示活性(图2A)。然而,在ATP存在条件下,与人Dnmt1催化速率相当,Dnmt5仅对半甲基化底物表现活性(图2A和2B)。此外,不管胞嘧啶甲基化状态如何,Dnmt5对CHG序列未表现出像对CG序列的活性,表明Dnmt5酶的CG二核苷酸特异性。
由于Dnmt5染色质域识别H3K9me,作者通过体外引入H3K9Me3肽分析是否存在重头甲基化活性。在甲基化或无甲基化修饰H3K9肽的形况下,半甲基化底物的比率相当,且对非甲基化DNA底物没有影响(图2C和2D)。作者进一步测试是否重组Uhrf1和Swi6可以触发重头甲基化活性,但并未发现。作者因此得出结论,Dnmt5是一种在体外作用于半甲基化底物的维持酶,对非甲基化底物没有活性。

图2
3. 5mC在机体内通过维持机制而繁殖
尽管Dnmt5在体外显示了维持酶的底物专一性,但在体内它与其它因子共同作用可能改变其性质。分析这个问题的一个方法是从细胞中去除Dnmt5,让甲基化丢失,然后重新引入这个酶。如果Dnmt5在细胞中起维持酶的作用,那么DNA甲基化将不能在基因组范围内被重新恢复,因为它的半甲基化底物已经丢失。
作者首先构建了半乳糖诱导启动子控制Dnmt5基因表达,pGAL-DMT5(图3A),该等位基因N末端进行2XFLAG标签表位标记。通过对葡萄糖抑制的20(I20)或45(I45)代筛选菌株诱导表达Dnmt5蛋白,作者通过Southern杂交未检测到5mC,而构建的Dnmt5表达显著(图3A)。为了评估pGAL-DMT5等位基因是否起作用,靶向构建物进而转化野生细胞,并运用诱导pGAL7进行筛选(图3B)。诱导条件下I0代菌株与野生型菌株的5mC无可辨差异,表明可诱导的标记的等位基因是发挥功能的。随后,抑制pGAL-DMT5,5mC丢失。进一步再次诱导40(I40)或90(I90)代菌株的Dnmt5,未能检测到5mC。为确认细胞仅部分去除5mC后,如通过核形成传播机制(nucleation-spread),甲基化修饰能否被恢复,作者建立了抑制pGAL-DMT5有限代数后(R)再诱导(I)的时序实验。在抑制条件下,25代后5mC大幅降低。然而,当转移到含半乳糖诱导培养基后,作者未观察到5mC的恢复(图3C和3D)。

图3
作者进一步运用不同方法进行了类似的分析。首先敲除部分DMT5基因,再重新引入缺失DNA序列(图4),重新引入的等位基因称为RI-DMT5。尽管RI-DMT5的表达与野生型相当,但Southern杂交检测不到5mC(图4A)。Dmt5敲除菌株全基因组甲基化测序显示所有CG位点无甲基化修饰(图4B)。对两株RI-dmt5菌株测序发现,除了两个位点外,5mC在全基因组范围缺失(图4B)。进一步通过液相色谱-质谱对从野生型、dmt5Δ和RI-DMT5基因组DNA分析,结果显示野生型gDNA中5mC(5mC/dG)为0.33%,dmt5Δ未检测到, RI-DMT5菌株为痕量(可检测但低于量化限值)(图4C)。这种痕量活性表明,Dnmt5对未修饰DNA的活性可能非常低,并来源于维持酶活性的记忆。
H3K9me组蛋白修饰在dmt5Δ和RI-DMT5菌株仍然全基因组分布,尽管与野生型相比分布有所改变,且在子端粒区域信号变强(图4D)。此外,ChIP-seq测序证明RI-Dnmt5蛋白仍旧定位在异染色质区域(图4E)。
为了明确RI-DMT5是否能够有效地维持DNA甲基化,一种着丝粒13被耐药标记(CEN13::natR)的野生型菌株,其与RI-DMT5菌株杂交(图4F)。从杂交后代筛选出具有RI-DMT5等位基因和着丝粒标记的减数分裂后代。CEN13特异性探针进行Southern杂交检测其5mC。所有子代都呈现野生型5mC模式(图4F)表明RI-DMT5等位基因可在存在甲基化底物条件下发挥功能。
许多物种需要一种或多种重头甲基化酶通过有性生殖建立5mC。有研究称,串联重复URA5标记的转基因在C. neoformans交配和减数分裂/产孢后,因RNA干扰而非DNA甲基化形成可遗传性沉默。尽管如此,为了确定非甲基化着丝粒经有性生殖能否发生甲基化修饰,作者通过两种遗传杂交分析减数分裂后代。在对照组,将CEN13::natR菌株与dmt5D菌株杂交,运用CEN13特异探针和Southern杂交分析同时包含CEN13标记和野生型DMT5等位基因的三个子代。如预期的那样,子代甲基化在5mC修饰的着丝粒上得以维持(图4G)。在实验组,将野生型菌株与含有dmt5Δ等位基因缺失且同时携带CEN13::natR的菌株杂交,同样分析了表达野生型DMT5等位基因和标记着丝粒的三个子代,它们是以非甲基化状态进入杂交的。结果显示子代仍然没有发生甲基化修饰,这表明Dnmt5能够维持甲基化,但是不能在完全失去甲基化的着丝粒上重新建立(图4H)。因此,有性生殖并不能有效恢复5mC。最后,作者将细胞置于多种应激条件下,确定RI-DMT5菌株能否重建甲基化,结果并未发现。


图4
为评估Dnmt5甲基化维持特异性是否依赖于内源序列特性,作者通过HpaII甲基转移酶(产生对称的CmCGG)体外构建了一个富含HpaII位点的DNA甲基化片段,并将其整合到C. neoformans着丝粒4右侧边界的基因组中,以使其接近H3K9me修饰的染色质(图5A-5C)。转化株DNA经5mC敏感性HpaII限制性内切酶消化,通过酶切位点两侧设计引物,qPCR分析甲基化相对水平(图5D)。结果显示除了CG二核苷酸特异性外,体内似乎没有维持甲基化的序列特异性要求,这与生化结果一致。

图5 A-D
接下来作者通过体外甲基化位点作为成核位点(nucleation sites)来检测5mC是否能在体内扩展。运用5mC敏感酶BstUI(CGCG)和Hpy99I(CGWCG)切割基因组DNA,并对这些位点进行qPCR分析。结果未观察到高于背景以上的任何信号,这表明5mC修饰未扩散到临近的CG二核苷酸上(图5E)。体外观察到类似的结果:作者将纯化的Dnmt5与不含CG位点、两个未甲基化CG位点或两个未甲基化CG位点且伴有一个对称甲基化CG位点的双链DNA底物孵育(图5F)。虽然在与细菌DMT M.SssI孵育的对照产物检测到甲基化产物,但与Dnmt5孵育的产物没有检测到甲基化信号,这表明即使存在临近甲基化位点,该酶也无重头甲基化活性(图5F)。
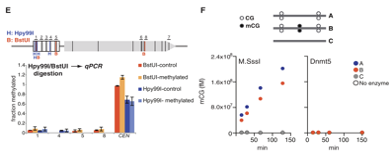
图5 E-F
4. 祖先物种重头甲基化酶基因丢失
鉴于Dnmt5是C. neoformans基因组预测的唯一的甲基转移酶,并且似乎起甲基化维持作用,那5mC是如何建立的?为了探索这一问题,作者研究C. neoformans属生物家族——银耳科(Tremellaceae)基因组。与C. neoformans基因组密切相关的物种包括人类致病菌Cryptococcus gattii和非致病菌Cryptococcus amylolentus和Cryptococcus wingfieldii,仅含一个预测的与Dnmt5是同源的DNA甲基转移酶(图6A)。C. deuterogattii所有转座子失活并失去RNAi和5mC。另一个最接近的物种,Cryptococcus depauperatus,缺乏预测的DNA甲基转移酶,同样暗含DMT5在其进化过程中丢失(图6A)。值得注意的是,较远物种同时编码Dnmt5和一种未知的预测性DNA甲基转移酶,称为DnmtX(基因座位:DMX1)(图6A)。该预测蛋白包含一个溴相关同源(BAH)域和一个Dnmt催化结构域。系统发育模式显示DMX1基因在C. neoformans和C. depauperatus的共同祖先中丢失,并且C. depauperatus后续同时丢失了DMT5基因。由于Dnmt5是一种维持性甲基化酶,作者提出DnmtX是一种重头甲基转移酶的设想。
作者分别从Kwoniella mangroviensis, Kwoniella bestiolae和Kwoniella pini中克隆DnmtX基因进行假设验证。克隆基因分别插入pGAL7半乳糖诱导启动子且N末端进行血球凝集素表位标签标记。随后将其转基因到C. neoformans菌株,且C. neoformans菌株先前通过基因改造,包括破坏Dnmt5基因,然后通过FLAG标记的等位基因(RI-DMT5)修复。之所以选用该技术是基于作者认为DnmtX酶可能难以有效生成5mC,特别是重头甲基化相关的重要辅酶因子没有被引入。
因为所预测用DnmtX酶建立5mC可能是低效的,特别是在没有引入对新甲基化重要的辅助因子的情况下。
含有半乳糖的培养基诱导每个菌株表达DnmtX,然后提取DNA。采用甲基化DNA免疫沉淀测序(MeDIP-seq)和全基因组亚硫酸盐测序(WGBS)两种技术进行全基因组5mC水平检测。两种检测技术在三种菌株中都观察到广泛的积累了5mC,主要集中在着丝粒区域(图6B)。这些数据表明,三种DnmtXs在体内都为重头甲基化转移酶,证明了祖先DnmtX也具有这种功能。

图6 A-B
5. 实验演化揭示维持5mC的保守性
祖先DnmtX基因丢失发生在C. neoformans和C. depauperatus分化之前,Tremella mesenterica和 Kwoniella heveanensis分化之后(图6A)。C. neoformans和C. gattii分化时间预计在34至49 百万年前。鉴于系统发育关系,C. neoformans和C. depauperatus的分化时间要更为远古。C. neoformans和T. mesenterica的共同祖先预计生活在153百万年前。因此,DMX1基因可能在150到50百万年前期间丢失。综上所述,这些数据表明,Dnmt5在相当长的时间内维持了5mC,致使致病性Cryptococcus种群的形成。
为了解5mC是如何自DnmtX远古消失以来进行持续的,作者对C. neoformans进行演化实验。野生型(第0代或g0代)单菌落在充足的液体培养基中繁殖120代,无菌落瓶颈。培养液稀释后涂布琼脂糖平板,形成单菌落。挑取两个独立克隆,同时对其亲代分别进行全基因组亚硫酸盐测序,进行重复性实验(图6C)。对比亲代分别检测到50个和38个甲基化位点丢失(图6D和图6E),表明99.0%和99.2%的5mC位点在120代得以维持。两个克隆共同丢失的甲基化位点只有两个,应该为偶然发生的重叠(图6E)。假设所有位点同等维持甲基化信息,并为一阶动力学,那么单个5mC位点每一代的丢失率为 9.3 X 10-5。与重复性实验一致,作者在每个实验中观察到新增5个甲基化位点(图6E)。通过未甲基化位点评估,每一代甲基化位点实际获得的概率为4.5*10-6。

图6 C-E
6. 进化分析揭示5mC甲基化图谱的保守型
作者首先试图比对具有四个明显分化枝,VNI、VNII、VNBI和VNBII,共同享有约4.7百万年前祖先的分化菌的着丝粒区域(图7A)。由于甲基化富集区着丝粒含有包括gypsy 和 copia 的逆转录转座子Tcn1-Tcn6 六个家族,数百个菌株的短读长测序数据仍无法组装。进一步作者通过牛津Nanopore(ONT)对四个分化枝的各两个分离株进行几乎全长测序(图7B)。着丝粒点阵图分析显示序列存在广泛的重排(插入、缺失、倒置),排除了端到端多序列比对。作者通过对八个组装基因组着丝粒片段间的局部比对实现一系列双端比对序列。这些菌株平均88.5%的着丝粒区域至少比对到另外一种菌株的同源着丝粒,序列中位数比对长度在607bp。对每一菌株重复性实验的全基因组甲基化数据分析,并将数据与组装序列进行关联。图7C展示了其中A1-35-8(红色片段)的CEN4与其它七个分离株(蓝色片段)着丝粒双比对序列关系,结果显示CG位点的5mC状态可以在任意选取菌株(A1-35-8)的着丝粒和其它七个菌株同源着丝粒的甲基化状态相关联。接下来,对于每一个菌株中的每一个着丝粒CG位点,确定对于给定CG位点能够通过比对鉴定到的菌株数量(N),然后计算甲基化CG位点在所有菌株N中的比例(图7D)。数据统计显示,与空白模型对照相比,不论作为参照菌株,还是着丝粒CG二核苷酸能够比对到的菌株数量,共享甲基化位点增量显著。
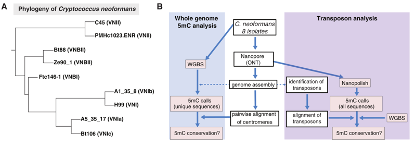

图7
由于C. deuterogattii伴有5mC丢失和完整转座子,因此可能如前所述,DNA甲基化主要发生在Tcn元件。作者进一步检测这些元件与无Tcn着丝粒序列相比,5mC的水平是否表现较高,并且甲基化图谱是否保守。在所有八个菌株中,作者鉴定了着丝粒区域与Tcn1-Tcn6高度同源的序列,包括一个包含两个LTRs的片段,将其作为候选活跃转座子。由于其序列长度以及重复性的特征,全基因组甲基化数据无法很好的覆盖Tcn元件,因此借用Nanopore长读长数据的甲基化信息对全基因组甲基化数据进行补充,且发现与全基因组甲基化数据高度一致。跟关注的Tcn元件甲基化活性相一致,作者发现着丝粒上Tcn相关序列CG甲基化比例远高于非Tcn相关序列(图7E)。
因为发现Tcn3相关序列呈现最长的中位数长度和最多的LTR侧翼元件,因此对其进行深入分析。将最长的Tcn3相关序列(上四分位数)的每一个CG二核苷酸与5mC数据比对。对于绝大部分比对的CG二核苷酸(图7F,垂直列),相对于独自5mC位点次序改变的Tcn3对照模型,共享甲基化比例更高(平均分数0.7对0.4149,p<10-16),表明菌株间5mC模式共享高于期望值。同时注意到,在一些情况下,单个菌株内不同Tcn3元件的甲基化模式相似(图7F),这更可能是重组的结果而非逆转录转座,因为后者通常认为是去除甲基化模式。小部分几乎无甲基化修饰的Tcn3可能在某个点上经历过逆转录转座而因此消除5mC,也可能是一些不再起作用的失活元件。
参考文献:
1.Catania et al., Evolutionary Persistence of DNA Methylation for Millions of Years after Ancient Loss of a De Novo Methyltransferase, Cell (2020).
- Du,J., Johnson, L.M., Jacobsen,S.E., and Patel, D.J. (2015). DNA methylation
pathways and their crosstalk withhistone methylation. Nat. Rev. Mol. CellBiol.
16, 519–532.
-
Jones, P.A. (2012). Functions of DNA methylation: islands, start sites, gene bodies and beyond. Nat. Rev. Genet. 13, 484–492.
-
Simpson, J.T., Workman, R.E., Zuzarte, P.C., David, M., Dursi, L.J., and Timp,W. (2017). Detecting DNA cytosine methylation using nanopore sequencing.
Nat. Methods 14, 407–410.




