大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
2019年12月23日,国家儿童医学中心(上海)、上海交通大学医学院附属上海儿童医学中心潘秋辉教授团队在《Molecular Cancer》(IF:41.444/Q1)杂志上发表了题为“m6A mRNA methylation regulates CTNNB1 to promote the proliferation of hepatoblastoma”的文章,研究通过MeRIP-seq(m6A-seq)等实验揭示了m6A甲基化修饰在肝母细胞瘤(Hepatoblastoma,HB)中的生物学作用和潜在机制。为探究RNA m6A甲基化修饰与肝母细胞瘤的发生发展机制的相关性奠定实验基础,为肝母细胞瘤干预治疗提供新的理论基础。

标题:m6A mRNA methylation regulates CTNNB1 to promote the proliferation of hepatoblastoma(m6A甲基化修饰调控CTNNB1促进肝母细胞瘤增殖)
时间:2019.12.23
期刊:Molecular Cancer
影响因子:IF 41.444
技术平台:MeRIP-seq、RNA-seq、RT-PCR、Western Blot等
项目设计
(1)样本选取:
HB标本和细胞系使用了来自25例患者的原发性HB肿瘤组织及其非肿瘤对应物(邻近正常组织)。
(2)项目设计流程图:

研究目的
m6A修饰对mRNA的稳定性、剪接和翻译的调控具有重要意义。然而,它在癌症中的作用尚未被详细研究,为进一步研究m6A修饰在肝母细胞瘤(HB)中的生物学作用和潜在机制,本研究评估了肝母细胞瘤(HB)组织中m6A的水平,及其关键催化基因和整体m6A转录组水平概况。研究结果表明METTL3是参与异常m6A修饰的主要因素。此外还分析靶基因CTNNB1,探讨了甲基转移酶METTL3在HB中参与调控CTNNB1的机制。
实验结果
(1)m6A修饰在HB中的潜在作用
研究检测了肿瘤和正常组织中的m6A水平,并在肿瘤组织和HB细胞中发现了m6A mRNA水平的升高。此外通过RT-qPCR和Western blotting评估了m6A writers、erasers 和 readers在肿瘤和邻近正常肝组织中的表达。与邻近正常组织相比,大多数肿瘤的METTL3、WTAP、FTO和YTHDF2水平显著上调。这些结果证实了m6A修饰在肿瘤组织中是上调的。
(2)METTL3、WTAP、FTO、YTHDF2在HB中的功能作用
METTL3、WTAP、FTO、YTHDF2基因的敲除显著抑制了HB细胞的增殖,并抑制了其集落形成能力。annexin V-PI双染检测结果显示,METTL3、WTAP、FTO、YTHDF2表达下调会诱导细胞凋亡。考虑到这些基因的催化功能以及m6A修饰的上调水平,最终选择METTL3作为候选分子作为HB中m6A异常修饰的标记。它与m6A水平呈正相关,可能是促进HB增殖的致癌基因以及m6A甲基转移酶的重要活性成分。
(3)METTL3可能是HB患者诊断和预后的生物标志物
对25对HB患者样本进行免疫组化分析。根据分级标准和Pearson s X2检验,发现HB中METTL3水平升高,这表明在mRNA和蛋白水平上与之前的观察结果一致。采用RT-qPCR方法检测组织标本中METTL3的表达,根据中位δ ct值分为高、低组,Kaplan Meier分析显示,METTL3表达增加的患者复发频繁,生存期较差。ROC曲线进一步证实了METTL3的诊断价值,这些结果表明METTL3可能是HB患者诊断和预后的生物标志物。
(4)METTL3促进体内HB肿瘤生长
研究人员通过裸鼠皮下植入实验分析METTL3敲除对HB致瘤性的影响。用慢病毒感染Huh6细胞,表达针对METTL3的shRNA (shMETTL3-1)及其阴性对照(shNC)。RT-qPCR分析证实,与阴性对照相比,shMETTL3-1感染Huh6细胞中METTL3的表达显著下调。在体内实验中观察到相对于非靶shRNA对照,稳定敲除METTL3可以导致肿瘤体积和重量显著减少。此外研究还证实了在METTL3敲除HB细胞Huh6和小鼠肿瘤组织中,CTNNB1下游转录因子TCF4和靶基因Cyclin D1的表达降低。这些实验支持了METTL3在HB肿瘤发生和发展中的致癌作用。
(5)HB中广泛的m6A修饰及其拓扑模式
对5个肿瘤组织和正常组织进行m6A-seq分析,发现五个肿瘤组均表现出高水平的总m6A。m6A-seq在肿瘤组织的1089个基因中鉴定出4071个peaks,对前1000名最重要的peaks值进行分析,发现在显著peaks值中至少有两个motif代表正常组织和肿瘤组织中最常见的consensus。此外研究发现在正常和肿瘤组织中,m6A-IP在终止密码子和CDS周围高度富集,正常组织中CDS(38%)和终止密码子(26.2%)附近的m6A peaks较多,其次是3’ UTR(19.4%)和起始密码子(11.2%)。肿瘤中m6A peaks分布在CDS(36.7%)和终止密码子(29.5%)附近,其次是3’UTR(19.4%)和起始密码子(11.2%)。
(6)m6A peaks与正常组织和肿瘤组织中差异表达基因mRNA的关系
正常组上调表达基因1440个,肿瘤组上调表达基因2868个。对上调基因中m6A peaks的位置进行评估发现大量的m6A peaks位于相应基因的5’端。将含有m6A的基因分为PeakStart和PeakStop组,并检查基因表达与m6A peaks值的位置关系,发现PeakStart和PeakStop类基因整体表达水平较高,且与肿瘤中的m6A peaks有良好的相关性。
由于HB是Wnt/β-catenin驱动的恶性肿瘤,假设上调的m6A甲基化可能通过激活Wnt/β-catenin信号通路促进肿瘤生长,发现CTNNB1、CCND1和NKD1等参与Wnt/β-catenin信号通路的基因,在肿瘤组织中m6A丰度显著增加。这些结果表明m6A在HB转录本中具有与基因表达呈正相关的倾向,并且激活Wnt/β-catenin信号通路。
(7)m6A甲基化调节CTNNB1的激活
CTNNB1是Wnt/β-catenin信号通路的关键组成部分,几乎50 - 90%的HB都存在CTNNB1突变。研究人员首先证实了CTNNB1在HB肿瘤组织中的上调,敲低CTNNB1会显著降低HB细胞的活性并诱导细胞凋亡。此外还发现CTNNB1与METTL3的表达水平显著正相关。通过敲除METTL3细胞来评估CTNNB1表达的稳定性。结果表明,降低m6A甲基化可以降低CTNNB1的稳定性。综上所述,CTNNB1可能是HB中METTL3的下游靶点。
关键图形
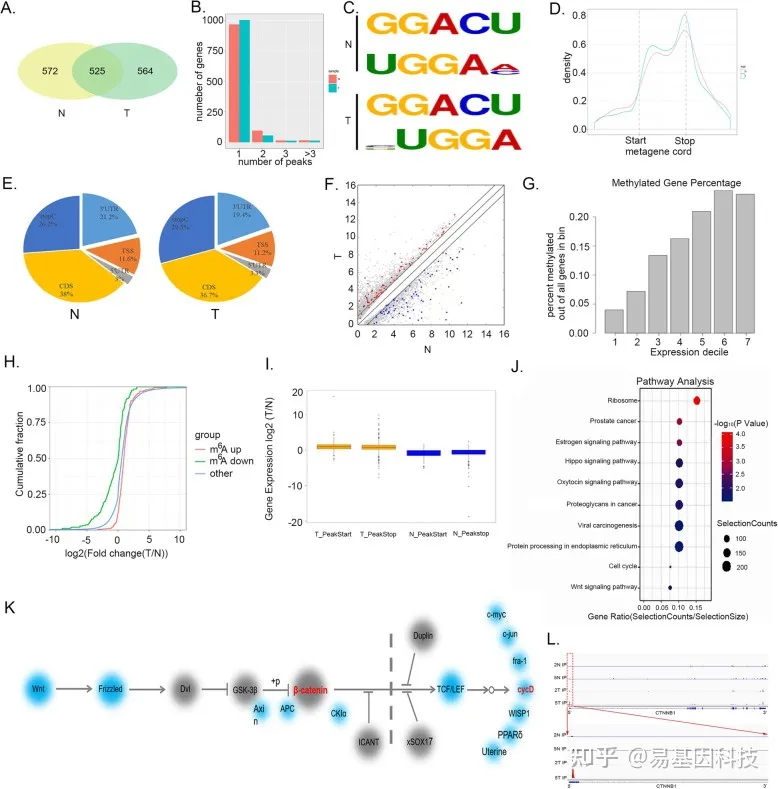
Figure4 HB的m6A- seq分析

Figure5 m6A甲基化与CTNNB1的表达和稳定性呈显著正相关
结论:
该研究在建立肝母细胞瘤RNA m6A修饰研究平台及技术体系基础上,首次发现RNA m6A甲基化修饰在肝母细胞瘤中异常活跃,且筛选出甲基转移酶METTL3与肝母细胞瘤预后相关,有望成为新的预后生物标志物;还发现经典通路Wnt/β-catenin中的CTNNB1发生m6A的异常修饰,并详细阐述了CTNNB1 m6A甲基化修饰的作用机制,该发现是在原有认知的肝母细胞瘤致病机制基础上开阔了一个崭新的研究视野。
关于易基因RNA m6A甲基化测序(MeRIP-seq)技术
易基因MeRIP-seq技术利用m6A特异性抗体富集发生m6A修饰的RNA片段(包括mRNA、lncRNA等rRNA去除所有RNA),结合高通量测序,可以对RNA上的m6A修饰进行定位与定量,总RNA起始量可降低至10μg,最低仅需1μg总RNA。广泛应用于组织发育、干细胞自我更新和分化、热休克或DNA损伤应答、癌症发生与发展、药物应答等研究领域;可应用于动物、植物、细胞及组织的m6A检测。
大样本量m6A-QTL性状关联分析,传统MeRIP单个样品价格高,通常难以承担。易基因开发建立MeRIP-seq2技术,显著提成IP平行性,实现不同样本间相对定量,降低检测成本。
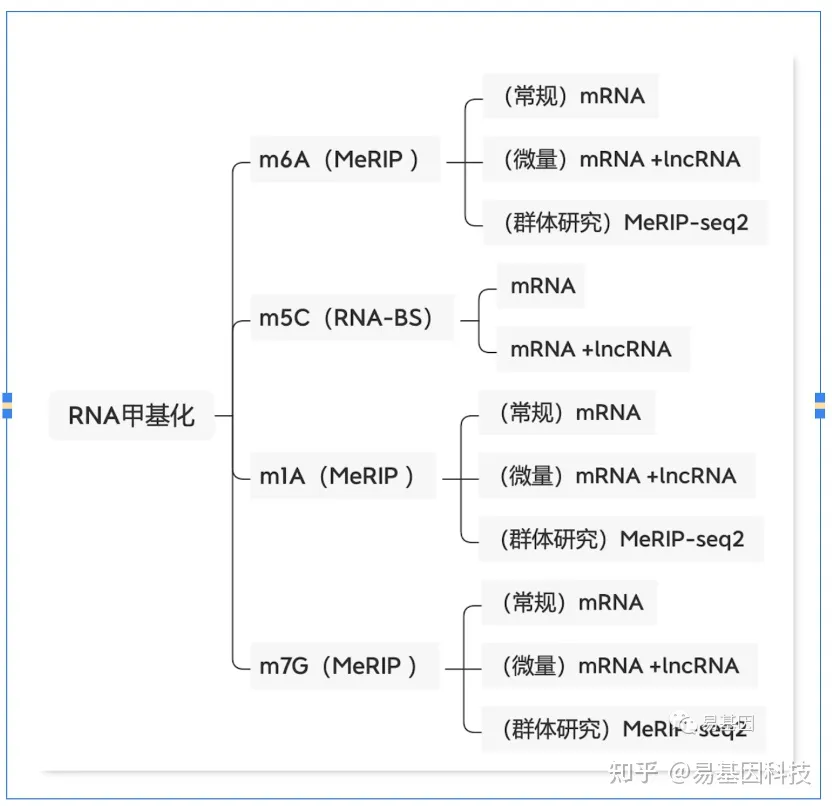
易基因提供适用于不同科研需求的MeRIP技术:
- m6A甲基化-常量mRNA 甲基化测序(MeRIP-seq)
- m6A甲基化-常量mRNA +lncRNA甲基化测序(lnc-MeRIP-seq)
- m6A甲基化-微量mRNA +lncRNA甲基化测序(Micro-lnc-MeRIP-seq)
- 高通量m6A甲基化-常量mRNA甲基化测序(MeRIP-seq2)
技术优势:
- 起始量低:样本起始量可降低至10-20μg,最低仅需1μg总RNA;
- 转录组范围内:可以同时检测mRNA和lncRNA;
- 样本要求:可用于动物、植物、细胞及组织的m6A检测;
- 重复性高:IP富集重复性高,最大化降低抗体富集偏差;
- 应用范围广:广泛应用于组织发育、干细胞自我更新和分化、热休克或DNA损伤应答、癌症的发生与发展、药物应答等研究领域。
研究方向:
m6A甲基化目前主要运用在分子机制的理论性研究
- 疾病发生发展:肿瘤、代谢疾病(如肥胖/糖尿病)、神经和精神疾病(如阿尔兹海默症/抑郁症)、炎症…
- 发育和分化:早期胚胎发育、个体/组织/器官生长发育、干细胞分化与命运决定、衰老
- 环境暴露与响应:污染、抗逆、生活方式
关于m6A甲基化研究思路
(1)整体把握m6A甲基化图谱特征:m6A peak数量变化、m6A修饰基因数量变化、单个基因m6A peak数量分析、m6A peak在基因元件上的分布、m6A peak的motif分析、m6A peak修饰基因的功能分析
(2)筛选具体差异m6A peak和基因:差异m6A peak鉴定、非时序数据的分析策略、时序数据的分析策略、差异m6A修饰基因的功能分析、差异m6A修饰基因的PPI分析、候选基因的m6A修饰可视化展示
(3)m6A甲基化组学&转录组学关联分析:Meta genes整体关联、DMG-DEG对应关联、m6A修饰目标基因的筛选策略
(4)进一步验证或后期试验

易基因科技提供全面的RNA甲基化研究整体解决方案,技术详情了解请致电易基因。
参考文献:
Liu L, Wang J, Sun G, Wu Q, Ma J, Zhang X, Huang N, Bian Z, Gu S, Xu M, Yin M, Sun F, Pan Q. m6A mRNA methylation regulates CTNNB1 to promote the proliferation of hepatoblastoma. Mol Cancer. 2019 Dec 23;18(1):188.
相关阅读:
干货系列:m6A RNA甲基化研究的数据挖掘思路
干货系列:高通量测序后的下游实验验证方法——m6A RNA甲基化篇
干货系列:高通量测序后的下游实验验证方法——m6A RNA甲基化篇
项目集锦 | 易基因近期m6A甲基化(MeRIP-seq)研究成果