 为进一步提高《微生物组实验手册》稿件质量,本项目新增大众评审环节。文章在通过同行评审后,采用公众号推送方式分享全文,任何人均可在线提交修改意见。公众号格式显示略有问题,建议电脑端点击文末阅读原文下载PDF审稿。在线文档(https://kdocs.cn/l/cL8RRqHIL)大众评审页面登记姓名、单位和行号索引的修改建议。修改意见的征集截止时间为推文发布后的72小时,文章将会结合有建设性的修改意见进一步修改后获得DOI在线发表,同时根据贡献程度列为审稿人或致谢。感谢广大同行提出宝贵意见。
为进一步提高《微生物组实验手册》稿件质量,本项目新增大众评审环节。文章在通过同行评审后,采用公众号推送方式分享全文,任何人均可在线提交修改意见。公众号格式显示略有问题,建议电脑端点击文末阅读原文下载PDF审稿。在线文档(https://kdocs.cn/l/cL8RRqHIL)大众评审页面登记姓名、单位和行号索引的修改建议。修改意见的征集截止时间为推文发布后的72小时,文章将会结合有建设性的修改意见进一步修改后获得DOI在线发表,同时根据贡献程度列为审稿人或致谢。感谢广大同行提出宝贵意见。
原生质体法制备根系腐生型共生菌(伞菌目)单核化菌丝
Protoplast regeneration to obtain monokaryotic strain from the Quercus-associated Clitopilus hobsonii
王玉宸1, 2,王欣玉 2, $,彭龙1, 2,袁志林1, 2 *
1林木遗传育种国家重点实验室,中国林业科学研究院,北京;2,中国林业科学研究院亚热带林业研究所,杭州;$现工作单位:台州学院,医学院,台州
*通讯作者邮箱: yuanzl@caf.ac.cn
摘要: 菌体单核化的方法主要包括机械分割法、化学处理法、孢子分离法和原生质体再生法等。机械分割法对细胞破坏程度大,分割后细胞存活率降低,而且分割产物不均匀,单核体得率低;化学处理法对于处理条件要求严格,具有细胞毒性;孢子分离法仅适用于产孢菌株单核体的制备,且单核孢子的形成需要经过遗传重组、减数分裂,因而遗传信息不能完整保留;原生质体再生法制备的单核体未经过减数分裂及遗传重组,亲本细胞核内的遗传信息能更为完整、准确地保留,而且经过再生后能够获得两种核型的单核体。本文介绍了异核双核体担子真菌原生质体再生单核化技术,以及单核体菌株的鉴定方法,为开展真菌基因组测序和高质量组装提供技术依据。
关键词:担子菌,单核体,原生质体
研究背景:荷伯生氏斜盖伞Clitopilus hobsonii(Agaricales)首次分离自栎类林腐木(Orton , 1960),是典型的土壤腐生真菌。笔者发现该菌在大果栎Quercus macrocrpa、舒玛栎Quercus shumardii、牛栎Quercus michauxii、琴叶栎Quercus lyrata、白栎Quercus fabri等新鲜菌根组织中均能分离到。进一步研究发现,该菌能够促进栎类幼苗生长,且与多种苗木建立共生关系,形成类似“微菌核”的侵染结构,促进苗木根系发育和养分吸收。笔者认为破译该菌的遗传信息可能为丰富外生菌根真菌起源于腐生真菌这一学说提供更多的基因组进化证据。然而该菌株为异核双核体,基因组存在不同程度的杂合率,这将给基因组组装和注释带来一定挑战,影响后续进化生物学分析和功能研究。单核化作为“简化”复杂基因组的一种策略,是有效开展异核双核真菌全基因组测序分析的重要环节。因此,制备荷伯生氏斜盖伞单核体菌株将为顺利开展基因组测序和组装奠定基础。
材料与试剂
1.荷伯生氏斜盖伞双核体菌株
2.DAPI (sigma, catalog number: D9542)
3.细胞壁裂解酶 (sigma, catalog number: L1412)
4.真菌基因组DNA快速提取试剂盒 (Aidlab, catalog number: DN4102)
5.I-5™ 2x High-Fidelity Master Mix (TSINGKE, catalog number: TP001)
6.琼脂糖 (Genview, catalog number: 9012-36-6)
7.超纯水
8.ddH2O
9.PDA培养基 (见溶液配方)
10.YMG液体培养基 (见溶液配方)
11.MYG再生培养基 (见溶液配方)
12.0.5 M蔗糖溶液 (见溶液配方)
13.0.1 M PBS缓冲液 (见溶液配方)
14.葡萄糖 (Macklin, catalog number: D810588)
15.酵母提取物 (Oxoid, catalog number: LP0021)
16.琼脂 (BD, catalog number: 214010)
17.麦芽提取物(Cool Chemistry, catalog number: BL360016)
18.蔗糖 (沪试, catalog number: 10021418)
19.NaCl (沪试, catalog number: 10019318)
20.KH2PO4 (沪试, catalog number: 10017618)
21.K2HPO4 (沪试, catalog number: 20032118)
仪器设备
1.盖玻片
2.载玻片
3.打孔器
4.纱布
5.无纺布
6.10 ml注射器
7.250 ml锥形瓶
8.涂布棒
9.漏斗
10.镊子
11.直尺
12.0.45 μm滤膜
13.50 ml离心管
14.电泳仪 (六一, model: DYY-6C)
15.全温振荡器 (培英, model: THZ-C-1)
16.生化培养箱 (博讯, model: SPX-250B-Z)
17.台式高速冷冻离心机 (Sigma, model: 3K15)
18.电热鼓风干燥箱 (新诺, model: 101-0AS)
19.立式压力蒸汽灭菌器 (申安, model: LDZF-50KB-II)
20.激光共聚焦荧光显微镜 (Carl Zeiss, model: LSM700)
21.PCR扩增仪 (BIO RAD, model: MyCyclerTM Thermal Cycler)
22.光学显微镜 (Carl Zeiss, model: Axio Scope A1)
实验步骤
一.原生质体的制备及再生
1.原生质体制备
1.1以0.5 M蔗糖溶液作为渗透压稳定剂配置浓度为10 μg/ml的裂解酶溶液20 ml, 先以0.45 μm 滤膜过滤除菌,再以0.22μm滤膜二次过滤除菌后转移至50 ml离心管中备用。
1.2用6 mm打孔器取10个菌丝块接种至100 ml的YMG液体培养基中,25 °C、120 r/min避光培养7 d,然后以四层纱布过滤收集菌丝体,用无菌水洗涤三次,再用0.5 M蔗糖溶液洗涤一次。
1.3将洗净后的菌丝体置于10 μg/ml的裂解酶溶液中使用手持式搅拌机打碎,使其均匀分布 (1 g菌丝/5 ml酶液),于37 °C、70-80 r/min条件下震荡酶解3 h。
1.4酶解后将菌丝酶解液经无纺布过滤,得到较纯的原生质体初悬液,将所得滤液在4 °C下3000 r/min离心10 min,弃上清,得到原生质体沉淀;用0.5 M蔗糖溶液冲洗2次,弃上清,保留沉淀。
1.5用1 ml 0.5 M蔗糖溶液重悬沉淀。
1.6将得到的原生质体重悬液置于光学显微镜下进行镜检,用血球计数板计数并记录拍照原生质体形态。
2.原生质体的再生
2.1将100 μl原生质体重悬液均匀涂布于MYG再生培养基上,25 °C避光培养,每天观察菌落生长情况。
2.2挑取10 d后长出的再生单菌落转接至PDA固体培养基上,25 °C避光条件下培养14 d后即可获得原生质体再生菌株,一次挑选10株为宜。
二.原生质体再生菌株的鉴定
1.再生菌株与出发双核体菌落的培养特征比较分析
1.1菌落形态特征观察,用打孔器沿再生菌株、出发菌株的菌落边缘打孔,分别 接种于PDA固体培养基中心,置于25 °C培养箱,避光培养12 d后观察菌落表型特征 (包括菌落正反面及边缘特征、色素、生长速度等),并拍照记录。
1.2再生菌株与出发菌株生长速度测定,用打孔器沿原生质体再生菌落、出发菌 落的边缘打孔,分别接种至PDA固体培养基中心,25 °C避光培养,以十字交叉法测量菌落直径,计算菌丝的生长速度 (mm/d),每个菌株设置3次生物学重复。
2.再生菌株与出发双核体菌株的显微形态比较分析
2.1菌丝形态观察,用打孔器沿原生质体再生菌落、出发菌落的边缘打孔,分别 接种至PDA固体培养基中心,25 °C避光培养3 d,在菌落边缘斜插入无菌盖玻片,待菌丝延伸至盖玻片的2/3处时,在无菌条件下取出盖玻片,制片后置光学显微镜下镜检,100×油镜下观察两类菌株的菌丝是否存在锁状联合结构。
2.2细胞核荧光染色及观察,参考胡晓棣等 (2016) 的方法并稍作改动。用镊子取出经插片培养后附着有菌丝体的盖玻片,擦去背面菌丝,向菌丝附着位置均匀滴加50 μl DAPI染色液,避光条件下对再生菌株、出发菌株的菌丝进行细胞核染色3-5 min,然后用磷酸盐缓冲液 (PBS) (pH 6.8,0.1 M) 洗涤3次。将带有菌落一侧盖玻片朝下,盖到滴有PBS缓冲液 (pH 6.8,0.1 mol/L) 的载玻片上,置于荧光显微镜下镜检 (激发光与发射光波长分别为340、488 nm)。
3.基因杂合位点鉴定
3.1用真菌基因组DNA快速提取试剂盒分别提取荷伯生氏斜盖伞再生菌株、出发菌株的基因组DNA。
3.2采用fRPB2-5F/fRPB2-7cR和983F/2218R两对引物分别扩增rpb2 (Liu等,1999) 和tef-1α (Rehner和Buckley 2005) 基因片段。PCR扩增体系 (50 μl) 如下:25 μl 1-5TM2× High-Fidelity Master Mix,1 μl模板DNA (10-20 ng/μL),上下游引物各2 μl (10 μmol/L),加20 μl ddH2O。rpb2基因PCR扩增程序:95 °C预变性4 min;95 °C变性40 s,55 °C退火50 s,72 °C延伸1.5 min,共扩增35个循环;最后72 °C延伸10 min。采用降落PCR (touch-down PCR) 方法扩增tef-1α基因,降落PCR程序参考Kluting等 (2014) 的方法并稍作改动:94 °C预变性4 min;94 °C变性40 s,66 °C退火50 s,每循环下降1 °C,72 °C延伸1.5 min,10个循环;94 °C变性40 s,56 °C退火50 s,72 °C延伸1.5 min,共扩增30个循环;最后72 °C延伸10 min。
3.3PCR扩增产物经1.5 %琼脂糖凝胶电泳检测出条带后送擎科生物科技有限公司进行测序。采用SeqMan v.5.0软件分别对再生菌株、出发菌株的rpb2基因和tef基因进行核酸序列比对和双峰杂合位点鉴定。
结果与分析
本实验通过酶解实验成功获得了荷伯生氏斜盖伞的原生质体,经再生培养可观察到两种形态的菌落:一类表现为菌丝快速萌发 (大多出现在再生培养前期),菌丝形态略稀疏且呈毛毡状,菌落边缘放射状,与出发菌株的菌落并无明显差异(图1B);另一类表现为菌丝缓慢萌发 (大多出现在再生培养后期),菌丝形态浓白且呈绒毛状,菌落边缘明显,与出发菌落形态差异较大 (图1C),该类型菌落体可能是潜在的单核体菌落。
以其中一株菌落特征具有较明显差异的再生菌株为材料,比较该菌株与出发菌株的菌落形态特征和生长速度差异:培养结果表明再生菌株呈辐射状生长,菌丝呈浓密绒毛状,在平板反面形成裂褶状纹路并有深黄色色素分泌 (图2A)(表1)。而出发菌株生长呈现轮纹状扩大,菌丝稀疏毛毡状,无裂痕特征,有淡黄色色素分泌 (图2B)。此外,出发菌株的菌丝生长速度为5.72±0.07 mm/d,再生菌株的菌丝生长速度为2.82±0.03 mm/d,显著低于出发菌株的菌丝生长速度 (表1)。
锁状联合结构是双核体担子菌特有的菌丝结构,通常作为双、单核体担子菌的鉴定依据 (丛倩倩等 2019;徐思佳等 2020)。然而,镜检结果表明,本研究中出发菌株与再生菌株的菌丝中均未发现锁状联合结构 (图3),这与Singer (1986)研究结果一致。此外,DAPI染色结果表明,再生菌株的菌丝中相邻细胞核之间被隔膜分开,每个菌丝细胞中仅有一个细胞核 (图4A),而出发菌株的菌丝细胞中含有两个明显可见的细胞核 (图4B)。据此可判断本研究所获得菌株为单核体。
由于异核体菌株基因型杂合,因此在基因Sanger测序峰图中通常会出现套峰,即存在杂合位点;而单核体菌株基因型理论上应该是纯合的 (Huang等,2010),不存在杂合位点。通过对rpb2和tef-1α基因片段序列峰图比对发现,出发菌株存在多个杂合位点,尤其是rpb2基因 (图5A;图5C);而再生菌株均未发现杂合位点 (图5B;图5D)。
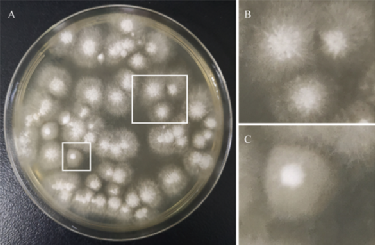
图1. 原生质体再生菌落形态
注:A:再生菌落;B:再生菌落类型l;C:再生菌落类型II。
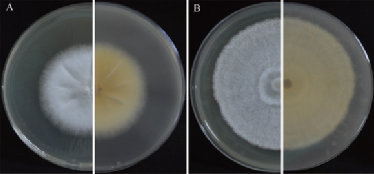
图2.荷伯生氏斜盖伞再生菌株与原出发双核体菌株菌落形态
注:A:再生菌株菌落(正、反);B:出发菌株菌落(正、反)。

图3. 荷伯生氏斜盖伞再生菌株与原出发双核体菌株菌丝显微形态
注:A:再生菌株;B:出发菌株。
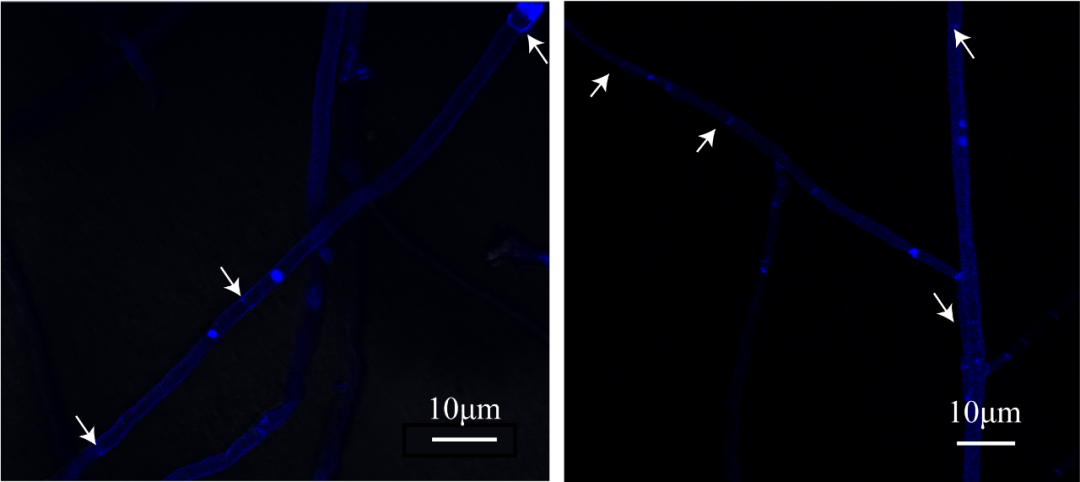
图4. 荷伯生氏斜盖伞再生菌株与原出发双核体菌株细胞核染色
注:A:再生菌株;B:出发菌株. 箭头:隔膜。
表1. 再生菌株与出发菌株的生长特征比较
菌株材料 | 菌落生长速度(mm/d) | 菌丝致密程度 |
再生菌株 | 2.82±0.03 b | +++ |
出发菌株 | 5.72±0.07 a | + |
注:t检验进行差异显著性分析,小写字母表示数据间差异显著,P<0.01< span="">;“+++”表示菌丝浓密,“+”表示菌丝稀疏。

图5. rpb2、tef-1α基因部分序列比对
溶液配方
1.PDA培养基
马铃薯 200 g (去皮煮熟后过滤取汁液)
葡萄糖 20 g
琼脂 15 g
加超纯水至1 L,调pH至7.0,115 °C高温高压灭菌30 min
2.YMG液体培养基
葡萄糖 10 g
酵母提取物 4 g
麦芽提取物 10 g
加超纯水至1 L,调pH至7.0,115 °C高温高压灭菌30min
3.MYG再生培养基
葡萄糖 10 g
酵母提取物 4 g
麦芽提取物 10 g
琼脂 15 g
加0.5 M蔗糖溶液至1 L,调pH至7.0,115 °C高温高压灭菌30min
4.DAPI染色液
用无菌水将DAPI配制成50 μg/ml的母液,-20 °C条件下保存
染色时,用PBS缓冲液 (pH = 6.8,0.1 M) 稀释成5 μg/ml的工作液,4 °C、避光条件下使用
5.0.1 M 磷酸盐缓冲液 (PBS)
0.1 M KH2PO4 510 ml
0.1 M K2HPO4 490 ml
NaCl 8 g,pH = 6.8
6.裂解酶
用0.5 M蔗糖溶液配制浓度为10 μg/ml的裂解酶溶液,先以0.45 μm滤膜过滤除菌,再以0.22μm滤膜二次过滤除菌。
7.0.5 M蔗糖溶液
蔗糖171.5 g
加超纯水至1 L,以0.45 μm滤膜过滤除菌再以0.22μm滤膜二次过滤除菌。
致谢
本工作得到国家自然科学基金项目 (项目编号:31722014; 31901290) 的经费资助。
参考文献
1.丛倩倩,崔晓,王庆武. (2019). 灵芝原生质体单核菌株的制备及形态特性比较. 食用菌 41(2): 34-36.
2.胡晓棣,李熠,任蜀豫,姚一建. (2016). 冬虫夏草、蛹虫草菌丝隔膜和细胞核荧光染色. 菌物学报 35(9): 1099-1105.
3.徐思佳,万佳宁,李焱,李燕,尚俊军,鲍大鹏. (2020). 刺芹侧耳交配型基因敲入单核体后锁状联合和核相的表征观察. 菌物学报 39(6): 1130-1138.
4.Huang, C., Xu, J., Gao, W., Chen, Q., Wang, H. and Zhang, J. (2010). A reason for overlap peaks in direct sequencing of rRNA gene ITS in Pleurotus nebrodensis. FEMS Microbiology Letters 305(1): 14-17.
5.Kluting, K.L., Baroni, T.J. and Bergemann, S. E. (2014). Toward a stable classification of genera within the Entolomataceae: a phylogenetic re-evaluation of the Rhodocybe-Clitopilus clade. Mycologia 106(6): 1127-1142.
6.Liesche, J., Ziomkiewicz, I. and Schulz, A. (2013). Super-resolution imaging with Pontamine Fast Scarlet 4BS enables direct visualization of cellulose orientation and cell connection architecture in onion epidermis cells. BMC Plant Biology 13: 226-226.
7.Liu, Y. J., Whelen, S. and Hall, B. D. (1999). Phylogenetic relationships among ascomycetes: evidence from an RNA polymerse II subunit. Molecular Biology and Evolution 16: 1799-1808.
8.Rehner, S. A. and Buckley, E. (2005). A Beauveria phylogeny inferred from nuclear ITS and EF1-α sequences: evidence for cryptic diversification and links to Cordyceps teleomorphs. Mycologia 97: 84-98.
9.Singer, R. (1986). The Agaricales in modern taxonomy. In: Koeltz Scientific Books. 4th edition. Germany, Koenigstein. 699-700.
猜你喜欢
10000+:菌群分析 宝宝与猫狗 梅毒狂想曲 提DNA发Nature Cell专刊 肠道指挥大脑
系列教程:微生物组入门 Biostar 微生物组 宏基因组
专业技能:学术图表 高分文章 生信宝典 不可或缺的人
一文读懂:宏基因组 寄生虫益处 进化树
必备技能:提问 搜索 Endnote
文献阅读 热心肠 SemanticScholar Geenmedical
扩增子分析:图表解读 分析流程 统计绘图
16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
在线工具:16S预测培养基 生信绘图
科研经验:云笔记 云协作 公众号
编程模板: Shell R Perl
生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流、快速解决科研困难,我们建立了“宏基因组”专业讨论群,目前己有国内外5000+ 一线科研人员加入。参与讨论,获得专业解答,欢迎分享此文至朋友圈,并扫码加主编好友带你入群,务必备注“姓名-单位-研究方向-职称/年级”。PI请明示身份,另有海内外微生物相关PI群供大佬合作交流。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍未解决群内讨论,问题不私聊,帮助同行。

学习16S扩增子、宏基因组科研思路和分析实战,关注“宏基因组”
 点击阅读原文下载PDF审稿,或浏览器直接访问下载链接:http://210.75.224.110/github/MicrobiomeProtocol/04Review/210218/2003653ZhilinYuan952658/Protocol2003653.pdf
点击阅读原文下载PDF审稿,或浏览器直接访问下载链接:http://210.75.224.110/github/MicrobiomeProtocol/04Review/210218/2003653ZhilinYuan952658/Protocol2003653.pdf


![孢子(SPORE)试玩3小时[纯水贴 ^_^]](https://p-blog.csdn.net/images/p_blog_csdn_net/linqingfeng/EntryImages/20080910/孢子_第三阶段_003.jpg)

