来源:融视影像科技
综述
医学影像的处理有两个特质。一是复杂,整个处理流程涉及多种算法,需要调整的参数较多。二是发展快,从采集、重建到后处理,新处理方法层出不穷,每年都有新工具,每五年又有个大热点。因此影像后处理工具主要有两方面的要求:易用性(交互参数少)和易拓展性(加入新功能快,第三方开发容易)。所有的工具都必须要平衡这两方面上找到一个定位(下图)。

果要强调易用性,整个软件架构就要封闭一些,首先开放给用户调整的参数就不能太多,其次要保证计算速度,设计上多考虑并发和内存传递,最后是要有一个设计良好的用户界面。各种工作站就是此类产品的例子,操作按照解剖部位和处理需求进行流程优化,基本上点点鼠标就能完成。但是这样一来,可扩展性就不强,虽然也有可能做一些插件接口支持用户开发(如飞利浦的PRIDE和GE的Func2),但开发必须符合设定好的框架,空间很小。更极端的例子是CAD软件,譬如invivo的DynaCAD之类,使用上非常简便,但基本不考虑扩展。
如果要强调可拓展性,整个软件架构就要非常开放。开放的极致就是一个各种工具组成的包,譬如FSL,DIPY等,用户自己设计处理流程,自由组合工具函数,并加入自己开发的模块。可扩展性极强。稍微用户友好一些的是MITK(作者为德国癌症研究中心的那套软件www.mitk.org),MRtrix之类带图形界面的后处理工具包,用户可以通过修改编译脚本来自行调配处理流程和组件。开放的代价是易用性很差,如果科研团队里没有熟悉编程的计算机背景的成员,几乎不能真正用上这些工具。
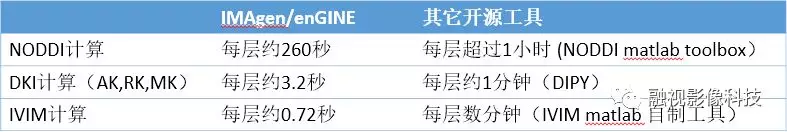
我们在开发IMAge/enGINE(核磁弥散处理工具新版本)的时候,考虑面向的用户是没有工程团队支持的医学背景的大夫和研究生,因此尽量向易用性优化了,因此界面操作简单,计算速度快。举例来说,在i5-4570 CPU的计算机上测试,与其它开源工具比较如下。

OsiriX(http://www.osirix-viewer.com)是一款运行在Mac OS上的DICOM浏览器 (只有Mac OS版,无MAC的同学请跳过此节 )。其收费版,也就是OsiriX MD版是通过FDA认证的,可以在很多国家和地区直接用于临床。OsiriX也提供免费版,代价是运行过程中有弹窗跳出,需要等待若干秒才能继续使用。OsiriX的功能主要集中在影像融合,影像渲染,影像标注上,计算参数图并不是强项。其提供了第三方开发接口(https://github.com/pixmeo/osirixplugins),有很多第三方的插件,譬如提供结构化报告的Pi-RADS Bi-RADS 冠脉分析等插件,也有计算ADC的IB diffusion,计算DCE的IB DCE等等。
3DSlicer (https://www.slicer.org/)可能是发展最好,最全面的免费+开源影像处理工具了。它可以运行在Mac OS, windows和linux上。3DSlicer主要是哈佛大学医学院布列根妇女医院(BWH),麻省总医院(MGH),依阿华大学(University of Iowa)和GE医疗开发的。3DSlicer是用04年到现在超过47个的NIH经费灌溉出来的。光弥散方向的插件,就有04,13,15年的3项经费。另外还有放疗引导,手术引导,放射组学,深度学习,XNAT接口,胸部影像平台(Chest Imaging Platform)等海量功能组件。而且要相信哈佛医学院系统拿funding的能力,他们可以一直搞新玩意儿,因此开发社区也极其活跃,新东西层出不穷。譬如这两年火爆的深度学习,就已经有Deep Infer提供的前列腺分割和肺外形分割组件了。其中前列腺分割号称DICE85+,建议各位把自己数据灌进去玩一玩,省得天天吹CNN也不知道最后替谁背了锅。基本上,掌握了3DSlicer,大部分花钱的科研平台就是来搞笑的……(部分组件见下图)
更加难能可贵的是,这是一个开源平台(Why?在美国,纳税人经费做的当然要开源),许可非常宽松,允许改造、商用等等。我们仔细阅读过它的源码,在开发IMAgen/enGINE的过程中,也借鉴了它的很多模块(如DICOM导入管理部分)。我们码农界有句老话,叫Talk is cheap, show me the code (吹水无用,请给代码)。我们看了Slicer的代码,真心膜拜人家在代码架构,多团队合作管理,版本控制方面的专业性。
但是,正如它在易用性-易扩展性图中的位置一样,它为了多单位合作,支持更多的开发者,将插件接口做得极其开放。因此有2个缺点,1是暴露的参数太多了,使用非常复杂,必须仔细看文档或者参加培训班才容易上手。自己瞎玩的话,大概率连个弥散数据都灌不进去。(或许……我们可以开个培训班赚钱?)2是计算速度慢,缘于其使用了大量的硬盘文件缓存方式进行模块间通讯。举例来说,下图是UKF(Unscented Karman Filter)白质纤维束追踪插件的参数界面,此种纤维束追踪方法比较新,对数据量要求小追踪效果好纤维束不容易断,而且支持NODDI模型的追踪。不过我感觉对于不懂技术细节的临床背景研究者,只能抓瞎了:


ImageJ(https://imagej.nih.gov)是一个工程师界的传奇。它是NIH一个工作人员Wayne Rasband利用自己的业余时间搭建的影像处理工具,开发者2010年从NIH退休之后,还在继续做很多工作。它也拥有大量的第三方插件(列表见此https://imagej.nih.gov/ij/plugins)。和大部分影像处理软件不同,这是个使用Java语言开发的工具,曾经还以applet方式提供在线使用。假设您团队的开发者更熟悉Java系的工具,这应该是您首选的科研工具。不过它也存在用户友好性的问题,而且界面比3DSlicer更加粗陋。因为我们不用Java,这个工具我们用得比较少。各位可以自己官网查阅资料。
MITK

The Medical Imaging Toolkit (MITK, www.mitk.org)是德国癌症协会(German Cancer Research Center)搞的一套跨平台的免费+开源软件。虽然有图形界面,但它更像是一套工具集而不是一个软件。它可以理解为基于QT,ITK,VTK,OpenCV编写了一些自有的图形组件和算法库。然后通过灵活编写cmake脚本和使用它的预编译宏,可以把这些组件组成一个应用软件。譬如官方搞的MITK Diffusion,可以做HARDI之类的运算和追踪。下图是一个DTI的一个界面:

根据上次文章所说的易用性和开放性成反比的理论,MITK架构设计灵活,易用性就比较差。根据我的使用经验,以它的工具库组织起来的软件往往不太稳定,bug比较多。各位可以试着下载安装版试试,不经历一番摸爬滚打恐怕是用不了的。MITK更像是给研究团队中的工程技术人员根据科研需求,快速定制自制软件用的。同样在德国的一家公司出品的mevislab软件应该是延续了这个思路(https://www.mevislab.de/),工程团队可以用mevislab快速把算法界面化,给医疗团队使用。这种研究团队种包含了有开发实力的工程技术人员的情况,在国内可能不太多见。这也是我们把IMAge/enGINE往易用性方面优化的原因。
另外再介绍两种专门用来做分割的后处理工具:
ITK Snap

ITK Snap (http://itksnap.org) 是跨平台的免费开源软件。支持Mac OS,WIN和Linux。其界面走技术硬核风,如下图:
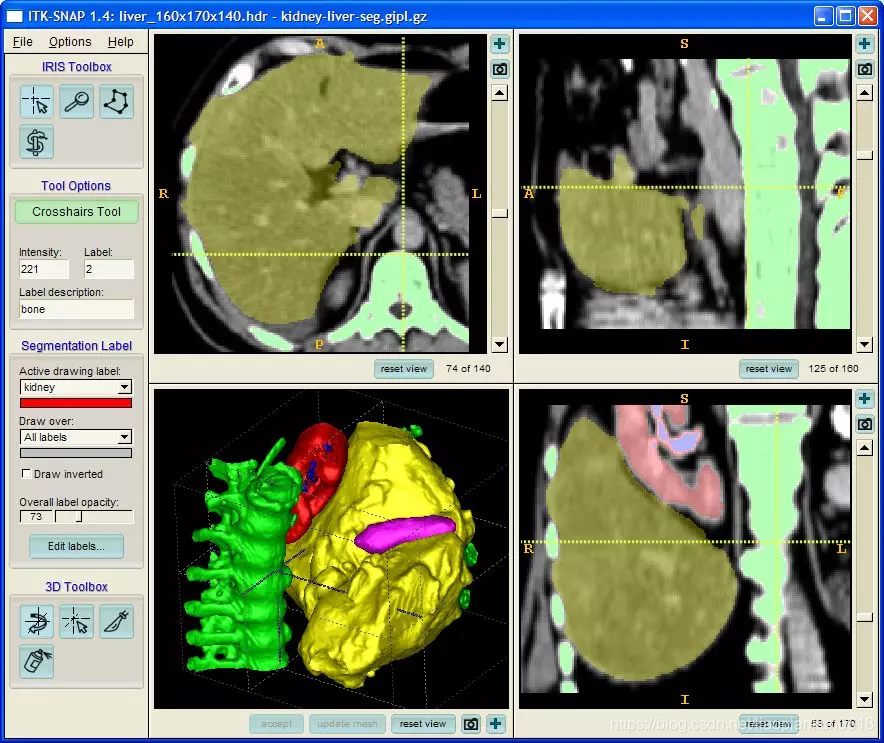
手动分割工具齐全而且实现了大量的ITK半自动方法,而且软件架构比较开放,很方便往里塞ITK写的新方法,比较适合做分割算法开发的工程师们用来调参数或者尝试新算法。对于医学背景的同学们,我更推荐下面这个软件:
Seg3D
Seg3D (http://www.sci.utah.edu/download/seg3d/)是一款交互设计非常独树一帜的免费开源影像处理软件。考虑到它的开发单位犹他大学与Photoshop的渊源,Seg3D基于“蒙版”和“图层”的使用也并不意外。每一步分割算法都是生成一个新的“蒙版”,蒙版之间也可以便捷的逻辑运算。不过Seg3D里的半自动方法比较有限,只有区域生长,阈值之类的几种。软件比较封闭,想要二次开发加入新的方法也不是那么容易。下图是一个CT增强的颅内血管分割的小例子
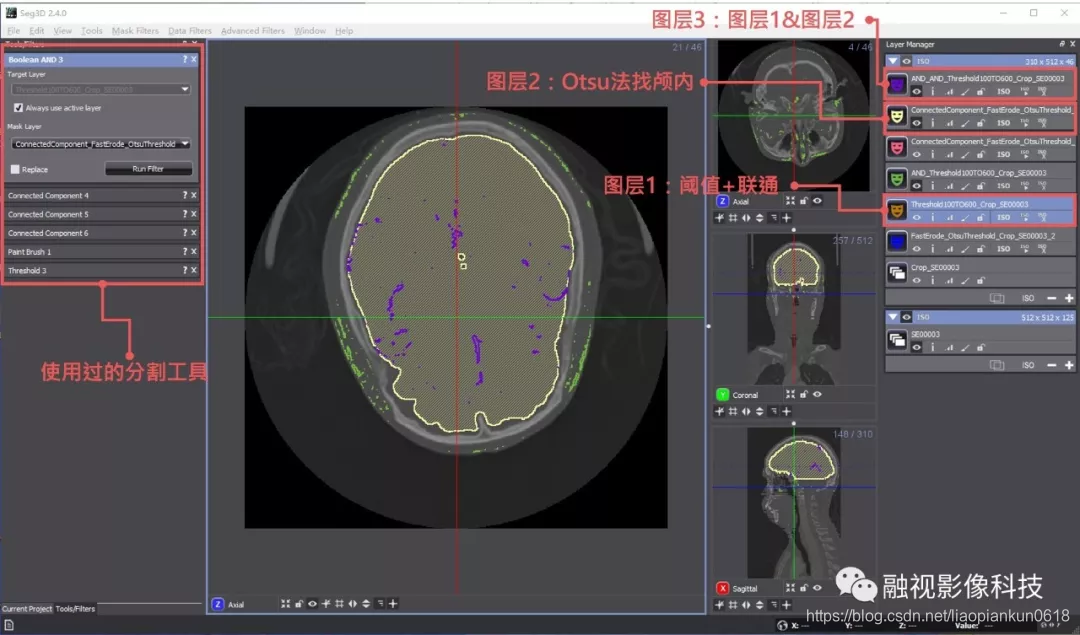
首先可以使用阈值和联通域找到血管(图层一,绿色),但很容易连到骨皮之类其它类似信号的区域上。
然后再从原始数据使用Otsu法找到颅内,进行几次腐蚀,确保颅内区域远离骨皮(图层二,黄色)。
最后把图层一和图层二做一个“与”逻辑(图层三,紫色),就能粗略地得到颅内的血管部分了。
结合手动方法,愿意花时间的话,应该说可以满足绝大多数医学影像分割需求了。还可以把分割好的模型直接3D打印。