PubChem
【官网 https://pubchem.ncbi.nlm.nih.gov/】
简介
PubChem is the world’s largest collection of freely accessible chemical information. Search chemicals by name, molecular formula, structure, and other identifiers. Find chemical and physical properties, biological activities, safety and toxicity information, patents, literature citations and more.
PubChem,即有机小分子生物活性数据,是一种化学模组的数据库,由美国国家健康研究院( US National Institutes of Health,NIH)支持,美国国家生物技术信息中心负责维护。
PubChem数据库包括 3个子数据库:
- PubChem Compound用于存储整理后的化合物化学结构信息
- PubChem Substance用于存储机构和个人上传的化合物原始数据
- PubChem Bioassay用于存储生化实验数据,实验数据来自于高通量筛选和文献
PubChem数据库属于NCBI旗下,收录11100万种化合物结构信息存储于PubChem Compound子数据库,27100万种用户上传的化合物数据存储于PubChem Substance子数据库,29800万种实验结果或文献支持的化合物生物活性数据存储于PubChem BioAssay子数据库,还有3200万篇相关文献和250万相关专利,以及90426个靶基因、96561个靶蛋白和23915条通路信息,上述数据来源总计799个。PubChem数据库包含大量化合结构信息、理化性质及生物活性、毒性和安全性数据,并提供详实的文献或专利支持,广为生物医药与生物化学交叉领域的科研人员所青睐。
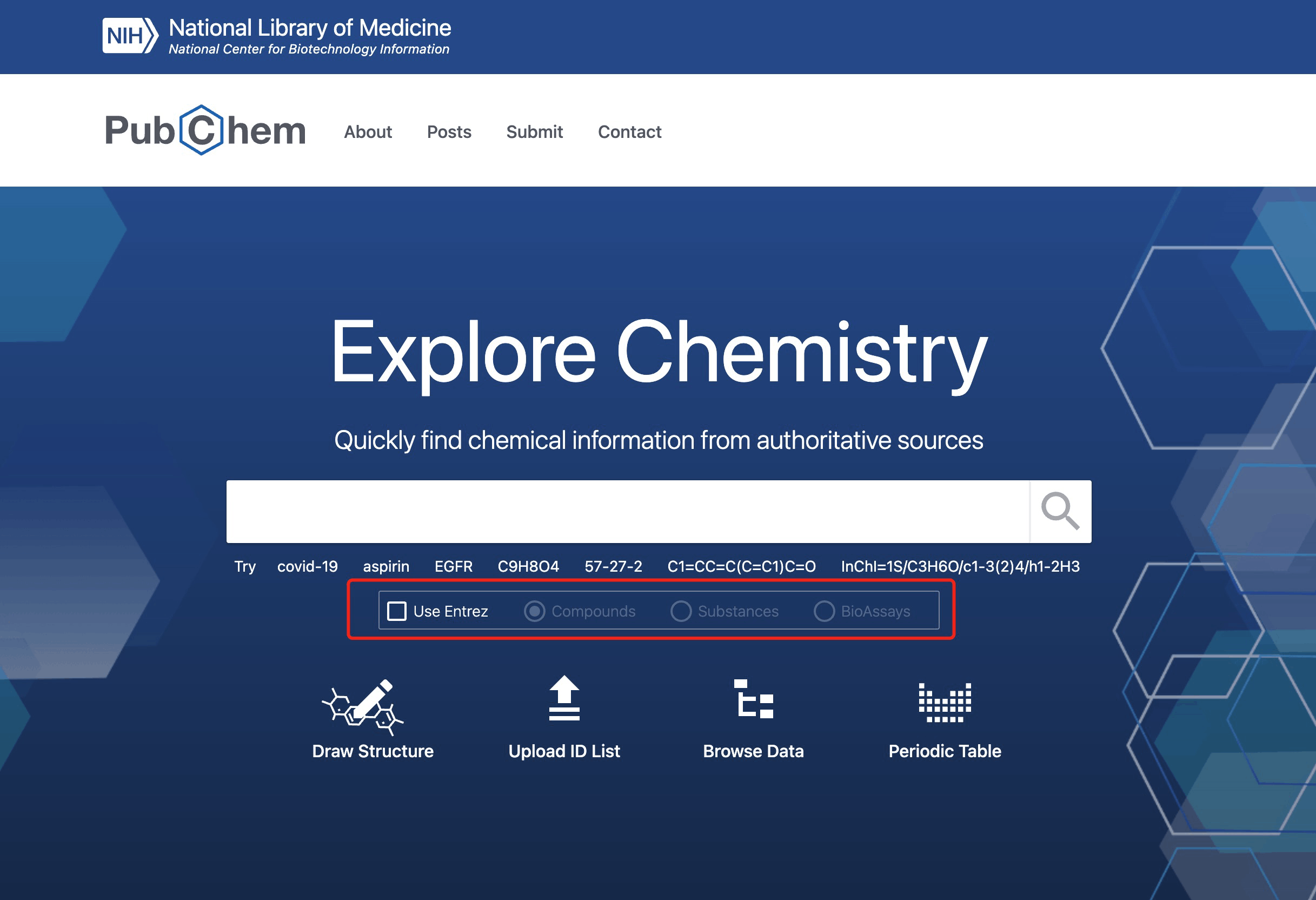
数据查询方法
- 关键词检索
在主页检索框输入关键词进行快速检索,检索词支持输入化合物名称、化学式、CAS ID号、SMELE和InchI表达式,或基因名,并提供Covid-19专题检索功能。
以阿司匹林aspirin为例,检索结果有121条化合物结构信息,包括阿司匹林单体和混合药物,有25条通路数据,1998条药物活性数据,近7万文献或专利。
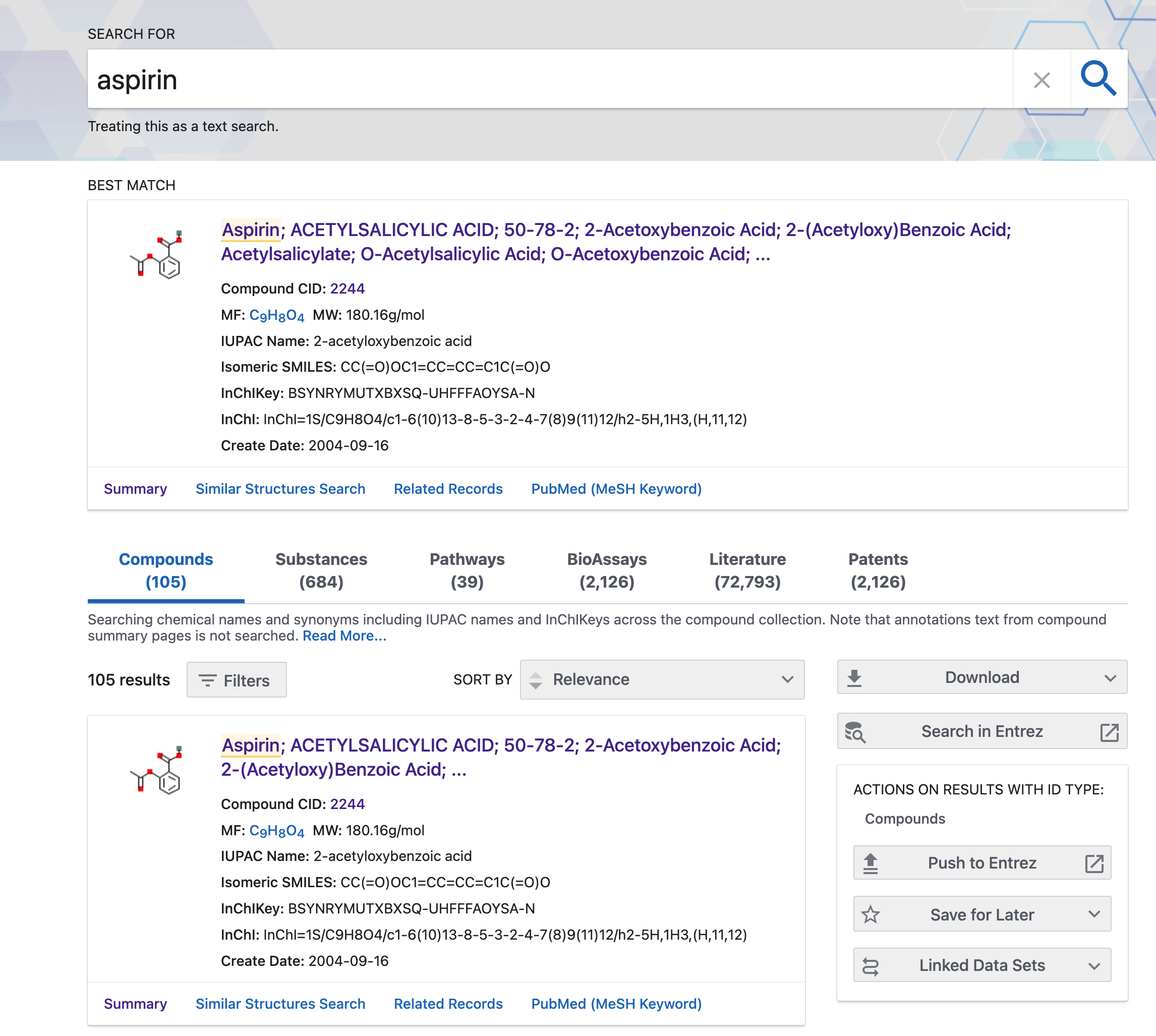
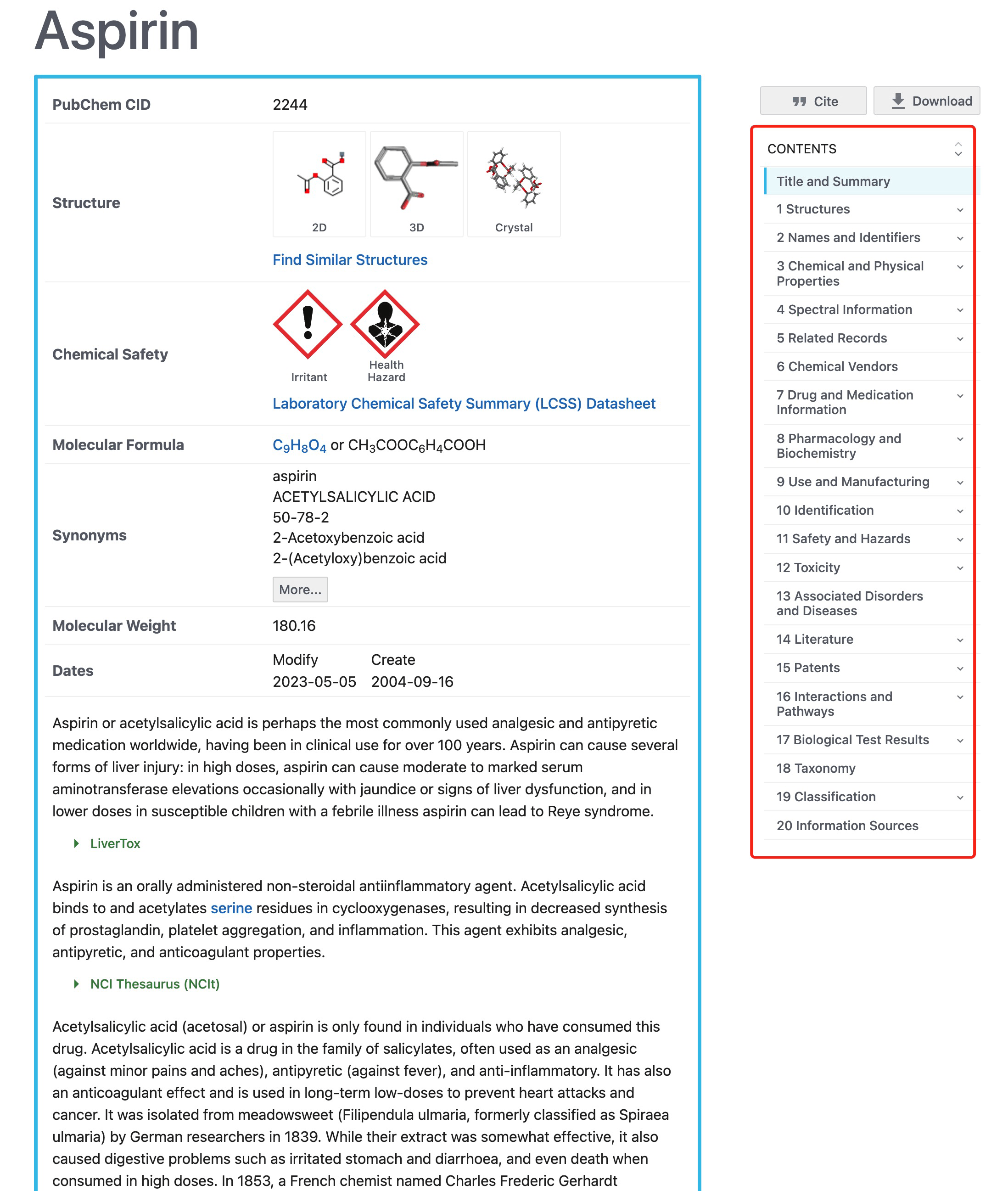
点击Compounds目录下第一条查看详情。首先映入眼帘的是aspirin信息概览,包括PubChem CID、化合物结构、化学安全分类、分子式、同义词、分子量和数据更新时间,并提供aspirin药理功能注释和肝毒性信息,提供NCI Thesaurus、LiverTox和DrugBank数据库链接。右侧栏为该页面所展示的信息目录。
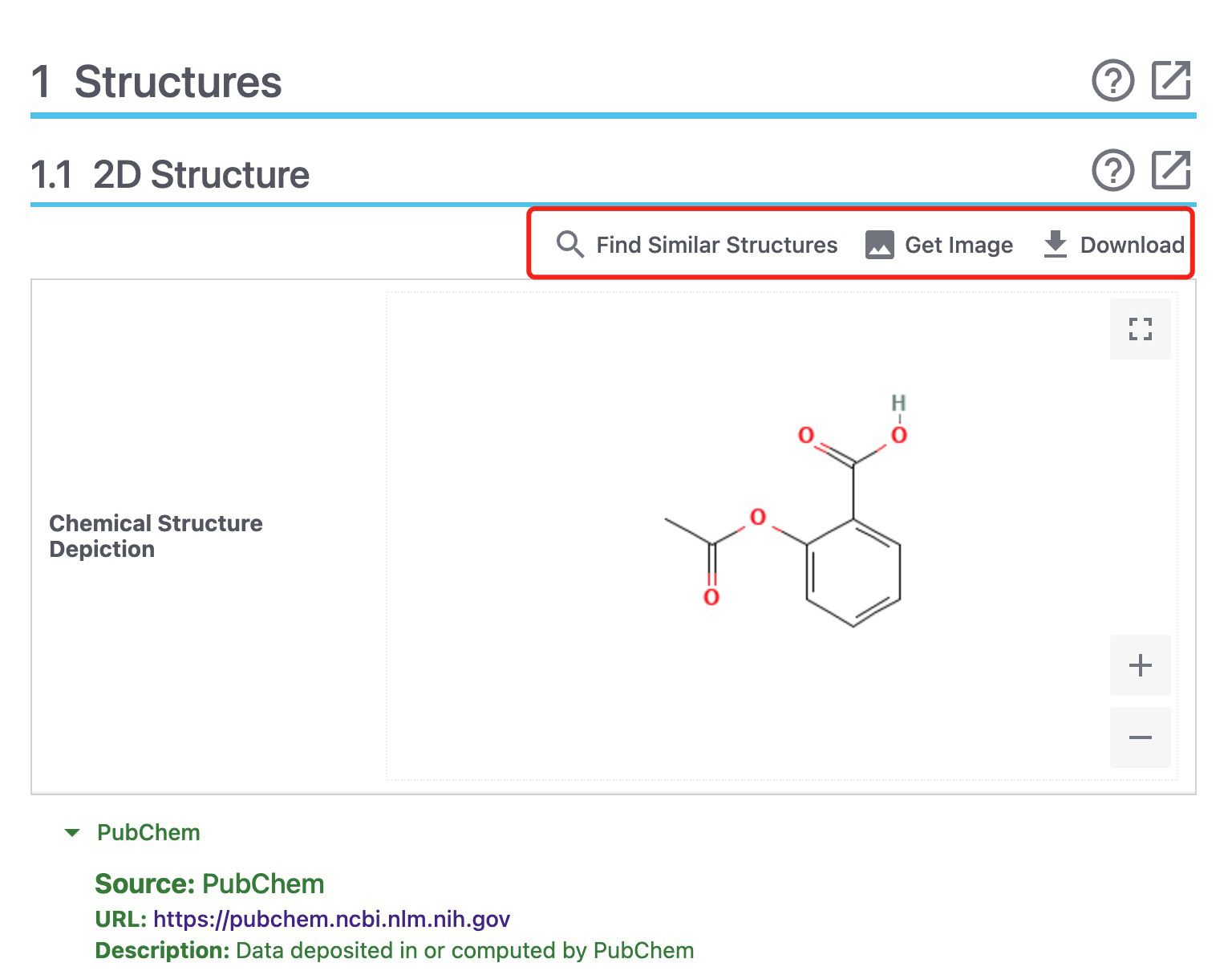
接下来介绍各部分详细信息:Structure部分展示aspirin的2D、3D和晶体结构,右上角可下载结构信息文件或保存图片,可在数据库检索结构类似的化合物。
Names and Identifiers部分展示aspirin的各种表达式、别名和标识符。IUPAC Name为2-乙酰氧基苯甲酸(2-acetyloxybenzoic acid),InChI=1S/C9H8O4/c1-6(10)13-8-5-3-2-4-7(8)9(11)12/h2-5H,1H3,(H,11,12),SMILES:CC(=O)OC1=CC=CC=C1C(=O)O
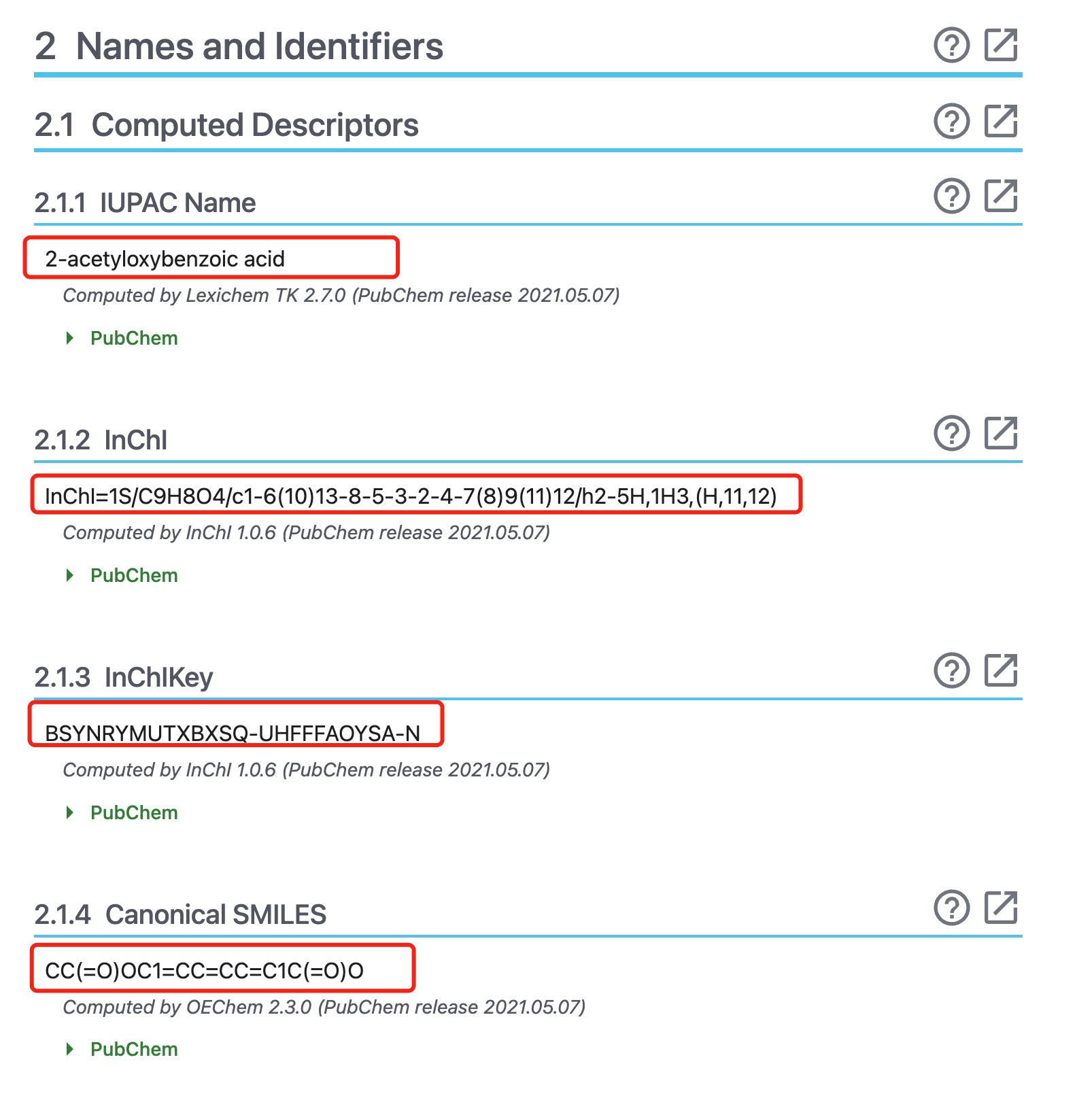
提供CAS、EC、ICSC和UNII等数据库ID,以及各种同义词。
Chemical and Physical Properties部分展示aspirin理化性质,比如溶解度。

接下来比较重要的一部分:Biomolecular Interactions and Pathways,展示aspirin的靶基因、靶蛋白及结构、通路、药物相互作用和药物食物相互作用等信息。
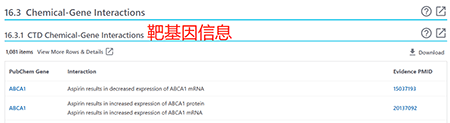
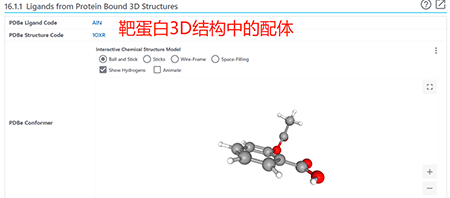
[外链图片转存失败,源站可能有防盗链机制,建议将图片保存下来直接上传(img-YodFMH2O-1685934661965)(https://gitee.com/bellacaoyh/pics/raw/master/img/202306051050652.png)]
[外链图片转存失败,源站可能有防盗链机制,建议将图片保存下来直接上传(img-2FqzT1pV-1685934661966)(assets/1627019887718177.png)]
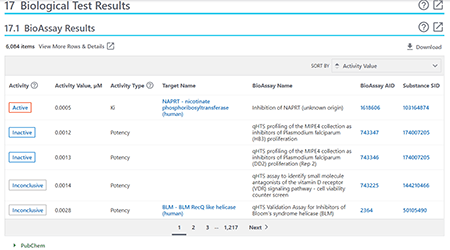
Biological Test Results部分展示药物活性信息。
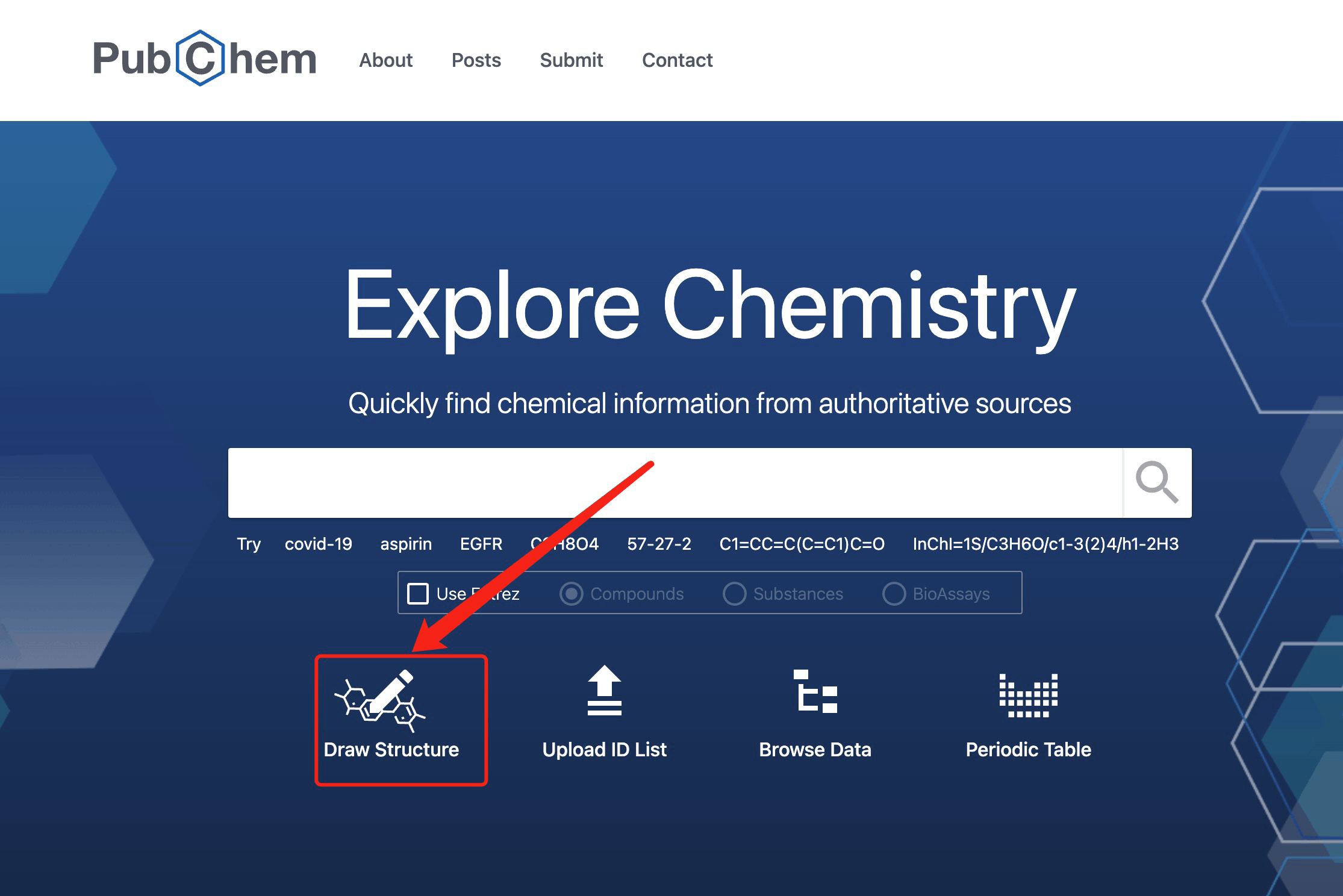
- 以结构式进行检索
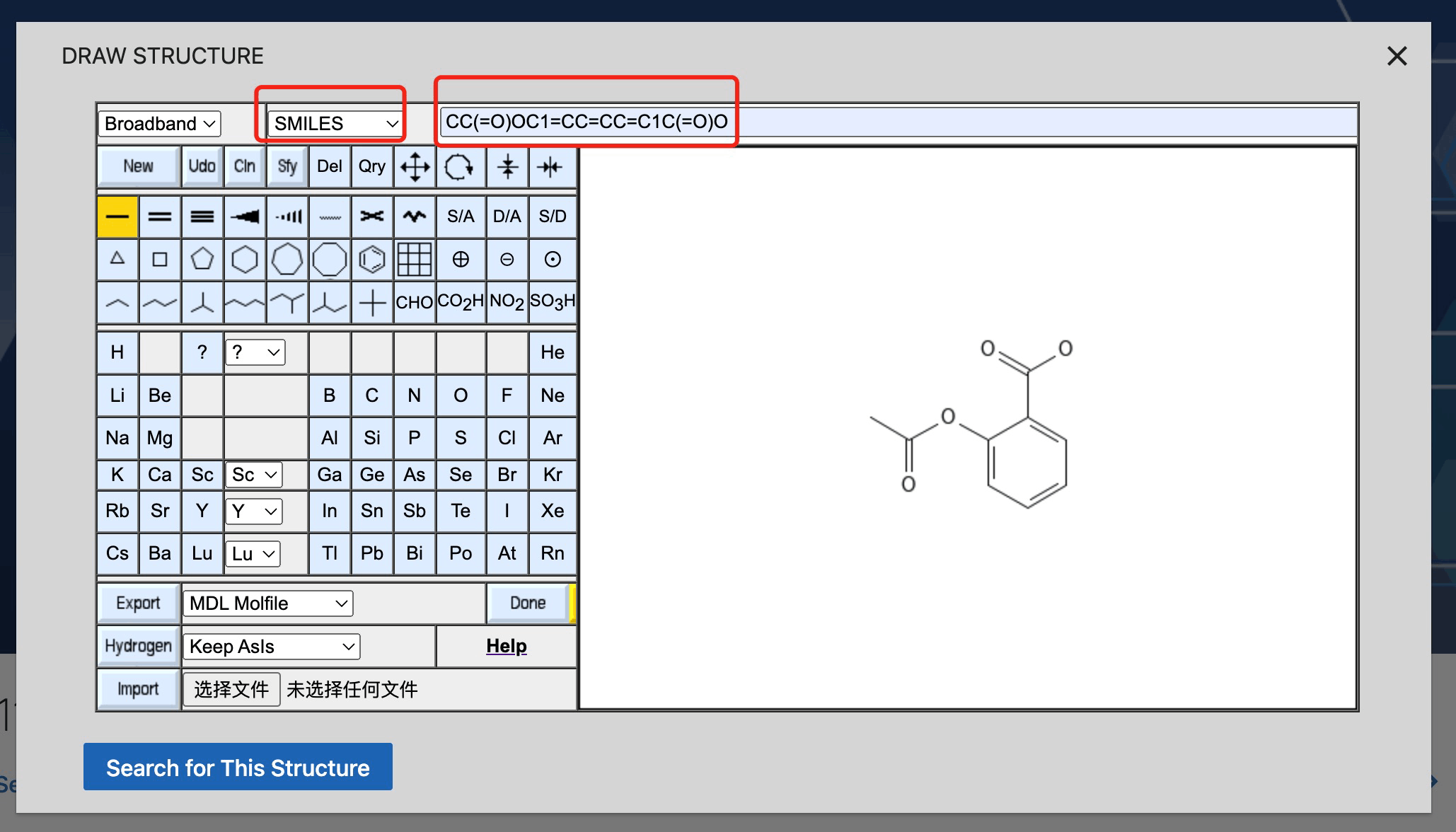
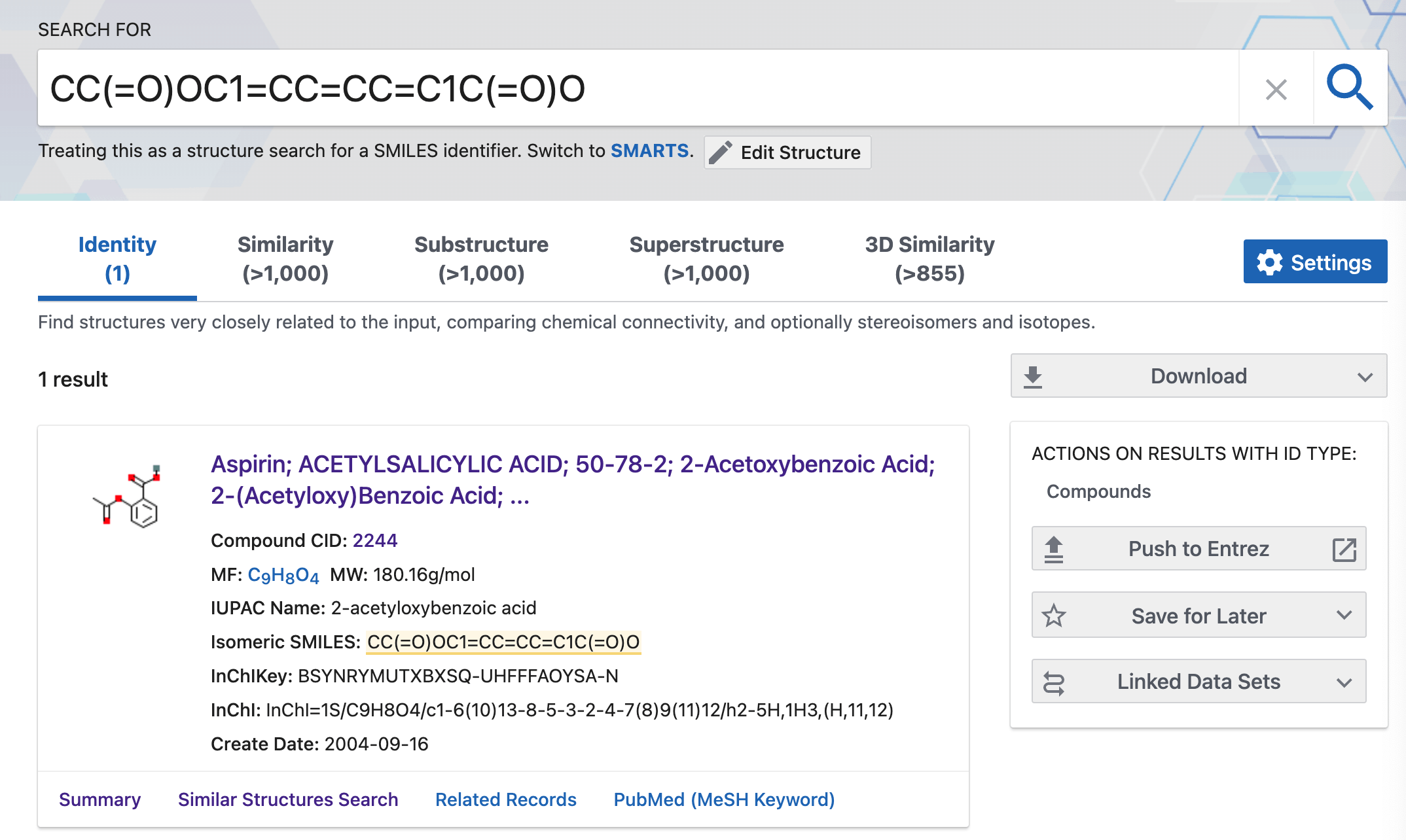
点击Draw Structure进入检索界面,以检索阿司匹林为例,输入其smiles表达式(CC(=O)OC1=CC=CC=C1C(=O)O),回车后自动绘制结构图。检索结果包括同一性、相似性、子结构和上层结构,进入各化合物详情页面,内容基本与前文类似。
数据打包下载
【官方下载链接】
PubChem PUG REST
【官网API说明】
构建 PUG REST 的基本单元是 PubChem 标识符,它具有三种类型——物质的 SID、化合物的 CID 和化验的 AID。使用这些标识符的该服务的概念框架是由三部分组成的请求:
- 输入——即我们在谈论什么标识符;
- 操作——如何处理这些标识符;
- 输出——应该返回什么信息。
这种设计的美妙之处在于请求的这三个部分中的每一个(大部分)都是独立的,允许组合扩展您可以在单个请求中执行的操作。这意味着,例如,指定某组 CID 的任何形式的输入都可以与处理 CID 的任何操作以及与所选操作相关的任何输出格式相结合。
[外链图片转存失败,源站可能有防盗链机制,建议将图片保存下来直接上传(img-JyX158JI-1685934661967)(assets/image-20230604205731947.png)]
设计URL
| https://pubchem.ncbi.nlm.nih.gov/rest/pug | /compound/name/vioxx | /property/InChI | /TXT |
|---|---|---|---|
| prolog | input | operation | output |
- prolog: 服务本身的 HTTP 地址,它对所有 PUG REST 请求都是通用的
- Input: 在本例中表示“我想在 PubChem 化合物数据库中查找与名称‘vioxx’匹配的记录。” 请注意,这里有一些微妙之处,因为名称必须已经存在于 PubChem 数据库中,并且一个名称可以引用多个 CID。但基本原则是我们根据名称指定一组 CID;在撰写本文时,只有一个 CID 使用此名称。
- operation: 在本例中是“我想检索此 CID 的 InChI 属性”。
- output : 最后是输出格式规范,“我想取回纯文本。”
j
| 输出格式 | 描述 |
|---|---|
| XML | 标准 XML,其模式可用 |
| JSON | JSON、JavaScript 对象表示法 |
| JSONP | JSONP,像 JSON 但包装在一个回调函数中 |
| ASNB | 标准二进制 ASN.1,在许多情况下是 NCBI 的本机格式 |
| ASNT | NCBI 的 ASN.1 人类可读文本风格 |
| SDF | 化学结构数据 |
| CSV文件 | 逗号分隔值,兼容电子表格 |
| PNG | 标准PNG图像数据 |
| TXT | 纯文本 |
URL中的特殊字符
大多数 PUG REST URL 都可以写成一个简单的 URL“路径”,其中元素由“/”字符分隔。但是一些输入,例如 SMILES(具有立体化学)和 InChI,包含“/”或其他与 URL 语法冲突的特殊字符。在这些情况下,PUG REST 可以将输入字段作为URL 编码的CGI 参数值(在 URL 中的“?”之后),使用出现在路径中的相同参数名称。例如,要使用 InChI 字符串
InChI=1S/C9H8O4/c1-6(10)13-8-5-3-2-4-7(8)9(11)12/h2-5H,1H3,(H,11,12)
作为输入,仅在 URL 的路径部分使用**/inchi/** ,并将参数 **inchi= (URL-encoded-string)**作为 CGI 参数:
https://pubchem.ncbi.nlm.nih.gov/rest/pug/compound/inchi/cids/JSON?inchi=InChI%3D1S%2FC9H8O4%2Fc1-6%2810%2913-8-5-3-2-4-7%288%299%2811%2912%2Fh2-5H%2C1H3%2C%28H%2C11%2C12%29
访问 substance 和 compound
| 方法 | 说明 | 举例 |
|---|---|---|
| 按标识符 | 直接指定 SID 或 CID | https://pubchem.ncbi.nlm.nih.gov/rest/pug/substance/sid/10000/synonyms/XML |
| 按名字 | 用名称来指代一种化学品(但是一种名称可能有多个匹配结果,也可以指定为检索结果的第一个,或者选择完全匹配检索) | https://pubchem.ncbi.nlm.nih.gov/rest/pug/compound/name/glucose/cids/TXT |
| 按结构标识 | 有许多方法可以使用 SMILES、InChI、InChI 键或 SDF 来按化学结构指定化合物,InChI 和 SDF 需要使用 POST(之后介绍),SMILES 字符串或 InChIkey进行指定非常简单 | https://pubchem.ncbi.nlm.nih.gov/rest/pug/compound/smiles/CCCC/cids/TXT |
| 按结构搜索 | PubChem 搜索方法的一些重新设计使得通过身份、相似性(2D 和 3D)、子结构和上层结构进行更快的搜索成为可能。这些方法是同步输入,这意味着不需要等待/轮询,因为在大多数情况下,它们将在一次调用中返回结果。(如果搜索过于广泛或复杂,则可能会超时。) | https://pubchem.ncbi.nlm.nih.gov/rest/pug/compound/fastidentity/cid/5793/cids/TXT?identity_type=same_connectivity |
访问记录选择
全记录
PUG REST 可用于检索整个记录,采用 PubChem 支持的常用格式
- ASN.1(NCBI 的本机格式)
- XML
- SDF
- JSON§
事实上,如果不指定其他操作,则全记录检索是默认操作
eg:
https://pubchem.ncbi.nlm.nih.gov/rest/pug/compound/cid/2244/SDF
https://pubchem.ncbi.nlm.nih.gov/rest/pug/compound/cid/2244/record/XML
图片
图像确实是全记录输出的一种风格,因为它们将结构描述为一个整体。因此,检索图像所需要做的就是指定 PNG 格式输出,而不是上一节中描述的其他数据格式之一。请注意,虽然图像请求只会显示输入标识符列表中的第一个 SID 或 CID,但目前无法在单个请求中获取多个图像。(但是, PubChem 的下载服务 可用于获取多个图像。)图像检索与所有各种输入法完全兼容,因此例如您可以使用它来获取化学名称、SMILES 字符串、InChI 键等的图像.:
eg:
https://pubchem.ncbi.nlm.nih.gov/rest/pug/compound/name/lipitor/PNG
https://pubchem.ncbi.nlm.nih.gov/rest/pug/compound/smiles/CCCCC=O/PNG
化合物属性
如果将以逗号分隔的属性标签列表写入 URL 中,则可以请求多个属性。属性表的有效输出格式为:
- XML
- ASNT/B
- JSON
- CSV
- TXT(仅限于单个属性)。
可用的属性包括:
| Property | Notes | 笔记 |
|---|---|---|
| MolecularFormula | Molecular formula. | 分子式。 |
| MolecularWeight | The molecular weight is the sum of all atomic weights of the constituent atoms in a compound, measured in g/mol. In the absence of explicit isotope labelling, averaged natural abundance is assumed. If an atom bears an explicit isotope label, 100% isotopic purity is assumed at this location. | 分子量是化合物中组成原子的所有原子量的总和,以克/摩尔为单位。在没有明确的同位素标记的情况下,假定平均自然丰度。如果原子带有明确的同位素标签,则假定该位置的同位素纯度为 100%。 |
| CanonicalSMILES | Canonical SMILES (Simplified Molecular Input Line Entry System) string. It is a unique SMILES string of a compound, generated by a “canonicalization” algorithm. | Canonical SMILES(简化分子输入行输入系统)字符串。它是化合物的唯一 SMILES 字符串,由“规范化”算法生成。 |
| IsomericSMILES | Isomeric SMILES string. It is a SMILES string with stereochemical and isotopic specifications. | 同分异构的SMILES字符串。它是具有立体化学和同位素规范的 SMILES 字符串。 |
| InChI | Standard IUPAC International Chemical Identifier (InChI). It does not allow for user selectable options in dealing with the stereochemistry and tautomer layers of the InChI string. | 标准 IUPAC 国际化学标识符 (InChI)。在处理 InChI 字符串的立体化学和互变异构层时,它不允许用户选择选项。 |
| InChIKey | Hashed version of the full standard InChI, consisting of 27 characters. | 完整标准 InChI 的散列版本,由 27 个字符组成。 |
| IUPACName | Chemical name systematically determined according to the IUPAC nomenclatures. | 根据 IUPAC 命名法系统地确定化学名称。 |
| Title | The title used for the compound summary page. | 用于化合物摘要页面的标题。 |
| XLogP | Computationally generated octanol-water partition coefficient or distribution coefficient. XLogP is used as a measure of hydrophilicity or hydrophobicity of a molecule. | 计算生成的辛醇-水分配系数或分配系数。XLogP用作分子亲水性或疏水性的量度。 |
| ExactMass | The mass of the most likely isotopic composition for a single molecule, corresponding to the most intense ion/molecule peak in a mass spectrum. | 单个分子最可能的同位素组成的质量,对应于质谱中最强烈的离子/分子峰。 |
| MonoisotopicMass | The mass of a molecule, calculated using the mass of the most abundant isotope of each element. | 分子的质量,使用每种元素最丰富的同位素的质量计算得出。 |
| TPSA | Topological polar surface area, computed by the algorithm described in the paper by Ertl et al. | 拓扑极地表面积,由Ertl 等人在论文中描述的算法计算得出。 |
| Complexity | The molecular complexity rating of a compound, computed using the Bertz/Hendrickson/Ihlenfeldt formula. | 化合物的分子复杂性评级,使用 Bertz/Hendrickson/Ihlenfeldt 公式计算。 |
| Charge | The total (or net) charge of a molecule. | 分子的总(或净)电荷。 |
| HBondDonorCount | Number of hydrogen-bond donors in the structure. | 结构中氢键供体的数量。 |
| HBondAcceptorCount | Number of hydrogen-bond acceptors in the structure. | 结构中氢键受体的数量。 |
| RotatableBondCount | Number of rotatable bonds. | 可轮换债券的数量。 |
| HeavyAtomCount | Number of non-hydrogen atoms. | 非氢原子数。 |
| IsotopeAtomCount | Number of atoms with enriched isotope(s) | 具有富集同位素的原子数 |
| AtomStereoCount | Total number of atoms with tetrahedral (sp3) stereo [e.g., ®- or (S)-configuration] | 具有四面体 (sp3) 立体结构的原子总数 [例如,®- 或 (S)- 配置] |
| DefinedAtomStereoCount | Number of atoms with defined tetrahedral (sp3) stereo. | 具有定义的四面体 (sp3) 立体结构的原子数。 |
| UndefinedAtomStereoCount | Number of atoms with undefined tetrahedral (sp3) stereo. | 具有未定义四面体 (sp3) 立体的原子数。 |
| BondStereoCount | Total number of bonds with planar (sp2) stereo [e.g., (E)- or (Z)-configuration]. | 具有平面 (sp2) 立体 [例如 (E)- 或 (Z)- 配置] 的键总数。 |
| DefinedBondStereoCount | Number of atoms with defined planar (sp2) stereo. | 具有定义的平面 (sp2) 立体的原子数。 |
| UndefinedBondStereoCount | Number of atoms with undefined planar (sp2) stereo. | 具有未定义平面 (sp2) 立体的原子数。 |
| CovalentUnitCount | Number of covalently bound units. | 共价结合单位的数量。 |
| PatentCount | Number of patent documents linked to this compound. | 与该化合物相关的专利文件数量。 |
| PatentFamilyCount | Number of unique patent families linked to this compound (e.g. patent documents grouped by family). | 与该化合物相关联的独特专利家族的数量(例如,按家族分组的专利文件)。 |
| LiteratureCount | Number of articles linked to this compound (by PubChem’s consolidated literature analysis). | 与该化合物相关的文章数量(通过 PubChem 的综合文献分析)。 |
| Volume3D | Analytic volume of the first diverse conformer (default conformer) for a compound. | 化合物的第一个不同构象异构体(默认构象异构体)的分析体积。 |
| XStericQuadrupole3D | The x component of the quadrupole moment (Qx) of the first diverse conformer (default conformer) for a compound. | 化合物的第一个不同构象异构体(默认构象异构体)的四极矩 (Qx) 的 x 分量。 |
| YStericQuadrupole3D | The y component of the quadrupole moment (Qy) of the first diverse conformer (default conformer) for a compound. | 化合物的第一个不同构象异构体(默认构象异构体)的四极矩 (Qy) 的 y 分量。 |
| ZStericQuadrupole3D | The z component of the quadrupole moment (Qz) of the first diverse conformer (default conformer) for a compound. | 化合物的第一个不同构象异构体(默认构象异构体)的四极矩 (Qz) 的 z 分量。 |
| FeatureCount3D | Total number of 3D features (the sum of FeatureAcceptorCount3D, FeatureDonorCount3D, FeatureAnionCount3D, FeatureCationCount3D, FeatureRingCount3D and FeatureHydrophobeCount3D) | 3D 特征总数(FeatureAcceptorCount3D、FeatureDonorCount3D、FeatureAnionCount3D、FeatureCationCount3D、FeatureRingCount3D 和 FeatureHydrophobeCount3D 的总和) |
| FeatureAcceptorCount3D | Number of hydrogen-bond acceptors of a conformer. | 构象异构体的氢键受体数量。 |
| FeatureDonorCount3D | Number of hydrogen-bond donors of a conformer. | 构象异构体的氢键供体数。 |
| FeatureAnionCount3D | Number of anionic centers (at pH 7) of a conformer. | 构象异构体的阴离子中心数(pH 7)。 |
| FeatureCationCount3D | Number of cationic centers (at pH 7) of a conformer. | 构象异构体的阳离子中心数(pH 7)。 |
| FeatureRingCount3D | Number of rings of a conformer. | 构象异构体的环数。 |
| FeatureHydrophobeCount3D | Number of hydrophobes of a conformer. | 构象异构体的疏水基数。 |
| ConformerModelRMSD3D | Conformer sampling RMSD in Å. | 以 Å 为单位的一致性采样 RMSD。 |
| EffectiveRotorCount3D | Total number of 3D features (the sum of FeatureAcceptorCount3D, FeatureDonorCount3D, FeatureAnionCount3D, FeatureCationCount3D, FeatureRingCount3D and FeatureHydrophobeCount3D) | 3D 特征总数(FeatureAcceptorCount3D、FeatureDonorCount3D、FeatureAnionCount3D、FeatureCationCount3D、FeatureRingCount3D 和 FeatureHydrophobeCount3D 的总和) |
| ConformerCount3D | The number of conformers in the conformer model for a compound. | 化合物构象模型中构象异构体的数量。 |
| Fingerprint2D | Base64-encoded PubChem Substructure Fingerprint of a molecule. | Base64 编码的 PubChem 子结构分子指纹。 |
eg:
- 单一化合物的单一属性:https://pubchem.ncbi.nlm.nih.gov/rest/pug/compound/cid/2244/property/MolecularWeight/TXT
- 多个化合物和多个属性的列表:https://pubchem.ncbi.nlm.nih.gov/rest/pug/compound/cid/1,2,3,4,5/property/MolecularWeight,MolecularFormula,HBondDonorCount,HBondAcceptorCount,InChIKey,InChI/CSV
PubChemPy
PubChemPy 完全依赖于通过其 PUG REST Web 服务提供的 PubChem 数据库和化学工具包。该服务为程序提供了一个界面,可以自动执行您可能通过PubChem 网站手动执行的任务。
使用 PubChemPy 时要记住这一点很重要:您发出的每个请求都会传输到 PubChem 服务器,进行评估,然后发回响应。这有一些缺点:它不太适合机密工作,它需要持续的互联网连接,而且有些任务会比在您自己的计算机上本地执行要慢。另一方面,这意味着我们拥有 PubChem 数据库和化学工具包的大量资源供我们使用。因此,可以在几秒钟内对包含数千万种化合物的数据库进行复杂的相似性和子结构搜索,而无需您本地计算机上的任何存储空间或计算能力。
安装
pip install pubchempy
引用
import pubchempy as pcp
Compound
>>> c = pcp.Compound.from_cid(962)
>>> c.to_dict(properties=['atoms', 'bonds', 'inchi'])
{'atoms': [{'aid': 1, 'element': 'o', 'x': 2.5369, 'y': -0.155},{'aid': 2, 'element': 'h', 'x': 3.0739, 'y': 0.155},{'aid': 3, 'element': 'h', 'x': 2, 'y': 0.155}],'bonds': [{'aid1': 1, 'aid2': 2, 'order': 'single'},{'aid1': 1, 'aid2': 3, 'order': 'single'}],'inchi': u'InChI=1S/H2O/h1H2'}
>>> c.inchi
'InChI=1S/H2O/h1H2'
对应的API:
| API | 描述 |
|---|---|
| elements | List of element symbols for atoms in this Compound. |
| atoms | List of Atoms in this Compound. |
| bonds | List of Bonds between Atoms in this Compound. |
| synonyms | A ranked list of all the names associated with this Compound. |
| sids | Requires an extra request. Result is cached. |
| aids | Requires an extra request. Result is cached. |
| charge | Formal charge on this Compound. |
| molecular_formula | |
| molecular_weight | |
| canonical_smiles | Canonical SMILES, with no stereochemistry information. |
| isomeric_smiles | Isomeric SMILES. |
| inchi | |
| inchikey | |
| iupac_name | Preferred IUPAC name. |
| xlogp | |
| exact_mass | |
| monoisotopic_mass | 单一同位素质谱 |
| tpsa | Topological Polar Surface Area. 拓扑极地表面积 |
| fingerprint | Raw padded and hex-encoded fingerprint 原始的填充和十六进制的指纹 |
| cactvs_fingerprint | PubChem CACTVS fingerprint. Each bit in the fingerprint represents the presence or absence of one of 881 chemical substructures. |
| heavy_atom_count | |
| isotope_atom_count | |
| atom_stereo_count | |
| defined_atom_stereo_count | Defined atom stereocenter count. |
| undefined_atom_stereo_count | |
| bond_stereo_count | |
| defined_bond_stereo_count | |
| undefined_bond_stereo_count | |
| covalent_unit_count | Covalently-bonded unit count. 共价键单元数 |
Substance
>>> substance = pcp.Substance.from_sid(223766453)
>>> substance.synonyms
['2-(Acetyloxy)-benzoic acid', '2-(acetyloxy)benzoic acid', '2-acetoxy benzoic acid', '2-acetoxy-benzoic acid', '2-acetoxybenzoic acid', '2-acetyloxybenzoic acid', 'BSYNRYMUTXBXSQ-UHFFFAOYSA-N', 'acetoxybenzoic acid', 'acetyl salicylic acid', 'acetyl-salicylic acid', 'acetylsalicylic acid', 'aspirin', 'o-acetoxybenzoic acid']
>>> print substance.source_id
BSYNRYMUTXBXSQ-UHFFFAOYSA-N
>>> substance.standardized_cid
2244
>>> substance.standardized_compound
Compound(2244)

Searching
- 通过 name 搜索
>>> results = pcp.get_compounds('Glucose', 'name')
>>> results
[Compound(79025), Compound(5793), Compound(64689), Compound(206)]
- 通过 smiles 搜索
>>> pcp.get_compounds('C1=CC2=C(C3=C(C=CC=N3)C=C2)N=C1', 'smiles')
[Compound(1318)
- API说明

pandas 集成
获取化合物、获取物质和获取属性都可以返回一个pandas DataFrame
df1 = pcp.get_compounds('C20H41Br', 'formula', as_dataframe=True) #返回符合条件的所有化合物的所有属性
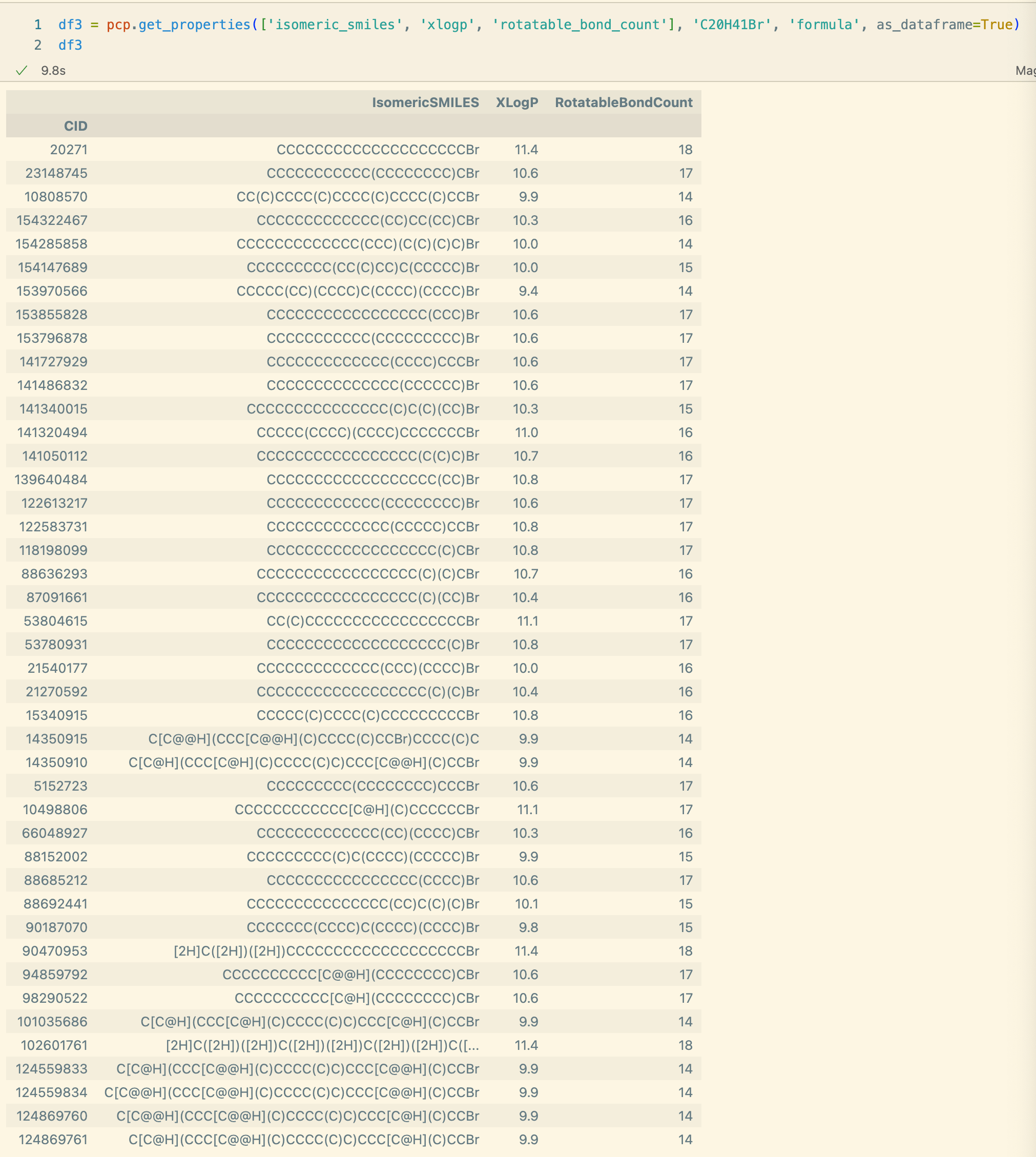
df2 = pcp.get_substances([1, 2, 3, 4], as_dataframe=True) #
df3 = pcp.get_properties(['isomeric_smiles', 'xlogp', 'rotatable_bond_count'], 'C20H41Br', 'formula', as_dataframe=True) # 返回满足条件的指定属性