序列组装
一、摘要
- 加深序列组装原理的理解;
- 熟悉已有的基因组序列组装软件的使用;
- 掌握常用的序列组装软件的使用;
- 能够独立地使用自己所掌握的编程语言编写序列组装的简化程序。
二、材料和方法
1、硬件平台
处理器:Intel(R) Core(TM)i7-4710MQ CPU @ 2.50GHz
安装内存(RAM):16.0GB
2、系统平台
Windows 8.1、Ubuntu
3、软件平台
【1】AllPathsLG
【2】SOAP
【3】GenomeABC http://crdd.osdd.net/raghava/genomeabc/
【4】Python3.5
【5】Biopython
4、数据库资源
NCBI数据库:https://www.ncbi.nlm.nih.gov/
5、研究对象
噬菌体基因组Dickeya phage RC-2014
https://www.ncbi.nlm.nih.gov/genome/?term=NC_025452
6、方法
序列组装软件的概览
进入pubmed搜索序列组装相关的文献,仔细阅读并尝试安装、使用其中的组装软件。
待拼接序列数据源的准备
使用实验1的模拟测序程序,生成待拼接序列数据源
小规模序列拼接软件的使用练习
GenomeABC 在线平台,对第2 步获得的数据源进行拼接和评估。
大规模基因组序列拼接软件的使用练习
练习序列拼接软件AllPathsLG、soapdenovo2、Velvet
序列组装程序的编写
下载安装python,并且安装biopython扩展模块,编写程序,对测序结果进行组装
不同组装结果的对比
把上述软件和自行编程得到的拼接结果进行比较,分析它们之间的覆盖率,以及contigs 的差异等
三、结果
1、序列组装软件
生物信息学中的一个重要问题就是基因组的组装,NGS技术产生了大量的基因组序列的片段,随之而来的是需要大量内存空间的基因组组装。伴随着最近测序技术的进步,可以预见的是内存空间占用将会急剧增加,并且会成为处理基因组测序数据的限制因素。
常见的序列组装软件有:
SSAKE:将short reads严格地聚类组装成contigs
Ray:主要用于组装Illumina, 454, SOLiD测序生成的reads
a5-miseq:生成高质量的细菌基因组组装。
Orione:是一个包含公共的软件和pipline的框架,针对微生物测序的组装
SGA:基于线性图的组装软件,由于使用压缩表示DNA测序reads的方法,节约内存。

图表 1相关文献

图表 2汇总组装软件的网站
文献链接:https://www.ncbi.nlm.nih.gov/pubmed/24086547
网站链接:https://omictools.com/genome-assembly-category
PS:其他常见软件如AllPathsLG、soapdenovo2、Velvet,此网站也有涉及,由于下面将要具体应用,此处不一一详述。
这里以安装SOAP为例
安装SOAP,首先unzip解压

图表 3解压缩
然后进行make进行编译

图表 4编译
最后,配置环境变量。
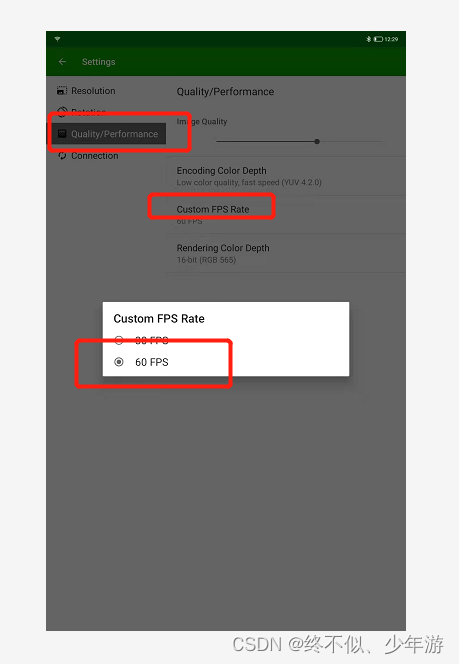
图表 5配置环境变量
测试其运行情况

图表 6程序测试
2、待拼接序列数据源的准备
使用之前模拟测序的程序,进行噬菌体基因组的模拟测序,由于后面做的是编程分支,python程序效率有限,因此这里N值仅仅为50,覆盖率为0.7251

图表 7模拟测序
3、小规模序列拼接软件的使用练习
选择模拟测序生成的virusSingleRead.fa文件,上传并且运行

图表 8上传测序文件

图表 9 GenomeABC结果下载
4、大规模基因组序列拼接软件的使用练习
首先,下载安装AllPaths-LG,并且下载测试数据。
接着./configure配置

图表 10配置
make命令,进行gcc编译,此步骤耗时很长

图表 11编译
make install进行安装

图表 12安装
使用测试数据进行组装准备,生成data文件夹

图表 13组装准备脚本

图表 14组装准备
接着进行组装

图表 15组装脚本

图表 16运行组装
组装结果,data文件夹下面都是运行的结果,总计拼出来一条scaffold

5、编程模拟组装结果
计算reads间的overlap

拼装生成Contig

测序覆盖度(m值)1.29,噬菌体基因组总长度:155.346kbp
拼接出的contig总长度:188.754kbp
拼接覆盖度:1.22%
6、不同组装结果的对比
特点:
自己编写的程序运行效率会低得多,但是由于之前模拟测序并没有引入测序错误,所以拼接出来的contig全都能完全匹配到原来的基因组。
AllPaths-LG运行速度相对较快,开源软件准确率有保证,但是运行结果有一大堆,如果在实际中,需要仔细阅读相关的介绍文档,才能正确使用
四、讨论和结论
程序运行方法
需要调整文件句柄参数,分别是读入的测序文件句柄、输出的组装文件句柄、读入进行后续比较的参考基因组文件句柄。
在is_overlap 方法中调整minoverlaplength,进而调整Reads间的最小overlap值
附录
from Bio import SeqIO
from Bio.SeqRecord import SeqRecordreadsList = []
for seq_record in SeqIO.parse("data/virusSingleRead.fa", "fasta"):readsList.append(seq_record)# 过滤掉互相包含的reads
readsfilterList = []
for i in range(len(readsList)):j = 0flag = Truewhile j < len(readsList):# noinspection PyProtectedMemberif i != j and readsList[i].seq in readsList[j].seq:flag = False# print(readsList[i].id + "包含于" + readsList[j].id)j += 1if flag:readsfilterList.append(readsList[i])del readsList# noinspection PyProtectedMember
# 分别比较两字符串开头、结尾,计算overlap的大小;最后返回最大的overlap值
def is_overlap(id1, id2):minoverlaplength = 10score1 = score2 = 0reads1 = readsfilterList[id1]reads2 = readsfilterList[id2]seq1 = reads1.seqseq2 = reads2.seqminlength = len(seq1) if len(seq1) < len(seq2) else len(seq2)for i in range(minoverlaplength, minlength):if seq1[-i:] == seq2[:i]:print(reads1.id + "\t" + reads2.id + "\t" + str(i) + "\tafter")score1 = ifor j in range(minoverlaplength, minlength):if seq1[:j] == seq2[-j:]:print(reads1.id + "\t" + reads2.id + "\t" + str(j) + "\tbefore")score2 = jif score1 == score2 == 0:return id1, id2, 0, "NoOverlap"if score1 > score2:return id1, id2, score1, "After"else:return id1, id2, score2, "Before"# noinspection PyShadowingNames
# 构造边的类,存储:有overlap关系的两个reads在readsfilterList中的序号;
# score值(overlap的程度);tips作为辅助;记录哪儿个reads在前,哪儿个reads在后
# PS:tips其实最后没有用到,存储时候就已经前后排序过
class Edge:def __init__(self, id1, id2, score, tips):self.id1 = id1self.id2 = id2self.score = scoreself.tips = tipsdef __str__(self):return str(self.id1) + " " + str(self.id2) + " " + str(self.score) + " " + self.tipsdef show(self):print(self)edgeList = []
for i in range(len(readsfilterList)):j = i + 1while j < len(readsfilterList):# print(readsfilterList[i].id + " " + readsfilterList[j].id)(id1, id2, score, tips) = is_overlap(i, j)if tips != "NoOverlap":# print(str(id1) + " " + str(id2) + " " + str(score) + " " + tips)if tips == "Before":edgeList.append(Edge(id2, id1, score, tips))if tips == "After":edgeList.append(Edge(id1, id2, score, tips))# edgeList.append(Edge(id1, id2, score, tips))j += 1
edgeList = sorted(edgeList, key=lambda x: x.score, reverse=True)# 将edge边组装成一条条路径
pathList = [[edgeList.pop(0)]]# 将edge按序号排列好
for edge in edgeList:existflag = Truefor path in pathList:if edge.id1 == path[-1].id2 and edge.tips == "After":path.append(edge)existflag = Falsebreakif edge.id2 == path[0].id1 and edge.tips == "Before":path.insert(0, edge)existflag = Falsebreakif existflag:pathList.append([edge])# 根据一条条路径,将reads拼到一起,生成contig
contigList = []
ContigID = 1
for path in pathList:contig = readsfilterList[path[0].id1].seqfor edge in path:contig = contig + readsfilterList[edge.id2].seq[edge.score:]contigList.append(SeqRecord(contig, id="Contig" + str(ContigID), description=""))print("Contig" + str(ContigID))print(contig)ContigID += 1
SeqIO.write(contigList, "result/virusResult.fa", "fasta")# 对生成的contigs进行测试
record_iterator = SeqIO.parse("data/NC_025452.fasta", "fasta")
chromosome = record_iterator.__next__()
contigLength = 0
for contig in contigList:flag = str(contigList[0].seq) in str(chromosome.seq)if flag:contigLength += len(contig)# print(contig.id+" in "+chromosome.id)else:print(contig.id + " not in " + chromosome.id)
print("基因组总长度:" + str(len(chromosome) / 1000) + "kbp")
print("contig总长度:" + str(contigLength / 1000) + "kbp")
print("覆盖度:" + str(round(contigLength / len(chromosome), 2)) + "%")