说在前面
不论是做药还是想发高分文章,神经退行性疾病一直是个难题,神经元死亡、异常蛋白累积这些是很复杂的东西。而且认知功能的衰退是整个大脑网络的结果,因此只挑单个区域队列数据做早期标志物筛选这种,emm也能做,就是还可以更好点。因此做阿尔兹海默症想做好点的,多队列+多组学应该是最低标准了
今天给大家分享一篇JCR一区,多组学的文章:Brain high-throughput multi-omics data reveal molecular heterogeneity in Alzheimer’s disease

- 标题:大脑高通量多组学数据揭示阿尔茨海默病的分子异质性
- 期刊名称:PLoS Biology
- 影响因子:9.8
- JCR分区:Q1
- 中科院分区:生物学1区 Top
- 小类:生化与分子生物学1区 生物学1区
摘要
我们采用机器学习方法,将多个人类AD队列的高通量转录组、蛋白组、代谢组和脂质组数据与临床和神经病理学数据进行整合,发现了4种独特的多模态分子谱。其中一种显示出认知功能差、疾病进展速度快、生存时间短、严重神经退行性变和星形胶质增生,以及代谢组谱水平降低的迹象。我们发现这种分子谱存在于与更高的Braak tau评分相关的多个受影响的皮层区域,以及与突触相关基因、内吞作用、吞噬体和在AD早期和晚期阶段发生改变的mTOR信号通路的显著失调有关。通过将AD跨组学数据与SNCA小鼠模型的转录组数据整合,我们发现了一种重叠的特征。此外,我们利用单核RNA测序数据识别了最有可能介导分子谱的不同细胞类型。最后,我们确定了揭示AD进展和可能认知的多模态聚类所需的脑脊液生物标志物。我们的跨组学分析为AD提供了新颖且关键的分子洞见。
结果
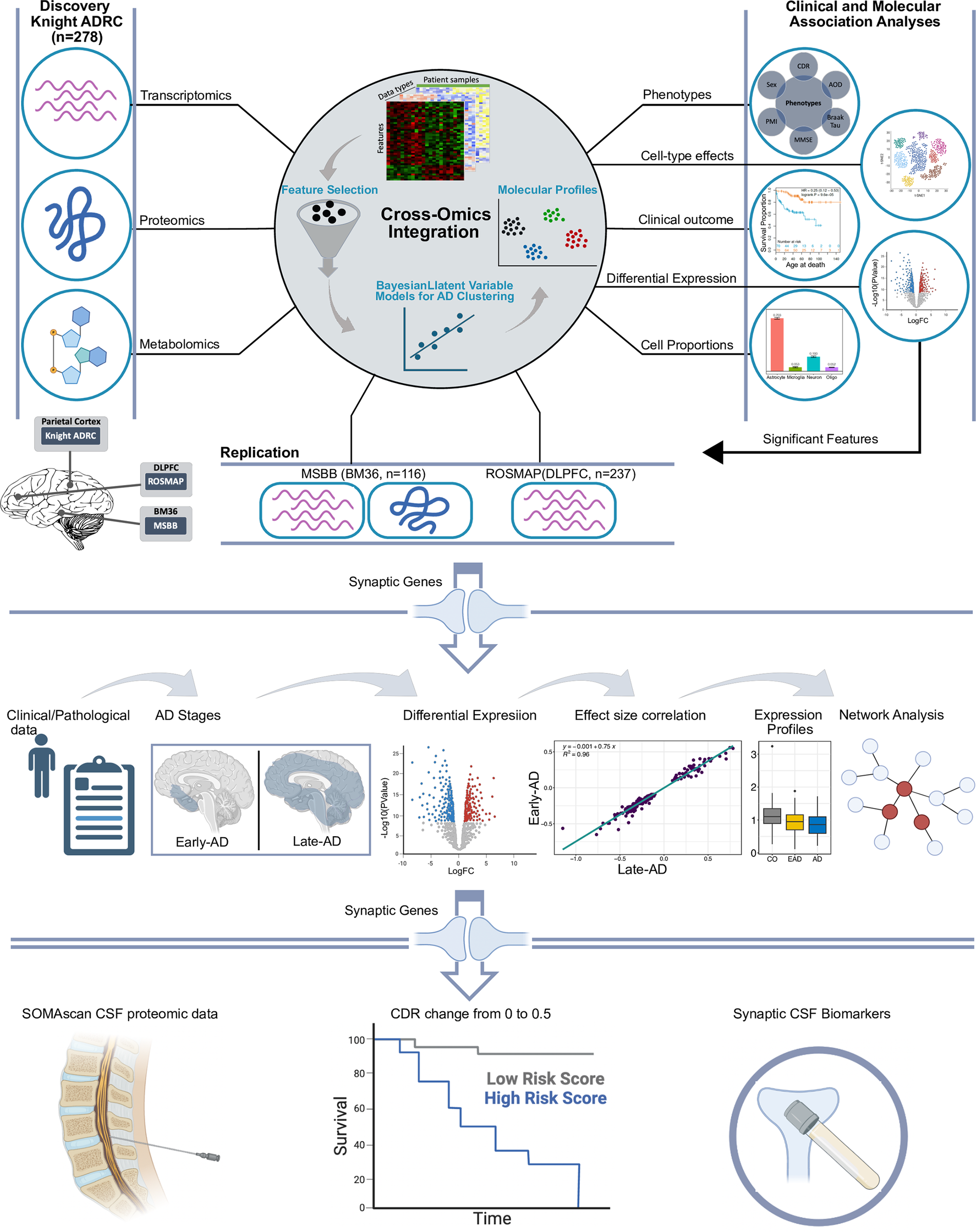
图1. 研究设计。
- 在这项研究中,我们使用了Knight ADRC参与者的逝世后顶叶皮层样本的转录组学、蛋白组学、代谢组学和脂质组学谱。三种组学共享了278个受试者(255例散发性AD和23例对照组)。为同一组样本(n = 278)和不同数量的特征(特征 = 60,754,蛋白质 = 1,092,代谢物 = 627)准备了表达/读取矩阵。
- 在整合之前,对所有特征数超过1,000的组学数据集进行特征选择,选择最顶级和最变异的特征。然后采用贝叶斯整合聚类方法将三种组学数据集整合在一起,并根据最大偏差比和最小BIC提取最佳聚类解决方案。接下来,将AD分子谱与多个临床和分子属性相关联,以检查是否存在与这些属性的关联。
- 此外,对这些分子谱进行了生存、差异表达、特定细胞类型效应和细胞比例推断分析。然后在来自MSBB BM36(AD = 93,CO = 23)和ROSMAP(AD = 144,CO = 93)的2个独立数据集中复制了这些分子谱。提取Knight-C4分子谱中的失调突触基因,并用于下游AD分期分析和在ROSMAP队列(早期AD对照组和晚期AD对照组)中。
- 通过生存分析确定出现在CSF SOMA数据集中的常见突触基因与痴呆风险增加相关,并将其确定为AD分期的CSF突触生物标志物。
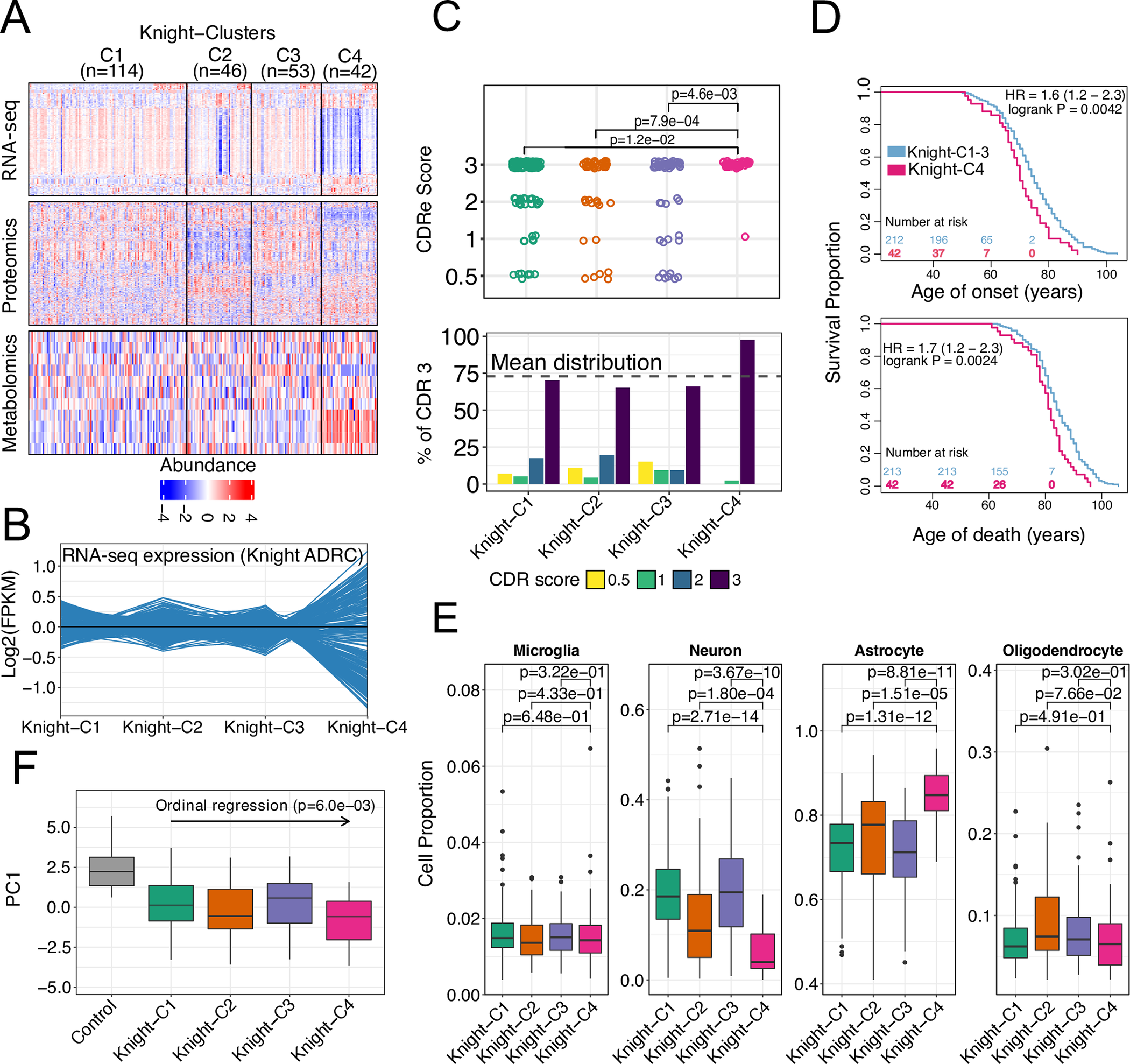
图2. 跨组学数据整合确定了4种与AD相关的不同分子谱,与更糟的临床结果和分子属性相关联。
- (A) 多组学特征的热图,显示了顶级显著特征的分布。
- (B) Top基因的转录组学谱(已标准化),显示对Knight-C4的整体失调。
- © 不同4个聚类中CDR评分的分布,Knight-C4与高CDR评分相关。
- (D) Kaplan–Meier曲线显示Knight-C4与发病年龄和死亡的不良结果相关联。
- (E) 使用从4种细胞类型进行的大规模RNA测序的反演分析,显示了4个聚类中的细胞比例。Knight-C4与明显较高和较低的星形胶质细胞和神经元比例相关。
- (F) 显示代谢组学谱第一主成分的箱线图,Knight-C2和Knight-C4显示比其他AD病例更低的代谢组特征。面板C、E和F下方的数据可在S1数据中找到。
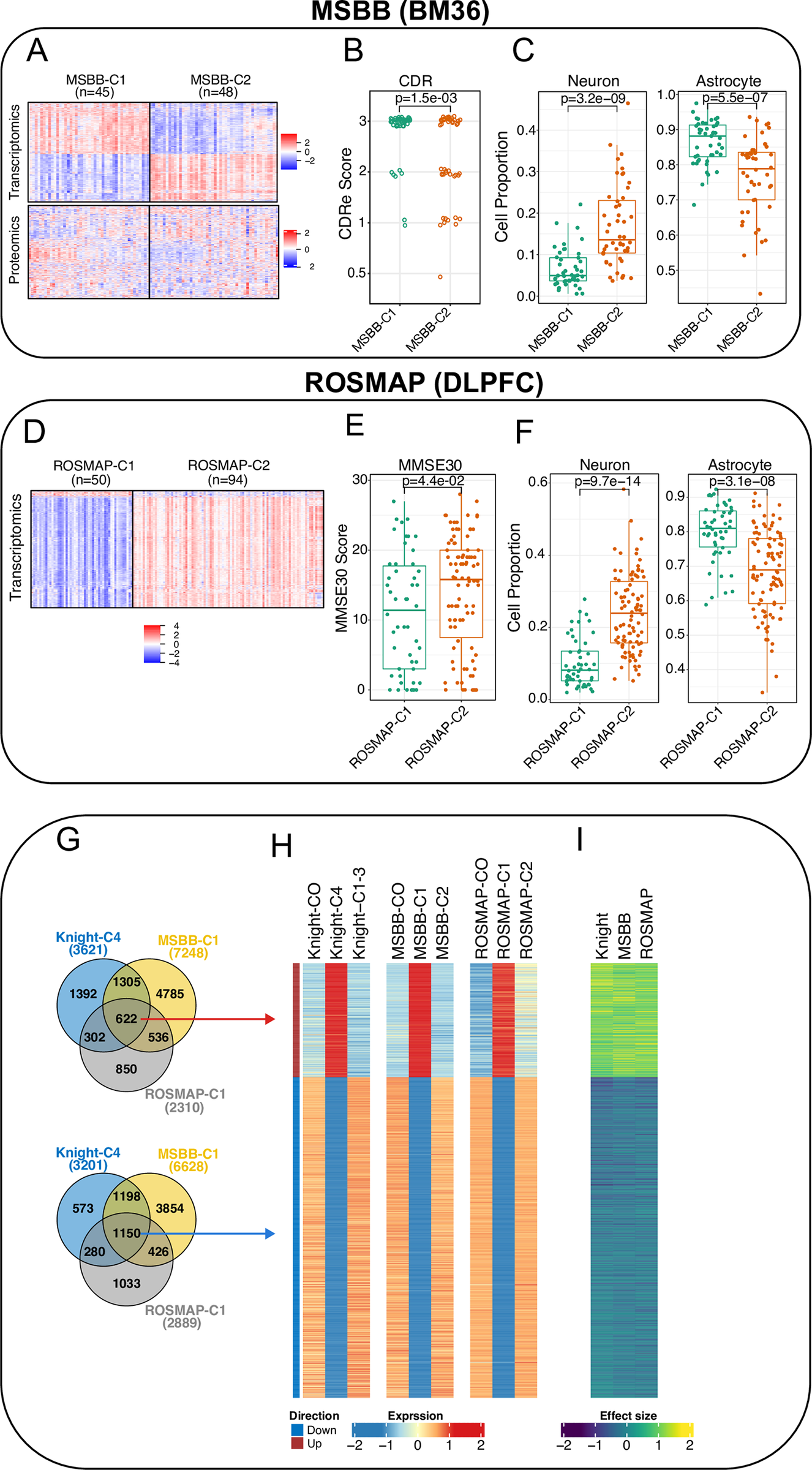
图3. Knight-C4的分子谱在2个独立数据集中得到复制。
- (A) 来自MSBB(BM36)的转录组学和蛋白组学谱的顶级特征的热图显示了2个不同的聚类。
- (B) 箱线图显示MSBB-C1与更高的CDR评分相关联,复制了Knight-C4在Knight ADRC中的结果。
- © 通过数字反演分析从MSBB(BM36)的大规模RNA测序数据中推断的细胞比例。MSBB-C1通过与明显更高和更低比例的星形胶质细胞和神经元相关,复制了Knight-C4的结果。
- (D) 来自ROSMAP(DLPFC)的顶级特征的转录组学谱的热图显示了2个不同的聚类。
- (E) 箱线图显示ROSMAP-C1与较低的MMSE30评分相关联。
- (F) 通过数字反演分析从ROSMAP(DLPFC)的大规模RNA测序数据中估计的细胞比例。与MSBB类似,ROSMAP-C1通过与明显更高和更低比例的星形胶质细胞和神经元相关,复制了Knight-C4的结果。
- (G) Venn图显示在发现和复制队列中的共同失调基因。
- (H) 显示在发现和复制队列中共享基因的平均表达的热图,显示了一致的表达模式。
- (I) 显示在发现和复制队列中检测到的效应大小的热图,显示了3个队列之间高效应大小相似性。

图4. 认知功能更差的参与者顶叶皮层显示出显著的分子失调。
- (A) 条形图显示每个聚类中检测到的显著基因与对照组的比较。黄色代表总基因数,绿色代表聚类特异基因,紫色表示在未聚类方法(所有AD病例 vs. 对照组)中遗漏的基因数。
- (B) 与“A”面板相同,但是针对蛋白质。
- © 与“A”面板相同,但是针对代谢物。
- (D) 环形图显示每个聚类与其他聚类之间显著特征的百分比,对于这3种组学。
- (E) Venn图显示Knight-C4中的显著基因与Fei等人的突触基因(“突触体”)之间的重叠。
- (F) 与“E”相同,但与SynGO数据集的重叠。重叠的显著性使用Fisher精确检验进行计算。
- (G) 与Knight-C4相关的前20个KEGG通路。
- (H) 与Knight-C4相关的前20个GO生物过程通路。
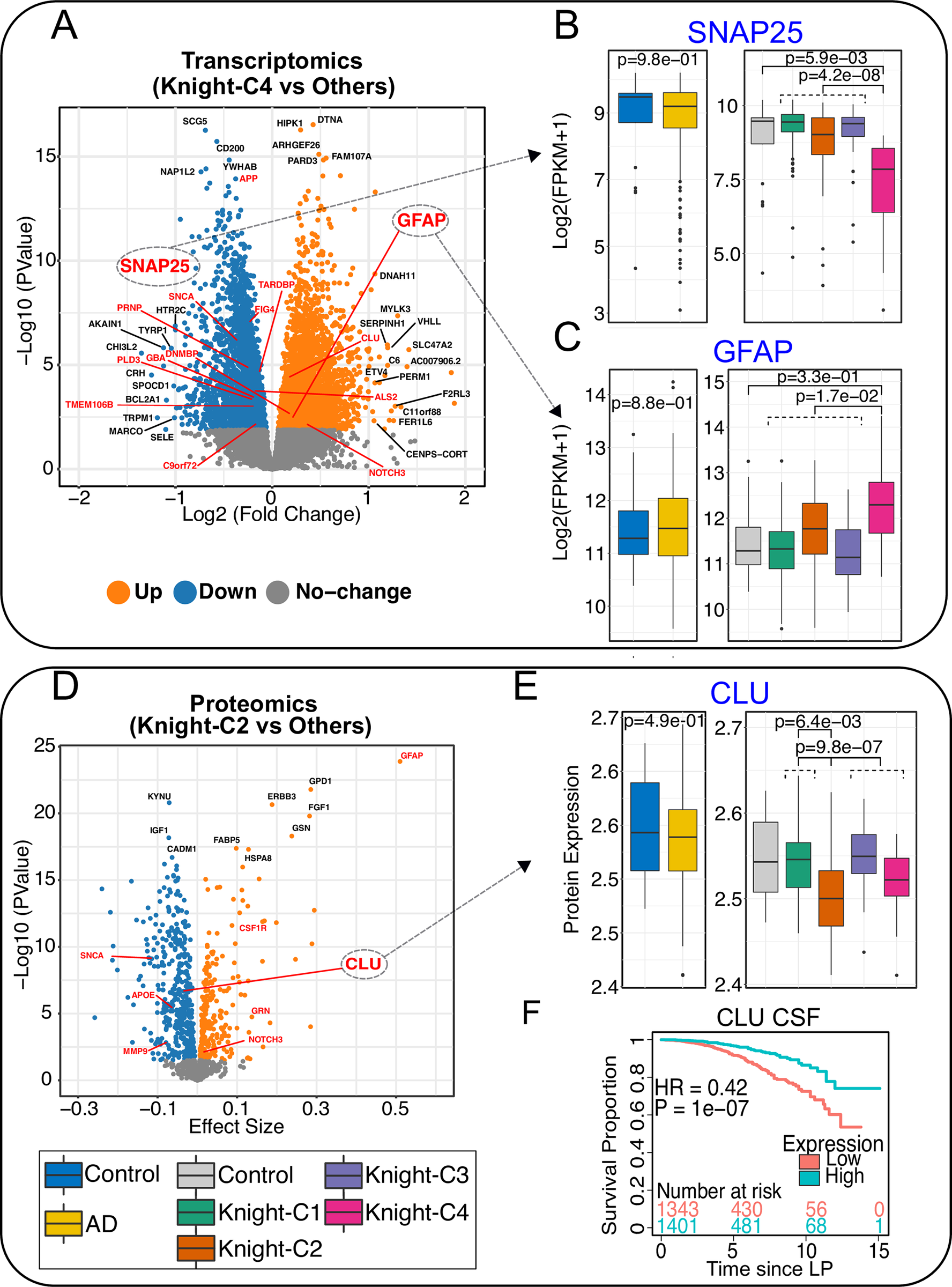
图5. 差异表达分析确定了与不同聚类相关的ND基因。
- (A) 显示Knight-C4与其他聚类之间上调和下调基因的火山图。红色基因是已知ND基因的示例。黑色基因是基于调整后的p值和log2(fold change) > 1的前10个显著基因。
- (B) 箱线图显示Knight ADRC队列中4个聚类(右侧)以及所有AD病例(左侧)的SNAP25转录组学谱(FPKM)。
- © 与“B”相同,但是针对GFAP。
- (D) 显示Knight-C2与其他聚类之间上调和下调蛋白质的火山图。
- (E) 箱线图显示Knight ADRC队列中4个聚类(右侧)以及所有AD病例(左侧)的CLU蛋白组学谱。
- (F) Kaplan–Meier曲线显示了在Knight ADRC参与者的CSF数据集中,Clusterin (ApoJ)蛋白浓度与痴呆进展风险增加(以CDR从0到0.5变化)的关联。
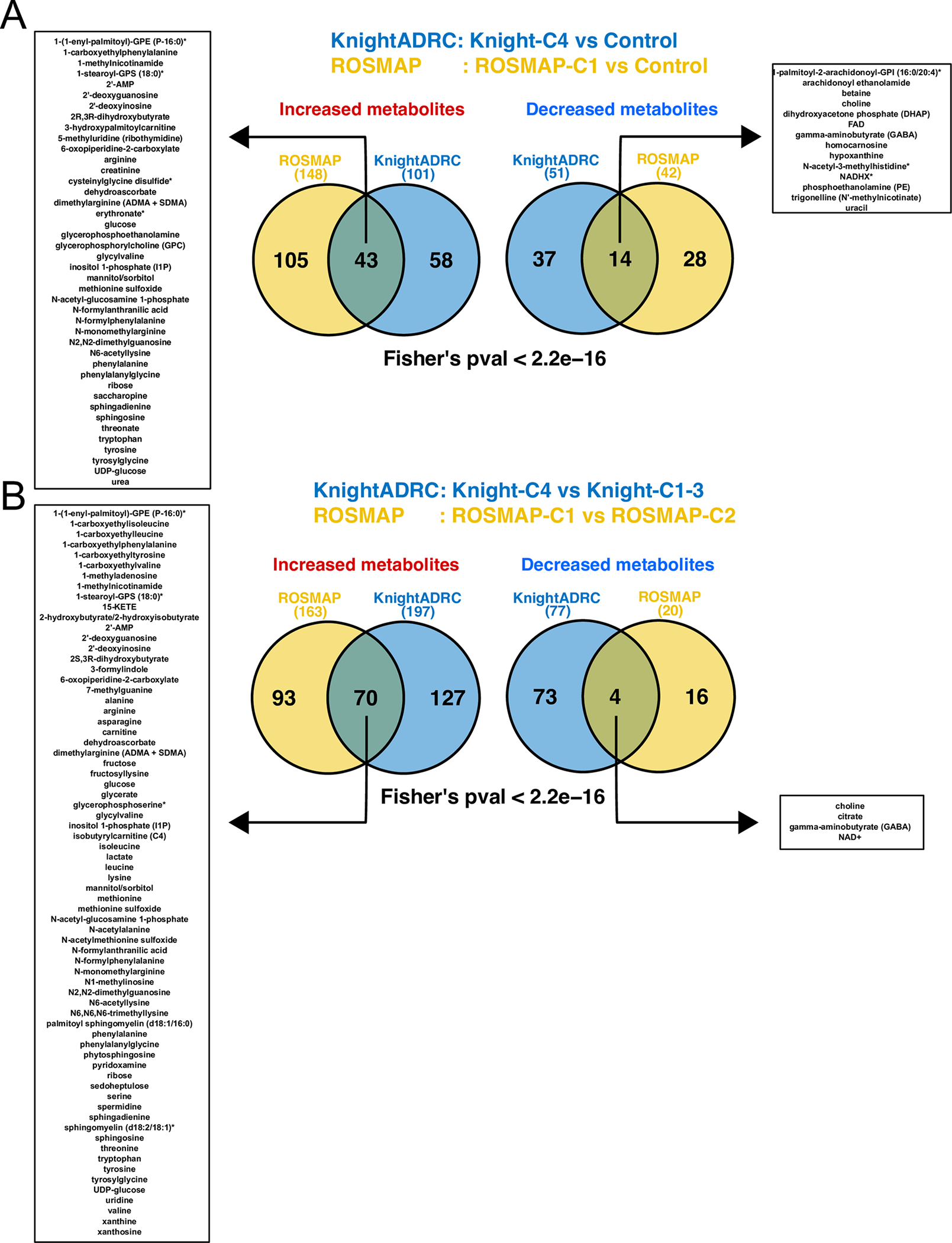
图6. 差异丰度分析确定了与Knight-C4相关的显著代谢物和通路。
- (A) Venn图显示与对照组相比,Knight-C4和ROSMAP-C1之间显著增加和减少的代谢物。左侧和右侧框显示共享代谢物的名称。
- (B) 与“A”面板类似,但是对比Knight-C4和ROSMAP-C1与其他病例之间共享的代谢物。重叠的显著性使用Fisher精确检验进行计算。

图7. 将单核数据与跨组学谱整合,确定了细胞类型特异性基因和通路。
- (A) 条形图显示在每个细胞类型中确定的上调和下调基因的百分比(聚类 vs. 对照组)。
- (B) 富集在星形胶质上调基因中的前20个KEGG通路。
- © 富集在神经元下调基因中的前20个GO生物过程通路。
- (D) 富集在神经元下调基因中的前20个KEGG通路。
- (E) 箱线图显示在Knight ADRC队列中确定的细胞类型特异性基因的转录组学谱(FPKM)。
- (F) 这些示例基因的效应大小是从Knight ADRC参与者的单核数据生成的。红色条代表基因在哪个细胞类型中过表达。
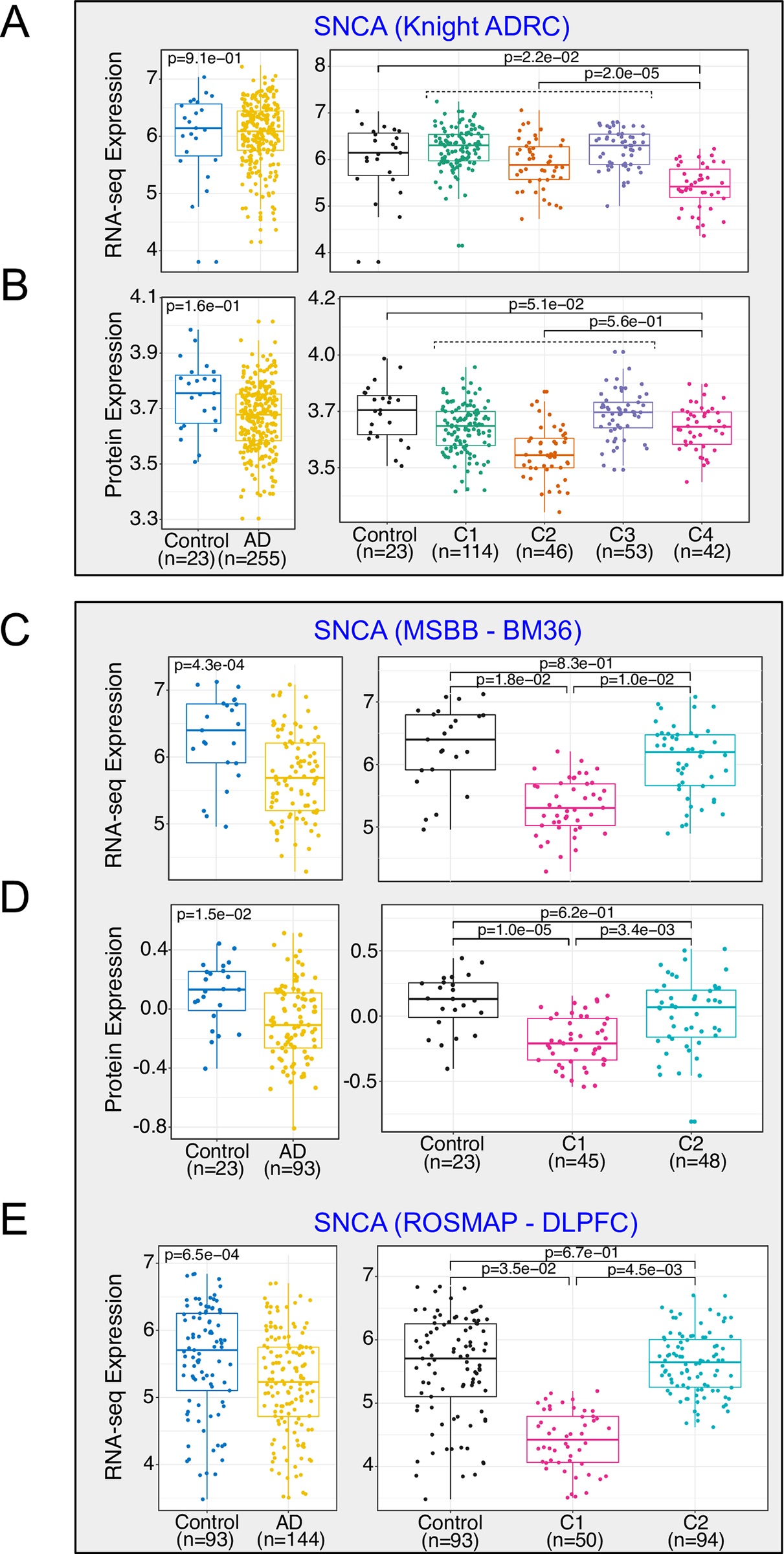
Fig 8. 跨组学整合确定了在多个脑区中,AD患者中α-突触核蛋白水平下调与认知功能更差的情况相关。
- (A) Knight ADRC队列中4个聚类(右侧)以及所有AD病例(左侧)的SNCA转录组学谱(FPKM)。
- (B) Knight ADRC队列中4个聚类(右侧)以及所有AD病例(左侧)的SNCA蛋白组学谱。
- © MSBB BM36队列中4个聚类(右侧)以及所有AD病例(左侧)的SNCA转录组学谱。
- (D) MSBB BM36队列中4个聚类(右侧)以及所有AD病例(左侧)的SNCA蛋白组学谱(TMT)。
- (E) ROSMAP DLPFC队列中4个聚类(右侧)以及所有AD病例(左侧)的SNCA转录组学谱。

Fig 9. 多个阶段的AD中的突触失调确定了CSF突触生物标志物用于AD的分子分期。
- (A) 散点图显示了Knight-C4和ROSMAP中常见的突触失调基因在早期AD(EAD)和晚期AD(AD)之间效应大小的相关性。
- (B) 热图显示在早期AD和晚期AD中检测到的最显著突触基因的效应大小相似性。
- © 箱线图显示IGF1、NRXN3和YWHAZ基因在对照组、早期AD和晚期AD中的一致表达(转录组学)。
- (D) Kaplan–Meier曲线显示了IGF1、NRXN3和YWHAZ基因与CSF蛋白质组学数据中痴呆进展风险增加的关联。
小结
- 主要数据及方法:
| Types | Notes |
|---|---|
| 分析数据 | NIAGADS数据库:NG00083、NG00102、NG00113、NG00108;Synapse数据库:syn3157743、syn25006650、syn17008934、syn25878459 |
| 分析方法 | iClusterPlus聚类;CellMix预测细胞类型比例;差异分析(DESeq2、glm、glmmTMB);生存分析;通路分析;WGCNA基因共表达网络 |
- 副表和副图里还有很多分析的阶段性结果,学习文章完整性整个研究思路强烈建议阅读原文全文内容