欢迎关注我的CSDN:https://spike.blog.csdn.net/
本文地址:https://spike.blog.csdn.net/article/details/132357976

Paper: EigenFold: Generative Protein Structure Prediction with Diffusion Models
EigenFold 是用于蛋白质结构预测的扩散生成模型(即,已知序列 至 结构分布)。基于谐波扩散,将键约束纳入扩散建模框架,并且产生一个级联分辨率的生成过程。
- 扩散生成模型 (Diffusion Generative Model):利用随机扩散过程,生成数据样本的机器学习模型。
- 谐波扩散 (Harmonic Diffusion):考虑谐波势能对于扩散过程的影响的数学模型。
- 键约束 (Bond Constraints):限制蛋白质中原子间距离和角度变化范围的物理条件。
- 级联分辨率 (Cascading-Resolution) :从粗糙到精细,逐步提高生成结果质量的方法。
- OmegaFold 嵌入向量(OmegaFold Embeddings):由 OmegaFold 模型产生的,表示蛋白质序列特征的向量。
关于 EigenFold,即:
We define a diffusion process that models the structure as a system of harmonic oscillators and which naturally induces a cascading-resolution generative process along the eigenmodes of the system.
扩散过程,即将结构模型化为谐振子 (Harmonic Oscillators) 系统,该过程自然地沿着系统的本征模式 (Eigenmodes),产生级联分辨率的生成过程。
EigenFold 算法重点:
- 蛋白质结构生成的新方法: 基于扩散模型的生成式模型,可以从给定的蛋白质序列生成一组可能的结构。该模型利用 OmegaFold 的预训练嵌入和得分网络来学习蛋白质结构的概率分布。
- 谐波扩散过程:定义新的扩散过程,将蛋白质结构建模为一系列谐振子,其势能为相邻残基之间的距离的二次函数。该过程可以保证采样的结构满足化学约束,并且可以沿着系统的本征模式进行投影,实现逐步精细化的生成过程。
- 得分网络架构:使用基于 E3NN 的图神经网络作为得分网络,输入为残基坐标和 OmegaFold 嵌入向量,输出为梯度向量。该网络具有 SE(3) 等变性,保证最终模型密度也具有 SE(3) 不变性。
EigenFold GitHub: https://github.com/bjing2016/EigenFold
1. 结构预测
准备 new.csv 文件,预测 7skh.B 的结构,即:
# with columns name, seqres (see provided splits for examples) and run
name,valid_alphas,seq,head,resolution,deposition_date,release_date,structure_method,seqres,seqlen
7skh.B.pdb,220,NAPVFQQPHYEVVLDEGPDTINTSLITVQALDGTVTYAIVAGNIINTFRINKHTGVITAAKELDYEISHGRYTLIVTATDQCPILSHRLTSTTTVLVNVNDINDNVPTFPRDYEGPFDVTEGQPGPRVWTFLAHDRDSGPNGQVEYSVVDGDPLGEFVISPVEGVLRVRKDVELDRETIAFYNLTICARDRGVPPLSSTMLVGIRVLDINDNLEHHHHHH,cell adhesion,2.27,2021-10-20,2022-10-26,x-ray diffraction,MNAPVFQQPHYEVVLDEGPDTINTSLITVQALDLDEGPNGTVTYAIVAGNIINTFRINKHTGVITAAKELDYEISHGRYTLIVTATDQCPILSHRLTSTTTVLVNVNDINDNVPTFPRDYEGPFDVTEGQPGPRVWTFLAHDRDSGPNGQVEYSVVDGDPLGEFVISPVEGVLRVRKDVELDRETIAFYNLTICARDRGVPPLSSTMLVGIRVLDINDNLEHHHHHH,227
运行命令:
python make_embeddings.py --out_dir ./embeddings --splits mydata/new.csv
python inference.py --model_dir ./pretrained_model --ckpt epoch_7.pt --pdb_dir ./structures --embeddings_dir ./embeddings --embeddings_key name --elbo --num_samples 5 --alpha 1 --beta 3 --elbo_step 0.2 --splits mydata/new.csv
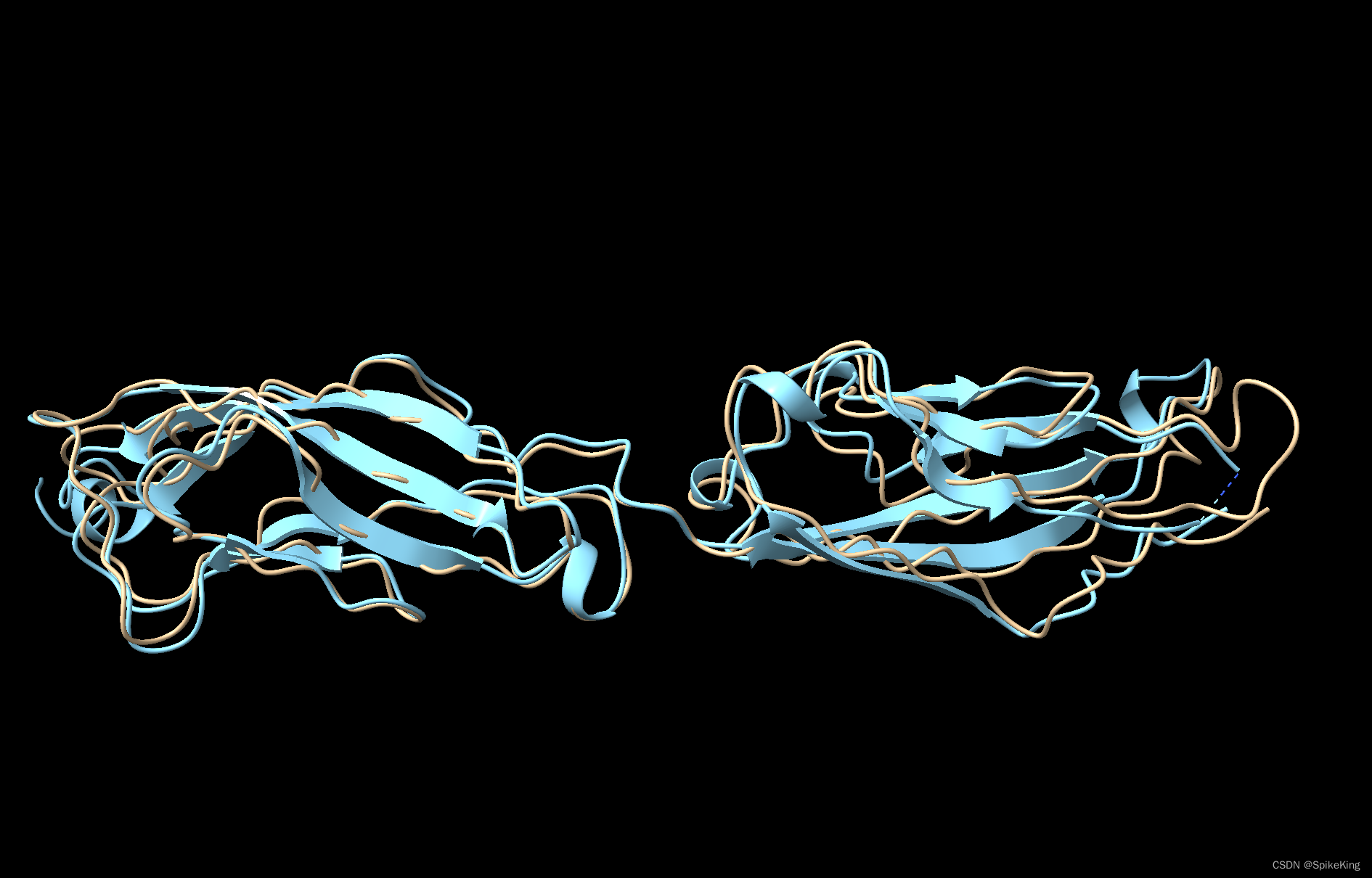
预测的蛋白质结构,如下:
- EigenFold 算法只能预测 CA 骨架,其余需要填充。
- 黄色是 EigenFold 的预测结构,蓝色是真实的 PDB 结构 (7skh.B)。
即:
2. 环境配置
下载 GitHub 工程:
git clone git@github.com:bjing2016/EigenFold.git
2.1 配置 Docker 环境
构建 Docker 环境:
nvidia-docker run -it --name eigenfold-[your name] -v [nfs path]:[nfs path] af2:v1.02
预先配置 Docker 环境中的 conda 源 与 pip 源,加速下载过程,参考 开源可训练的蛋白质结构预测框架 OpenFold 的环境配置
如果安装错误,清空 conda 环境,建议使用 rsync 快速删除,即:
mkdir tmp
rsync -a --delete tmp/ /opt/conda/envs/eigenfold
rm -rf /opt/conda/envs/eigenfold
配置 conda 环境,即:
# 安装 conda 环境
conda create -n eigenfold python=3.8
conda activate eigenfold
2.2 配置 PyTorch 系列包
安装 PyTorch,建议使用 conda 安装,而不是 pip 安装,参考 Installing Previous Versions of PyTorch 即:
# pip 安装异常,建议使用 conda 安装。
# pip install torch==1.11.0+cu113 torchvision==0.12.0+cu113 torchaudio==0.11.0 --extra-index-url https://download.pytorch.org/whl/cu113
conda install pytorch==1.11.0 torchvision==0.12.0 torchaudio==0.11.0 cudatoolkit=11.3 -c pytorch
预先测试 PyTorch 是否安装成功,即:
pythonimport torch
print(torch.__version__) # 1.11.0
print(torch.cuda.is_available()) # True
再安装 PyTorch 相关包,一共 5 个包,即 torch-scatter、torch-sparse、torch-cluster、torch-spline-conv、torch-geometric,建议逐个安装,排查问题,即:
pip install torch-scatter -f https://data.pyg.org/whl/torch-1.11.0+cu113.html
pip install torch-sparse -f https://data.pyg.org/whl/torch-1.11.0+cu113.html
pip install torch-cluster -f https://data.pyg.org/whl/torch-1.11.0+cu113.html
pip install torch-spline-conv -f https://data.pyg.org/whl/torch-1.11.0+cu113.html
pip install torch-geometric -f https://data.pyg.org/whl/torch-1.11.0+cu113.html
安装其他依赖包:
pip install e3nn pyyaml wandb biopython matplotlib pandas
2.3 配置 OmegaFold 依赖
安装 OmegaFold 依赖,即:
# 调用时,需要在 EigenFold 的根目录下。
wget https://helixon.s3.amazonaws.com/release1.pt
git clone https://github.com/bjing2016/OmegaFold
pip install --no-deps -e OmegaFold
注意需要预先下载 OmegaFold 的模型
release1.pt,大约 3 个 G左右。
OmegaFold GitHub: OmegaFold
This command will download the weight from https://helixon.s3.amazonaws.com/release1.pt to
~/.cache/omegafold_ckpt/model.ptand load the model
cd EigenFold
bypy info
bypy downfile /huggingface/eigenfold/omegafold-release1.pt model.pt
2.4 配置 TMScore 与 LDDT
安装 TMScore 与 LDDT,即:
mkdir /opt/bin
cd ~/binwget https://openstructure.org/static/lddt-linux.zip
unzip lddt-linux.zip
cp lddt-linux/lddt .
./lddt # 测试wget https://zhanggroup.org/TM-score/TMscore.cpp
g++ -static -O3 -ffast-math -lm -o TMscore TMscore.cpp
./TMscore # 测试export PATH="/opt/bin/:$PATH"
2.6 上传 Docker
提交 docker image,设置标签 (tag),以及上传 docker 至服务器,即:
# 提交 Tag
docker ps -l
docker commit [container id] eigenfold:v1.0# 准备远程 Tag
docker tag eigenfold:v1.0 harbor.[ip].com/[your name]/eigenfold:v1.0
docker images | grep "eigenfold"# 推送至远程
docker push harbor.[ip].com/[your name]/eigenfold:v1.0
# 从远程拉取
docker pull harbor.[ip].com/[your name]/eigenfold:v1.0# 或者保存至本地
docker save eigenfold:v1.0 | gzip > eigenfold_v1_0.tar.gz
# 加载已保存的 docker image
docker image load -i eigenfold_v1_0.tar.gz
docker images | grep "eigenfold"
BugFix
Bug1: torch_sparse 版本不兼容问题。
RuntimeError:
object has no attribute sparse_csc_tensor:File "/opt/conda/envs/eigenfold/lib/python3.8/site-packages/torch_sparse/tensor.py", line 520value = torch.ones(self.nnz(), dtype=dtype, device=self.device())return torch.sparse_csc_tensor(colptr, row, value, self.sizes())~~~~~~~~~~~~~~~~~~~~~~~ <--- HERE
参考: torch has no attribute sparse_csr_tensor
将 torch-sparse 降级至 0.6.14 版本,即可:
conda list torch-sparse
# packages in environment at /opt/conda/envs/eigenfold:
#
# Name Version Build Channel
torch-sparse 0.6.17 pypi_0 pypipip install torch-sparse==0.6.14 -f https://data.pyg.org/whl/torch-1.11.0+cu113.html
Bug2: Python 3.9 新特性不兼容问题
TypeError: unsupported operand type(s) for |: 'dict' and 'dict'
原因:What’s New In Python 3.9
方案1是升级至 Python3.9 版本,方案2是修改源码,位于EigenFold/utils/pdb.py,即:
# d[key] = {'CA': 'C'} | {key: val['symbol'] for key, val in atoms.items() if val['symbol'] != 'H' and key != 'CA'}
dict1 = {'CA': 'C'}
dict2 = {key: val['symbol'] for key, val in atoms.items() if val['symbol'] != 'H' and key != 'CA'}
d[key] = {**dict1, **dict2}
其余参考:
- Linux 下删除大量文件效率对比,看谁删的快!